+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22017 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Pig R615C RyR1 EGTA (all classes, open) | |||||||||
 マップデータ マップデータ | RyR1, FKBP12.6 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | receptor / calcium / channel / complex / TRANSPORT PROTEIN-ISOMERASE complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of sequestering of calcium ion / negative regulation of calcium-mediated signaling / negative regulation of insulin secretion involved in cellular response to glucose stimulus / neuronal action potential propagation / negative regulation of release of sequestered calcium ion into cytosol / insulin secretion involved in cellular response to glucose stimulus / response to redox state / negative regulation of heart rate / 'de novo' protein folding / FK506 binding ...positive regulation of sequestering of calcium ion / negative regulation of calcium-mediated signaling / negative regulation of insulin secretion involved in cellular response to glucose stimulus / neuronal action potential propagation / negative regulation of release of sequestered calcium ion into cytosol / insulin secretion involved in cellular response to glucose stimulus / response to redox state / negative regulation of heart rate / 'de novo' protein folding / FK506 binding / smooth muscle contraction / T cell proliferation / regulation of cardiac muscle contraction by regulation of the release of sequestered calcium ion / calcium channel inhibitor activity / regulation of release of sequestered calcium ion into cytosol by sarcoplasmic reticulum / Ion homeostasis / release of sequestered calcium ion into cytosol / calcium channel complex / sarcoplasmic reticulum membrane / protein maturation / calcium channel regulator activity / peptidylprolyl isomerase / peptidyl-prolyl cis-trans isomerase activity / calcium-mediated signaling / Stimuli-sensing channels / Z disc / positive regulation of cytosolic calcium ion concentration / protein refolding / transmembrane transporter binding / signaling receptor binding / membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||
 データ登録者 データ登録者 | Woll KW / Haji-Ghassemi O | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Pathological conformations of disease mutant Ryanodine Receptors revealed by cryo-EM. 著者: Kellie A Woll / Omid Haji-Ghassemi / Filip Van Petegem /  要旨: Ryanodine Receptors (RyRs) are massive channels that release Ca from the endoplasmic and sarcoplasmic reticulum. Hundreds of mutations are linked to malignant hyperthermia (MH), myopathies, and ...Ryanodine Receptors (RyRs) are massive channels that release Ca from the endoplasmic and sarcoplasmic reticulum. Hundreds of mutations are linked to malignant hyperthermia (MH), myopathies, and arrhythmias. Here, we explore the first MH mutation identified in humans by providing cryo-EM snapshots of the pig homolog, R615C, showing that it affects an interface between three solenoid regions. We also show the impact of apo-calmodulin (apoCaM) and how it can induce opening by bending of the bridging solenoid, mediated by its N-terminal lobe. For R615C RyR1, apoCaM binding abolishes a pathological 'intermediate' conformation, distributing the population to a mixture of open and closed channels, both different from the structure without apoCaM. Comparisons show that the mutation primarily affects the closed state, inducing partial movements linked to channel activation. This shows that disease mutations can cause distinct pathological conformations of the RyR and facilitate channel opening by disrupting interactions between different solenoid regions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22017.map.gz emd_22017.map.gz | 398.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22017-v30.xml emd-22017-v30.xml emd-22017.xml emd-22017.xml | 22.7 KB 22.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_22017.png emd_22017.png | 185.7 KB | ||
| Filedesc metadata |  emd-22017.cif.gz emd-22017.cif.gz | 7.9 KB | ||
| その他 |  emd_22017_additional_1.map.gz emd_22017_additional_1.map.gz emd_22017_half_map_1.map.gz emd_22017_half_map_1.map.gz emd_22017_half_map_2.map.gz emd_22017_half_map_2.map.gz | 36.7 MB 390.4 MB 390.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22017 http://ftp.pdbj.org/pub/emdb/structures/EMD-22017 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22017 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22017 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22017_validation.pdf.gz emd_22017_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22017_full_validation.pdf.gz emd_22017_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_22017_validation.xml.gz emd_22017_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  emd_22017_validation.cif.gz emd_22017_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22017 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22017 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22017 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22017 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22017.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22017.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RyR1, FKBP12.6 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
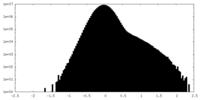
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Density modified map generated from PHENIX Resolve
| ファイル | emd_22017_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density modified map generated from PHENIX Resolve | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
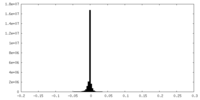
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_22017_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
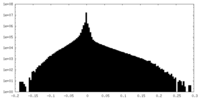
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_22017_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
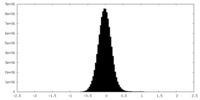
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ryanodine receptor-FKBP1B complex
| 全体 | 名称: ryanodine receptor-FKBP1B complex |
|---|---|
| 要素 |
|
-超分子 #1: ryanodine receptor-FKBP1B complex
| 超分子 | 名称: ryanodine receptor-FKBP1B complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|
-超分子 #2: ryanodine receptor
| 超分子 | 名称: ryanodine receptor / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: FKBP1B
| 超分子 | 名称: FKBP1B / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Peptidyl-prolyl cis-trans isomerase FKBP1B
| 分子 | 名称: Peptidyl-prolyl cis-trans isomerase FKBP1B / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: peptidylprolyl isomerase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.538191 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GVEIETISPG DGRTFPKKGQ TCVVHYTGML QNGKKFDSSR DRNKPFKFRI GKQEVIKGFE EGAAQMSLGQ RAKLTCTPDV AYGATGHPG VIPPNATLIF DVELLNL UniProtKB: Peptidyl-prolyl cis-trans isomerase FKBP1B |
-分子 #2: Ryanodine Receptor
| 分子 | 名称: Ryanodine Receptor / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 414.496875 KDa |
| 配列 | 文字列: FLRTDDEVVL QCNATVLKEQ LKLCLAAEGF GNRLCFLEPT SNAQNVPPDL AICCFVLEQS LSVRALQEML ANGHRTLLYG HAILLRHAH SGMYLSCLTT SRSMTDKLAF DVGLQEDATG EACWWTTHPA SKQRSEGEKV RVGDDLILVS VSSERYLHLS T ASGELQVD ...文字列: FLRTDDEVVL QCNATVLKEQ LKLCLAAEGF GNRLCFLEPT SNAQNVPPDL AICCFVLEQS LSVRALQEML ANGHRTLLYG HAILLRHAH SGMYLSCLTT SRSMTDKLAF DVGLQEDATG EACWWTTHPA SKQRSEGEKV RVGDDLILVS VSSERYLHLS T ASGELQVD ASFMQTLWNM NPICSGCEEG YVTGGHVLRL FHGHMDECLT ISPADSDDQR RLVYYEGGSV CTHARSLWRL EP LRISWSG SHLRWGQPLR IRHVTTGRYL ALIEDQGLVV DASKAHTKAT SFCFRISKEK LKRDVEGMGP PEIKYGESLC FVQ HVASGL WLTYAALKKK AILHQEGHMD DALSLTRCQQ EESQAARMIY STAGLYNHFI KGLDSFSGKP RGSGAPAGTA LPLE GVILS LQDLIGYFEP PSEELQHEEK QSKLRSLRNR QSLFQEEGML SLVLNCIDRL NVYTTAAHFA EFAGEEAAES WKEIV NLLY EILASLIRGN RANCALFSNN LDWLVSKLDR LEASSGILEV LYCVLIESPE VLNIIQENHI KSIISLLDKH GRNHKV LDV LCSLCVCNGV AVCSNQDLIT ENLLPGRELL LQTNLINYVT SIRPNIFVGR AEGTTQYSKW YFEVMVDEVV PFLTAQA TH LRVGWALTEG YSPYPGGGEG WGGNGVGDDL YSYGFDGLHL WTGHVPRLVT SPGQHLLAPE DVVSCCLDLS VPSISFRI N GCPVQGVFEA FNLNGLFFPV VSFSAGVKVR FLLGGRHGEF KFLPPPGYAP CHEAVLPRER LRLEPIKEYR REGPRGPHL VGPSRCLSHT DFVPCPLPPH LERIREKLAE NIHELWALTR IEQGWTYGPV RDDNKRLHPC LVDFHSLPEP ERNYNLQMSG ETLKTLLAL GCHVGMADEK AEDNLRKTKL PKTYMMSNGY KPAPLDAQTT LVDRLAENGH NVWARDRVAQ GWSYSAVQDI P ARRNPRLV PYRLLDEATK RSNRDSLCQA VRTLLGYGYN IERVRIFRAE KSYAVQSGRW YFEFEAVTTG EMRVGWARPE LR PDVELGA DELAYVFNGH RGQRWHLGSE LFGRPWQSGD VVGCMIDLTE NTIIFTLNGE VLMSDSGSET AFRDIEVGDG FLP VCSLGP GQVGHLNLGQ DVSSLRFFAI CGLQEGFEPF AINMQRPVTT WFSKSLPQFE AVPLEHPHYE VSRVDGTVDT PPCL RLTHR SLVEMLFLRL SLPVQFHQLN TTTYYYSVRV FAGQEPSCVW VGWVTPDYHQ HDMNFDLTKV RAVTVTMGDN IHSSL KCSN CYMVWGGDFV SHTDLVIGCL VDLATGLMTF TANGKESNTF FQVEPNTKLF PAVFVLPTHQ NVIQFELGKQ KNIMPL SAA MFLSERKNPA PQCPPRLEMQ MLMPVSWSRM PNHFLRVETR RAGERLGWAV PLTMMALHIP EENRCMDILE LSERLDL QQ FHSHTLRLYR AVCALGNNRV AHALCSHVDQ AQLLHALEDA HLPGPLRAGY YDLLISIHLE SACRSRRSML SEYIVPLT P ETRAITLFPP RHGLPGVGVT TSLRPPHHFS APCFVAALPE APARLSPSIP LEALRDKALR MLGEAVRDGG QHARDPVGG SVEFQFVPVL KLVSTLLVMG IFGDEDVKQI LKMIEPEVEE GLLQMKLPES VKLQMCNLLE YFCDQELQHR VESLAAFAER YVDKLQANQ RDRYGILMKA FTMTAAETAR RTREFRSPPQ EQINMLLHFK PLPDEIRQDL LEFHQDLLTH CGIQLQSLQE L VSHTVVRW AQEDFVQSPE LVRAMFSLLH RQYDGLGELL RALPRAYTIS PSSVEDTMSL LECLGQIRSL LIVQMGPQEE NL MIQSIGN IMNNKVFYQH PNLMRALGMH ETVMEVMVNV LRFPKMVTSC CRFLCYFCRI SRQNQRSMFD HLSYLLENSG STP LDVAAA SVIDNNELAL ALQEQDLEKV VSYLAGCGLQ SCPMLLAKGY PDIGWNPCGG ERYLDFLRFA VFVNGESVEE NANV VVRLL IRKPECFGPA LRLLATIEEA IAIMSFYAAL IDLLGRCAPE MHLIQAGKGE ALRIRAILRS LVPLDDLVGI ISLPL QIPT MSASFVPDHK ASMVLFLDRV YGFLLHVLDV GFALALNRYL CLAVLPLITK CAPLFAMVDS MLHTVYRLSR GRSLTK AQR DVIEECLMAL CRYIRPSMLQ HLLRRLVF(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)DPR PVETLNVIIP EKLDSFINKF AEYTHEKWAF DKIQNNWSYG ENIDEELKTH PM LRPYKTF SEKDKEIYRW PIKESLKAMI AWEWTIEKAR EGEYNPQPPD LSGVTLSREL QAMAEQLAEN YHNTWGRKKK QEL EAKGGG THPLLVPYDT LTAKEKARDR EKAQELLKFL QMNGYAVTR(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)EFSVLCRDL YALYPLLIRY VDNNRAHWLT E PNPSAEEL FRMVGEIFIY WSKSKHNFKR EEQNFVV(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)P LYNLPTHRAC NMFLESYKAA WILT EDHSF EDRMIDDLSK AKKPDPLHQL VLHFSRTALT EKSKLDEDYL YMAYADIMAK SCHLSFEEKE MEKQRLLYQQ ARLHN RGAA EMVLQMISAC KGETGAMVSS TLKLGISILN GGNADVQQKM LDYLKDKKEV GFFQSIQALM QTCSVLDLNA FERQNK AEG LGEKVMADDE FTQDLFRFLQ LLCEGHNNDF QNYLRTQTGN TTTINIIICT VDYLLRLQES ISDFYWYYSG KDVIEEQ GK RNFSKAMSVA KQVFNSLTEY IQGPCTGNQQ SLAHSRLWDA VVGFLHVFAH MMMKLAQDSS QIELLKELLD LQKDMVVM L LSLLEGNVVN GMIARQMVDM LVESSSNVEM ILKFFDMFLK LKDIVGSEAF QDYVTDPRGL ISKKDFQK(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)EFANR FQEPARDIGF NVAVLLTNLS EHVPHDPRLR NFLELAESIL EYFRPYLGRI EIMGA SRRI ERIYFEISET NRAQWEMPQV KESKRQFIFD VVNEGGESEK MELFVSFCED TIFEEVQRVK FLNYLSRNFY TLRFLA LFL AFAINFILLF YKVSDSPVYY FLEESTGYME PALRCLSLLH TLVAFLCIIG YNCLKVPLVI FKREKELARK LEFDGLY IT EQPVKGQWDR LVLNTPSFPS NYWDKFVKRK VLDKHGDIYG RERIAEGLLT WLMSIDVKYQ IWKFGVIFTD NSFLYLGW Y MVMSLLGHYN NFFFAAHLLD IAMGVKTLRT ILSSVTHNGK QLVMTVGLLA VVVYLYTVVA FNFFRKFYNK SEDEDEPDM KCDDMMTCYL FHMYVGVRAG GGIGDEIEDP AGDEYELYRV VFDITFFFFV IVILLAIIQG LIIDAFGELR DQQEQVREDM ETKCFICGI GSDYFDTTPH RFETHTLEEH NLANYMFFLM YLINKDETEH TGQESYVWKM YQERCWDFFP AGDCFRKQ |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: DIFFRACTION |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: PHENIX (ver. dev-3714) / 使用した粒子像数: 58822 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー




























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)