+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2139 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
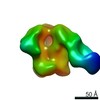
| タイトル | EM map of an influenza H3 hemagglutatin in complex with C05 Fab | |||||||||
 マップデータ マップデータ | Negative stain reconstruction of flu HA3 in complex with C5 fab | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Influenza A / flu / hemagglutanin / HA neutralizing antibody / C5 fab / C05 | |||||||||
| 生物種 |   Influenza A virus (A型インフルエンザウイルス) / Influenza A virus (A型インフルエンザウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 17.0 Å | |||||||||
 データ登録者 データ登録者 | Khayat R / Lee JH / Ekiert DC / Wilson IA / Ward AB | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2012 ジャーナル: Nature / 年: 2012タイトル: Cross-neutralization of influenza A viruses mediated by a single antibody loop. 著者: Damian C Ekiert / Arun K Kashyap / John Steel / Adam Rubrum / Gira Bhabha / Reza Khayat / Jeong Hyun Lee / Michael A Dillon / Ryann E O'Neil / Aleksandr M Faynboym / Michael Horowitz / ...著者: Damian C Ekiert / Arun K Kashyap / John Steel / Adam Rubrum / Gira Bhabha / Reza Khayat / Jeong Hyun Lee / Michael A Dillon / Ryann E O'Neil / Aleksandr M Faynboym / Michael Horowitz / Lawrence Horowitz / Andrew B Ward / Peter Palese / Richard Webby / Richard A Lerner / Ramesh R Bhatt / Ian A Wilson /  要旨: Immune recognition of protein antigens relies on the combined interaction of multiple antibody loops, which provide a fairly large footprint and constrain the size and shape of protein surfaces that ...Immune recognition of protein antigens relies on the combined interaction of multiple antibody loops, which provide a fairly large footprint and constrain the size and shape of protein surfaces that can be targeted. Single protein loops can mediate extremely high-affinity binding, but it is unclear whether such a mechanism is available to antibodies. Here we report the isolation and characterization of an antibody called C05, which neutralizes strains from multiple subtypes of influenza A virus, including H1, H2 and H3. X-ray and electron microscopy structures show that C05 recognizes conserved elements of the receptor-binding site on the haemagglutinin surface glycoprotein. Recognition of the haemagglutinin receptor-binding site is dominated by a single heavy-chain complementarity-determining region 3 loop, with minor contacts from heavy-chain complementarity-determining region 1, and is sufficient to achieve nanomolar binding with a minimal footprint. Thus, binding predominantly with a single loop can allow antibodies to target small, conserved functional sites on otherwise hypervariable antigens. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2139.map.gz emd_2139.map.gz | 3.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2139-v30.xml emd-2139-v30.xml emd-2139.xml emd-2139.xml | 10.5 KB 10.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2139.png emd_2139.png | 104.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2139 http://ftp.pdbj.org/pub/emdb/structures/EMD-2139 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2139 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2139 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2139_validation.pdf.gz emd_2139_validation.pdf.gz | 209.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2139_full_validation.pdf.gz emd_2139_full_validation.pdf.gz | 208.9 KB | 表示 | |
| XML形式データ |  emd_2139_validation.xml.gz emd_2139_validation.xml.gz | 5.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2139 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2139 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2139 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2139 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2139.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2139.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain reconstruction of flu HA3 in complex with C5 fab | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.18 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Influenza HA3 in complex with C5 Fab
| 全体 | 名称: Influenza HA3 in complex with C5 Fab |
|---|---|
| 要素 |
|
-超分子 #1000: Influenza HA3 in complex with C5 Fab
| 超分子 | 名称: Influenza HA3 in complex with C5 Fab / タイプ: sample / ID: 1000 / 集合状態: One HA trimer binds to 3 fabs / Number unique components: 2 |
|---|
-分子 #1: Influenza A/Perth/16/2009 hemagglutinin
| 分子 | 名称: Influenza A/Perth/16/2009 hemagglutinin / タイプ: protein_or_peptide / ID: 1 / Name.synonym: HA3 / コピー数: 3 / 集合状態: Trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス)株: A/Perth/16/2009 / 別称: Flu |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) / 組換プラスミド: pDCE198 unidentified baculovirus (ウイルス) / 組換プラスミド: pDCE198 |
-分子 #2: C05 antibody fab fragment
| 分子 | 名称: C05 antibody fab fragment / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) / 組換プラスミド: pFastBacDual unidentified baculovirus (ウイルス) / 組換プラスミド: pFastBacDual |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 20mM HEPES pH 7.5, 50mM NaCl |
| 染色 | タイプ: NEGATIVE / 詳細: Nano-W for 30 seconds |
| グリッド | 詳細: 400 mesh copper grid |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 最低: 293 K / 最高: 293 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism corrected at 100,000x |
| 詳細 | Images taken from 0 to 55 degrees in 5 degree tilt increments |
| 日付 | 2012年2月14日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GENERIC GATAN (4k x 4k) デジタル化 - サンプリング間隔: 0.109 µm / 実像数: 269 / 平均電子線量: 16 e/Å2 / 詳細: Images recorded using CCD / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 0.9 µm / 最小 デフォーカス(公称値): 0.7 µm / 倍率(公称値): 100000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / Tilt angle max: 55 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Particles were selected using an automated particle picking program. |
|---|---|
| CTF補正 | 詳細: Micrograph and each particle |
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 17.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Sparx / 使用した粒子像数: 28766 |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















