+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20735 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of CH235UCA bound to Man5-enriched CH505.N279K.G458Y.SOSIP.664 | |||||||||
 マップデータ マップデータ | Refined and sharpened map from Cryosparc | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CH235 lineage / CH505 virus / vaccine design / cryo-EM / VIRAL PROTEIN / VIRAL PROTEIN-Immune System complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated perturbation of host defense response / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell ...symbiont-mediated perturbation of host defense response / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Acharya P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2019 ジャーナル: PLoS Pathog / 年: 2019タイトル: Neutralization-guided design of HIV-1 envelope trimers with high affinity for the unmutated common ancestor of CH235 lineage CD4bs broadly neutralizing antibodies. 著者: Celia C LaBranche / Rory Henderson / Allen Hsu / Shay Behrens / Xuejun Chen / Tongqing Zhou / Kevin Wiehe / Kevin O Saunders / S Munir Alam / Mattia Bonsignori / Mario J Borgnia / Quentin J ...著者: Celia C LaBranche / Rory Henderson / Allen Hsu / Shay Behrens / Xuejun Chen / Tongqing Zhou / Kevin Wiehe / Kevin O Saunders / S Munir Alam / Mattia Bonsignori / Mario J Borgnia / Quentin J Sattentau / Amanda Eaton / Kelli Greene / Hongmei Gao / Hua-Xin Liao / Wilton B Williams / James Peacock / Haili Tang / Lautaro G Perez / Robert J Edwards / Thomas B Kepler / Bette T Korber / Peter D Kwong / John R Mascola / Priyamvada Acharya / Barton F Haynes / David C Montefiori /   要旨: The CD4 binding site (CD4bs) of the HIV-1 envelope glycoprotein is susceptible to multiple lineages of broadly neutralizing antibodies (bnAbs) that are attractive to elicit with vaccines. The CH235 ...The CD4 binding site (CD4bs) of the HIV-1 envelope glycoprotein is susceptible to multiple lineages of broadly neutralizing antibodies (bnAbs) that are attractive to elicit with vaccines. The CH235 lineage (VH1-46) of CD4bs bnAbs is particularly attractive because the most mature members neutralize 90% of circulating strains, do not possess long HCDR3 regions, and do not contain insertions and deletions that may be difficult to induce. We used virus neutralization to measure the interaction of CH235 unmutated common ancestor (CH235 UCA) with functional Env trimers on infectious virions to guide immunogen design for this bnAb lineage. Two Env mutations were identified, one in loop D (N279K) and another in V5 (G458Y), that acted synergistically to render autologous CH505 transmitted/founder virus susceptible to neutralization by CH235 UCA. Man5-enriched N-glycans provided additional synergy for neutralization. CH235 UCA bound with nanomolar affinity to corresponding soluble native-like Env trimers as candidate immunogens. A cryo-EM structure of CH235 UCA bound to Man5-enriched CH505.N279K.G458Y.SOSIP.664 revealed interactions of the antibody light chain complementarity determining region 3 (CDR L3) with the engineered Env loops D and V5. These results demonstrate that virus neutralization can directly inform vaccine design and suggest a germline targeting and reverse engineering strategy to initiate and mature the CH235 bnAb lineage. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20735.map.gz emd_20735.map.gz | 118 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20735-v30.xml emd-20735-v30.xml emd-20735.xml emd-20735.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20735.png emd_20735.png | 111.6 KB | ||
| Filedesc metadata |  emd-20735.cif.gz emd-20735.cif.gz | 6.9 KB | ||
| その他 |  emd_20735_additional.map.gz emd_20735_additional.map.gz | 112.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20735 http://ftp.pdbj.org/pub/emdb/structures/EMD-20735 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20735 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20735 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20735_validation.pdf.gz emd_20735_validation.pdf.gz | 504.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20735_full_validation.pdf.gz emd_20735_full_validation.pdf.gz | 504.3 KB | 表示 | |
| XML形式データ |  emd_20735_validation.xml.gz emd_20735_validation.xml.gz | 6.7 KB | 表示 | |
| CIF形式データ |  emd_20735_validation.cif.gz emd_20735_validation.cif.gz | 7.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20735 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20735 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20735 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20735 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20735.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20735.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refined and sharpened map from Cryosparc | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
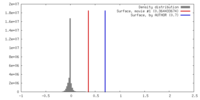
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Refined map from cryosparc sharpened in Phenix
| ファイル | emd_20735_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refined map from cryosparc sharpened in Phenix | ||||||||||||
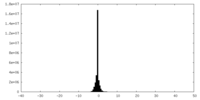
| 投影像・断面図 |
| ||||||||||||
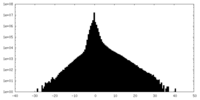
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of the CH235 unmutated common ancestor (UCA) bound to Man...
| 全体 | 名称: Complex of the CH235 unmutated common ancestor (UCA) bound to Man5-enriched CH505.N279K.G458Y.SOSIP.664 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of the CH235 unmutated common ancestor (UCA) bound to Man...
| 超分子 | 名称: Complex of the CH235 unmutated common ancestor (UCA) bound to Man5-enriched CH505.N279K.G458Y.SOSIP.664 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #2-#4 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 360 KDa |
-分子 #1: CH505.N279K.G458Y.SOSIP.664 gp120
| 分子 | 名称: CH505.N279K.G458Y.SOSIP.664 gp120 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 54.546402 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MPMGSLQPLA TLYLLGMLVA SVLAAENLWV TVYYGVPVWK EAKTTLFCAS DAKAYEKEVH NVWATHACVP TDPNPQEMVL KNVTENFNM WKNDMVDQMH EDVISLWDQS LKPCVKLTPL CVTLNCTNAT ASNSSIIEGM KNCSFNITTE LRDKREKKNA L FYKLDIVQ ...文字列: MPMGSLQPLA TLYLLGMLVA SVLAAENLWV TVYYGVPVWK EAKTTLFCAS DAKAYEKEVH NVWATHACVP TDPNPQEMVL KNVTENFNM WKNDMVDQMH EDVISLWDQS LKPCVKLTPL CVTLNCTNAT ASNSSIIEGM KNCSFNITTE LRDKREKKNA L FYKLDIVQ LDGNSSQYRL INCNTSVITQ ACPKVSFDPI PIHYCAPAGY AILKCNNKTF TGTGPCNNVS TVQCTHGIKP VV STQLLLN GSLAEGEIII RSENITKNVK TIIVHLNESV KIECTRPNNK TRTSIRIGPG QAFYATGQVI GDIREAYCNI NES KWNETL QRVSKKLKEY FPHKNITFQP SSGGDLEITT HSFNCGGEFF YCNTSSLFNR TYMANSTDMA NSTETNSTRT ITIH CRIKQ IINMWQEVGR AMYAPPIAGN ITCISNITGL LLTRDYGKNN TETFRPGGGN MKDNWRSELY KYKVVKIEPL GVAPT RCKR RVV UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: CH235 UCA heavy chain Fab
| 分子 | 名称: CH235 UCA heavy chain Fab / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 26.635938 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: METDTLLLWV LLLWVPGSTG DQVQLVQSGA EVKKPGASVK VSCKASGYTF TSYYMHWVRQ APGQGLEWMG IINPSGGSTS YAQKFQGRV TMTRDTSTST VYMELSSLRS EDTAVYYCAR DVGTEGSLLH FDYWGQGTLV TVSSASTKGP SVFPLAPSSK S TSGGTAAL ...文字列: METDTLLLWV LLLWVPGSTG DQVQLVQSGA EVKKPGASVK VSCKASGYTF TSYYMHWVRQ APGQGLEWMG IINPSGGSTS YAQKFQGRV TMTRDTSTST VYMELSSLRS EDTAVYYCAR DVGTEGSLLH FDYWGQGTLV TVSSASTKGP SVFPLAPSSK S TSGGTAAL GCLVKDYFPE PVTVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KR VEPKSCD KT |
-分子 #3: CH505.N279K.G458Y.SOSIP.664 gp41
| 分子 | 名称: CH505.N279K.G458Y.SOSIP.664 gp41 / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 18.146699 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GRRRRRRAVG IGAVFLGFLG AAGSTMGAAS MTLTVQARNL LSGIVQQQSN LLRAPEAQQH LLKLTVWGIK QLQARVLAVE RYLRDQQLL GIWGCSGKLI CCTNVPWNSS WSNRNLSEIW DNMTWLQWDK EISNYTQIIY GLLEESQNQQ EKNEQDLLAL D UniProtKB: Envelope glycoprotein gp160 |
-分子 #4: CH235 UCA light chain Fab
| 分子 | 名称: CH235 UCA light chain Fab / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.65957 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: METDTLLLWV LLLWVPGSTG DEIVMTQSPA TLSVSPGERA TLSCRASQSV SSNLAWYQQK PGQAPRLLIY GASTRATGIP ARFSGSGSG TEFTLTISSL QSEDFAVYYC QQYNNWWTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF Y PREAKVQW ...文字列: METDTLLLWV LLLWVPGSTG DEIVMTQSPA TLSVSPGERA TLSCRASQSV SSNLAWYQQK PGQAPRLLIY GASTRATGIP ARFSGSGSG TEFTLTISSL QSEDFAVYYC QQYNNWWTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF Y PREAKVQW KVDNALQSGN SQESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 42.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: C3 (3回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 4.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 22093 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)