+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human 20S proteasome assembly intermediate structure 2 | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Complex / HYDROLASE | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cerebellar granule cell precursor proliferation / purine ribonucleoside triphosphate binding / protein folding chaperone complex / Regulation of ornithine decarboxylase (ODC) / Proteasome assembly / Cross-presentation of soluble exogenous antigens (endosomes) / proteasome core complex / proteasome core complex assembly / Somitogenesis / mitotic spindle assembly checkpoint signaling ...cerebellar granule cell precursor proliferation / purine ribonucleoside triphosphate binding / protein folding chaperone complex / Regulation of ornithine decarboxylase (ODC) / Proteasome assembly / Cross-presentation of soluble exogenous antigens (endosomes) / proteasome core complex / proteasome core complex assembly / Somitogenesis / mitotic spindle assembly checkpoint signaling / proteasome binding / myofibril / immune system process / NF-kappaB binding / proteasome endopeptidase complex / proteasome core complex, beta-subunit complex / proteasome assembly / threonine-type endopeptidase activity / chaperone-mediated protein complex assembly / proteasome core complex, alpha-subunit complex / proteasome complex / proteolysis involved in protein catabolic process / sarcomere / Regulation of activated PAK-2p34 by proteasome mediated degradation / Autodegradation of Cdh1 by Cdh1:APC/C / APC/C:Cdc20 mediated degradation of Securin / Asymmetric localization of PCP proteins / Ubiquitin-dependent degradation of Cyclin D / SCF-beta-TrCP mediated degradation of Emi1 / NIK-->noncanonical NF-kB signaling / TNFR2 non-canonical NF-kB pathway / AUF1 (hnRNP D0) binds and destabilizes mRNA / Vpu mediated degradation of CD4 / Assembly of the pre-replicative complex / Ubiquitin-Mediated Degradation of Phosphorylated Cdc25A / Degradation of DVL / Cdc20:Phospho-APC/C mediated degradation of Cyclin A / Dectin-1 mediated noncanonical NF-kB signaling / lipopolysaccharide binding / Degradation of AXIN / Hh mutants are degraded by ERAD / negative regulation of inflammatory response to antigenic stimulus / Activation of NF-kappaB in B cells / P-body / Degradation of GLI1 by the proteasome / Hedgehog ligand biogenesis / G2/M Checkpoints / Defective CFTR causes cystic fibrosis / GSK3B and BTRC:CUL1-mediated-degradation of NFE2L2 / Autodegradation of the E3 ubiquitin ligase COP1 / Negative regulation of NOTCH4 signaling / Vif-mediated degradation of APOBEC3G / Regulation of RUNX3 expression and activity / Hedgehog 'on' state / Degradation of GLI2 by the proteasome / GLI3 is processed to GLI3R by the proteasome / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / APC/C:Cdh1 mediated degradation of Cdc20 and other APC/C:Cdh1 targeted proteins in late mitosis/early G1 / MAPK6/MAPK4 signaling / Degradation of beta-catenin by the destruction complex / response to virus / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / ABC-family proteins mediated transport / CDK-mediated phosphorylation and removal of Cdc6 / CLEC7A (Dectin-1) signaling / SCF(Skp2)-mediated degradation of p27/p21 / Regulation of expression of SLITs and ROBOs / FCERI mediated NF-kB activation / nuclear matrix / Regulation of PTEN stability and activity / Interleukin-1 signaling / Orc1 removal from chromatin / Regulation of RAS by GAPs / positive regulation of NF-kappaB transcription factor activity / Regulation of RUNX2 expression and activity / Separation of Sister Chromatids / The role of GTSE1 in G2/M progression after G2 checkpoint / UCH proteinases / KEAP1-NFE2L2 pathway / Antigen processing: Ubiquitination & Proteasome degradation / Downstream TCR signaling / RUNX1 regulates transcription of genes involved in differentiation of HSCs / Neddylation / ER-Phagosome pathway / regulation of inflammatory response / secretory granule lumen / endopeptidase activity / molecular adaptor activity / proteasome-mediated ubiquitin-dependent protein catabolic process / ficolin-1-rich granule lumen / Ub-specific processing proteases / nuclear speck / nuclear body / cilium / ribosome / intracellular membrane-bounded organelle / centrosome / ubiquitin protein ligase binding / Neutrophil degranulation / negative regulation of apoptotic process 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
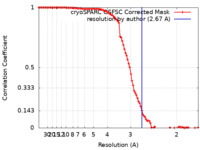
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.67 Å | |||||||||||||||
 データ登録者 データ登録者 | Schulman BA / Hanna JW / Harper JW / Adolf F / Du J / Rawson SD / Walsh Jr RM / Goodall EA | |||||||||||||||
| 資金援助 |  ドイツ, ドイツ,  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 | ジャーナル: bioRxiv / 年: 2024 タイトル: Visualizing chaperone-mediated multistep assembly of the human 20S proteasome. 著者: Frank Adolf / Jiale Du / Ellen A Goodall / Richard M Walsh / Shaun Rawson / Susanne von Gronau / J Wade Harper / John Hanna / Brenda A Schulman /   要旨: Dedicated assembly factors orchestrate stepwise production of many molecular machines, including the 28-subunit proteasome core particle (CP) that mediates protein degradation. Here, we report cryo- ...Dedicated assembly factors orchestrate stepwise production of many molecular machines, including the 28-subunit proteasome core particle (CP) that mediates protein degradation. Here, we report cryo-EM reconstructions of seven recombinant human subcomplexes that visualize all five chaperones and the three active site propeptides across a wide swath of the assembly pathway. Comparison of these chaperone-bound intermediates and a matching mature CP reveals molecular mechanisms determining the order of successive subunit additions, and how proteasome subcomplexes and assembly factors structurally adapt upon progressive subunit incorporation to stabilize intermediates, facilitate the formation of subsequent intermediates, and ultimately rearrange to coordinate proteolytic activation with gated access to active sites. The structural findings reported here explain many previous biochemical and genetic observations. This work establishes a methodologic approach for structural analysis of multiprotein complex assembly intermediates, illuminates specific functions of assembly factors, and reveals conceptual principles underlying human proteasome biogenesis. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18757.map.gz emd_18757.map.gz | 53.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18757-v30.xml emd-18757-v30.xml emd-18757.xml emd-18757.xml | 31.7 KB 31.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18757_fsc.xml emd_18757_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18757.png emd_18757.png | 54.8 KB | ||
| マスクデータ |  emd_18757_msk_1.map emd_18757_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18757.cif.gz emd-18757.cif.gz | 8.1 KB | ||
| その他 |  emd_18757_additional_1.map.gz emd_18757_additional_1.map.gz emd_18757_half_map_1.map.gz emd_18757_half_map_1.map.gz emd_18757_half_map_2.map.gz emd_18757_half_map_2.map.gz | 88.6 MB 95.8 MB 95.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18757 http://ftp.pdbj.org/pub/emdb/structures/EMD-18757 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18757 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18757 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8qylMC  8qyjC  8qymC  8qynC  8qyoC  8qysC  8qz9C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18757.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18757.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8512 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18757_msk_1.map emd_18757_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
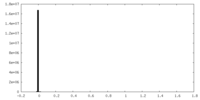
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_18757_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
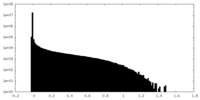
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18757_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
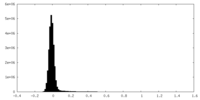
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18757_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
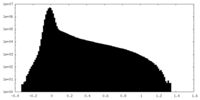
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
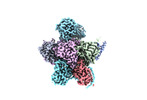
+全体 : Human 20S proteasome assembly intermediate map 2
+超分子 #1: Human 20S proteasome assembly intermediate map 2
+分子 #1: Proteasome subunit alpha type-2
+分子 #2: Proteasome subunit alpha type-4
+分子 #3: Proteasome subunit alpha type-7
+分子 #4: Proteasome subunit alpha type-5
+分子 #5: Proteasome subunit alpha type-1
+分子 #6: Proteasome subunit alpha type-3
+分子 #7: Proteasome subunit alpha type-6
+分子 #8: Proteasome maturation protein
+分子 #9: Proteasome assembly chaperone 1
+分子 #10: Proteasome assembly chaperone 2
+分子 #11: Proteasome subunit beta type-7
+分子 #12: Proteasome subunit beta type-3
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 66.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)












































 Trichoplusia ni (イラクサキンウワバ)
Trichoplusia ni (イラクサキンウワバ)