+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | ATP-bound IstB in complex to duplex DNA | |||||||||
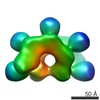
 マップデータ マップデータ | Sharpened Map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA Transposition / transposon / transpososome / AAA+ ATPases / target DNA / IS21 / IstB / Insertion sequence / DNA BINDING PROTEIN / cryo-electron microscopy. | |||||||||
| 機能・相同性 | DNA replication protein DnaC/insertion sequence putative ATP-binding protein / IstB-like ATP-binding protein / IstB-like ATP binding protein / ClpA/B family / ATPases associated with a variety of cellular activities / AAA+ ATPase domain / P-loop containing nucleoside triphosphate hydrolase / ATP binding / Insertion sequence IS5376 putative ATP-binding protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) | |||||||||
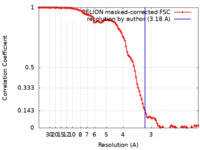
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.18 Å | |||||||||
 データ登録者 データ登録者 | de la Gandara A / Spinola-Amilibia M / Araujo-Bazan L / Nunez-Ramirez R / Berger JM / Arias-Palomo E | |||||||||
| 資金援助 |  スペイン, 2件 スペイン, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Molecular basis for transposase activation by a dedicated AAA+ ATPase. 著者: Álvaro de la Gándara / Mercedes Spínola-Amilibia / Lidia Araújo-Bazán / Rafael Núñez-Ramírez / James M Berger / Ernesto Arias-Palomo /   要旨: Transposases drive chromosomal rearrangements and the dissemination of drug-resistance genes and toxins. Although some transposases act alone, many rely on dedicated AAA+ ATPase subunits that ...Transposases drive chromosomal rearrangements and the dissemination of drug-resistance genes and toxins. Although some transposases act alone, many rely on dedicated AAA+ ATPase subunits that regulate site selectivity and catalytic function through poorly understood mechanisms. Using IS21 as a model transposase system, we show how an ATPase regulator uses nucleotide-controlled assembly and DNA deformation to enable structure-based site selectivity, transposase recruitment, and activation and integration. Solution and cryogenic electron microscopy studies show that the IstB ATPase self-assembles into an autoinhibited pentamer of dimers that tightly curves target DNA into a half-coil. Two of these decamers dimerize, which stabilizes the target nucleic acid into a kinked S-shaped configuration that engages the IstA transposase at the interface between the two IstB oligomers to form an approximately 1 MDa transpososome complex. Specific interactions stimulate regulator ATPase activity and trigger a large conformational change on the transposase that positions the catalytic site to perform DNA strand transfer. These studies help explain how AAA+ ATPase regulators-which are used by classical transposition systems such as Tn7, Mu and CRISPR-associated elements-can remodel their substrate DNA and cognate transposases to promote function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18136.map.gz emd_18136.map.gz | 85.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18136-v30.xml emd-18136-v30.xml emd-18136.xml emd-18136.xml | 27.3 KB 27.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18136_fsc.xml emd_18136_fsc.xml | 10.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18136.png emd_18136.png | 110.2 KB | ||
| マスクデータ |  emd_18136_msk_1.map emd_18136_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18136.cif.gz emd-18136.cif.gz | 6.9 KB | ||
| その他 |  emd_18136_additional_1.map.gz emd_18136_additional_1.map.gz emd_18136_additional_2.map.gz emd_18136_additional_2.map.gz emd_18136_half_map_1.map.gz emd_18136_half_map_1.map.gz emd_18136_half_map_2.map.gz emd_18136_half_map_2.map.gz | 69.6 MB 2.5 MB 70 MB 70 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18136 http://ftp.pdbj.org/pub/emdb/structures/EMD-18136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18136 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18136_validation.pdf.gz emd_18136_validation.pdf.gz | 905.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18136_full_validation.pdf.gz emd_18136_full_validation.pdf.gz | 905.4 KB | 表示 | |
| XML形式データ |  emd_18136_validation.xml.gz emd_18136_validation.xml.gz | 17.2 KB | 表示 | |
| CIF形式データ |  emd_18136_validation.cif.gz emd_18136_validation.cif.gz | 22.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18136 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18136 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18136 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18136 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8q3wMC  18144  8q4dC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18136.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18136.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened Map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18136_msk_1.map emd_18136_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : IstB transposition regulator AAA+ ATPase bound to target DNA
| 全体 | 名称: IstB transposition regulator AAA+ ATPase bound to target DNA |
|---|---|
| 要素 |
|
-超分子 #1: IstB transposition regulator AAA+ ATPase bound to target DNA
| 超分子 | 名称: IstB transposition regulator AAA+ ATPase bound to target DNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Complex of IstB transposition regulator AAA+ ATPase bound to target DNA |
|---|---|
| 分子量 | 理論値: 337 KDa |
-超分子 #2: Insertion sequence IS5376 putative ATP-binding protein
| 超分子 | 名称: Insertion sequence IS5376 putative ATP-binding protein タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
-超分子 #3: DNA (48-MER) Target DNA FW and Rv
| 超分子 | 名称: DNA (48-MER) Target DNA FW and Rv / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) / Synthetically produced: Yes Geobacillus stearothermophilus (バクテリア) / Synthetically produced: Yes |
-分子 #1: Insertion sequence IS5376 putative ATP-binding protein
| 分子 | 名称: Insertion sequence IS5376 putative ATP-binding protein タイプ: protein_or_peptide / ID: 1 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
| 分子量 | 理論値: 29.602275 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPNMKERIHE YCHRLHLPVM AERWSAMAEY ASTHNISYSE FLFRLLEAEI VEKQARSIQT LIKLSKLPYR KTIDTFDFTA QPSVDERRI RELLTLSFID RKENILFLGP PGIGKTHLAI SIGMEAIARG YKTYFITAHD LVNQLRRADQ EGKLEKKLRV F VKPTVLII ...文字列: GPNMKERIHE YCHRLHLPVM AERWSAMAEY ASTHNISYSE FLFRLLEAEI VEKQARSIQT LIKLSKLPYR KTIDTFDFTA QPSVDERRI RELLTLSFID RKENILFLGP PGIGKTHLAI SIGMEAIARG YKTYFITAHD LVNQLRRADQ EGKLEKKLRV F VKPTVLII DEMGYLKLDP NSAHYLFQVI ARRYEHAPII LTSNKSFGEW GEIVGDSVLA TAMLDRLLHH SIIFNLKGES YR LREKRLQ EEKQKDQ UniProtKB: Insertion sequence IS5376 putative ATP-binding protein |
-分子 #2: DNA (48-MER) Target DNA FW
| 分子 | 名称: DNA (48-MER) Target DNA FW / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
| 分子量 | 理論値: 18.401746 KDa |
| 配列 | 文字列: (DT)(DG)(DC)(DT)(DT)(DG)(DC)(DG)(DA)(DT) (DG)(DA)(DT)(DC)(DC)(DG)(DA)(DC)(DG)(DT) (DG)(DT)(DT)(DA)(DG)(DC)(DC)(DA)(DC) (DG)(DC)(DT)(DG)(DA)(DC)(DT)(DA)(DG)(DT) (DT) (DA)(DT)(DG)(DC)(DC) ...文字列: (DT)(DG)(DC)(DT)(DT)(DG)(DC)(DG)(DA)(DT) (DG)(DA)(DT)(DC)(DC)(DG)(DA)(DC)(DG)(DT) (DG)(DT)(DT)(DA)(DG)(DC)(DC)(DA)(DC) (DG)(DC)(DT)(DG)(DA)(DC)(DT)(DA)(DG)(DT) (DT) (DA)(DT)(DG)(DC)(DC)(DA)(DT)(DG) (DC)(DC)(DT)(DC)(DC)(DC)(DT)(DT)(DC)(DA) (DG)(DG) |
-分子 #3: DNA (48-MER) Traget DNA Rv
| 分子 | 名称: DNA (48-MER) Traget DNA Rv / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
| 分子量 | 理論値: 18.584918 KDa |
| 配列 | 文字列: (DC)(DC)(DT)(DG)(DA)(DA)(DG)(DG)(DG)(DA) (DG)(DG)(DC)(DA)(DT)(DG)(DG)(DC)(DA)(DT) (DA)(DA)(DC)(DT)(DA)(DG)(DT)(DC)(DA) (DG)(DC)(DG)(DT)(DG)(DG)(DC)(DT)(DA)(DA) (DC) (DA)(DC)(DG)(DT)(DC) ...文字列: (DC)(DC)(DT)(DG)(DA)(DA)(DG)(DG)(DG)(DA) (DG)(DG)(DC)(DA)(DT)(DG)(DG)(DC)(DA)(DT) (DA)(DA)(DC)(DT)(DA)(DG)(DT)(DC)(DA) (DG)(DC)(DG)(DT)(DG)(DG)(DC)(DT)(DA)(DA) (DC) (DA)(DC)(DG)(DT)(DC)(DG)(DG)(DA) (DT)(DC)(DA)(DT)(DC)(DG)(DC)(DA)(DA)(DG) (DC)(DA) |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 10 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 10 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均露光時間: 5.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 95.21 |
|---|---|
| 得られたモデル |  PDB-8q3w: |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X