+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1792 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EM map of TP901-1 BppU-BppL complex | |||||||||
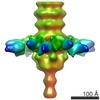
 マップデータ マップデータ | EM map of TP9101-1 BppU-BppL complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bacteriophage / TP9101-1 / BppU / BppL / ORF 48 / ORF 49 | |||||||||
| 生物種 |  Lactococcus phage TP901-1 (ファージ) Lactococcus phage TP901-1 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 24.8 Å | |||||||||
 データ登録者 データ登録者 | Bron P | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2010 ジャーナル: J Biol Chem / 年: 2010タイトル: Structure and molecular assignment of lactococcal phage TP901-1 baseplate. 著者: Cecilia Bebeacua / Patrick Bron / Livia Lai / Christina Skovgaard Vegge / Lone Brøndsted / Silvia Spinelli / Valérie Campanacci / David Veesler / Marin van Heel / Christian Cambillau /  要旨: P335 lactococcal phages infect the gram(+) bacterium Lactococcus lactis using a large multiprotein complex located at the distal part of the tail and termed baseplate (BP). The BP harbors the ...P335 lactococcal phages infect the gram(+) bacterium Lactococcus lactis using a large multiprotein complex located at the distal part of the tail and termed baseplate (BP). The BP harbors the receptor-binding proteins (RBPs), which allow the specific recognition of saccharidic receptors localized on the host cell surface. We report here the electron microscopic structure of the phage TP901-1 wild-type BP as well as those of two mutants bppL (-) and bppU(-), lacking BppL (the RBPs) or both peripheral BP components (BppL and BppU), respectively. We also achieved an electron microscopic reconstruction of a partial BP complex, formed by BppU and BppL. This complex exhibits a tripod shape and is composed of nine BppLs and three BppUs. These structures, combined with light-scattering measurements, led us to propose that the TP901-1 BP harbors six tripods at its periphery, located around the central tube formed by ORF46 (Dit) hexamers, at its proximal end, and a ORF47 (Tal) trimer at its distal extremity. A total of 54 BppLs (18 RBPs) are thus available to mediate host anchoring with a large apparent avidity. TP901-1 BP exhibits an infection-ready conformation and differs strikingly from the lactococcal phage p2 BP, bearing only 6 RBPs, and which needs a conformational change to reach its activated state. The comparison of several Siphoviridae structures uncovers a close organization of their central BP core whereas striking differences occur at the periphery, leading to diverse mechanisms of host recognition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1792.map.gz emd_1792.map.gz | 2.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1792-v30.xml emd-1792-v30.xml emd-1792.xml emd-1792.xml | 10.1 KB 10.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-1792.png EMD-1792.png | 46 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1792 http://ftp.pdbj.org/pub/emdb/structures/EMD-1792 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1792 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1792 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1792_validation.pdf.gz emd_1792_validation.pdf.gz | 201.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1792_full_validation.pdf.gz emd_1792_full_validation.pdf.gz | 200.4 KB | 表示 | |
| XML形式データ |  emd_1792_validation.xml.gz emd_1792_validation.xml.gz | 5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1792 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1792 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1792 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1792 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1792.map.gz / 形式: CCP4 / 大きさ: 2.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1792.map.gz / 形式: CCP4 / 大きさ: 2.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM map of TP9101-1 BppU-BppL complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : BppU/BppL complex from Lactococcal Phage TP901-1
| 全体 | 名称: BppU/BppL complex from Lactococcal Phage TP901-1 |
|---|---|
| 要素 |
|
-超分子 #1000: BppU/BppL complex from Lactococcal Phage TP901-1
| 超分子 | 名称: BppU/BppL complex from Lactococcal Phage TP901-1 / タイプ: sample / ID: 1000 / 集合状態: one trimer of BppU and three trimers of BppL / Number unique components: 2 |
|---|
-分子 #1: BppU
| 分子 | 名称: BppU / タイプ: protein_or_peptide / ID: 1 / Name.synonym: ORF48 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Lactococcus phage TP901-1 (ファージ) Lactococcus phage TP901-1 (ファージ) |
| 組換発現 | 生物種:  |
-分子 #2: BppL
| 分子 | 名称: BppL / タイプ: protein_or_peptide / ID: 2 / Name.synonym: ORF49 / コピー数: 3 / 集合状態: Trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Lactococcus phage TP901-1 (ファージ) Lactococcus phage TP901-1 (ファージ) |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 10 mM Hepes, 150 mM NaCl, 1 mM PMSF |
| 染色 | タイプ: NEGATIVE 詳細: Grids with adsorbed protein floated on 1% uranyl acetate for 1 min. |
| グリッド | 詳細: 400 mesh cupper griid |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2200FS |
|---|---|
| アライメント法 | Legacy - 非点収差: Astigmatism was corrected at 150,000 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: Omega filter エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 詳細 | Low-dose imaging |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 10 µm / 実像数: 8 / 平均電子線量: 18 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 50000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 0.4 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: JEOL |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 24.8 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: IMAGIC V / 使用した粒子像数: 8314 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 詳細 | The trimers were separately fitted by manual docking and refined using the docking program integrated into Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















