+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1734 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Rubisco in complex with Rubisco large subunit methyltransferase | |||||||||
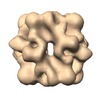
 マップデータ マップデータ | Cryo-EM volume of RuBisCO | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Rubisco / carbon fixation / methylation | |||||||||
| 生物種 |  Spinacia oleracea (ホウレンソウ) Spinacia oleracea (ホウレンソウ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 12.0 Å | |||||||||
 データ登録者 データ登録者 | Raunser S / Magnani R / Huang Z / Houtz RL / Trievel RC / Penczek PA / Walz T | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2009 ジャーナル: Proc Natl Acad Sci U S A / 年: 2009タイトル: Rubisco in complex with Rubisco large subunit methyltransferase. 著者: Stefan Raunser / Roberta Magnani / Zhong Huang / Robert L Houtz / Raymond C Trievel / Pawel A Penczek / Thomas Walz /  要旨: SET domain protein lysine methyltransferases (PKMT) are a structurally unique class of enzymes that catalyze the specific methylation of lysine residues in a number of different substrates. ...SET domain protein lysine methyltransferases (PKMT) are a structurally unique class of enzymes that catalyze the specific methylation of lysine residues in a number of different substrates. Especially histone-specific SET domain PKMTs have received widespread attention because of their roles in the regulation of epigenetic gene expression and the development of some cancers. Rubisco large subunit methyltransferase (RLSMT) is a chloroplast-localized SET domain PKMT responsible for the formation of trimethyl-lysine-14 in the large subunit of Rubisco, an essential photosynthetic enzyme. Here, we have used cryoelectron microscopy to produce an 11-A density map of the Rubisco-RLSMT complex. The atomic model of the complex, obtained by fitting crystal structures of Rubisco and RLSMT into the density map, shows that the extensive contact regions between the 2 proteins are mainly mediated by hydrophobic residues and leucine-rich repeats. It further provides insights into potential conformational changes that may occur during substrate binding and catalysis. This study presents the first structural analysis of a SET domain PKMT in complex with its intact polypeptide substrate. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1734.map.gz emd_1734.map.gz | 1.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1734-v30.xml emd-1734-v30.xml emd-1734.xml emd-1734.xml | 10 KB 10 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-1734.tif EMD-1734.tif | 763.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1734 http://ftp.pdbj.org/pub/emdb/structures/EMD-1734 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1734 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1734 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1734_validation.pdf.gz emd_1734_validation.pdf.gz | 229.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1734_full_validation.pdf.gz emd_1734_full_validation.pdf.gz | 228.4 KB | 表示 | |
| XML形式データ |  emd_1734_validation.xml.gz emd_1734_validation.xml.gz | 4.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1734 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1734 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1734 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1734 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1734.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1734.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM volume of RuBisCO | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Spinach RuBisCO
| 全体 | 名称: Spinach RuBisCO |
|---|---|
| 要素 |
|
-超分子 #1000: Spinach RuBisCO
| 超分子 | 名称: Spinach RuBisCO / タイプ: sample / ID: 1000 詳細: The sample was thawed from storage at -80 degrees Celsius before being loaded onto the grid. 集合状態: Hexadecamer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 534 KDa / 理論値: 534 KDa |
-分子 #1: RuBisCO
| 分子 | 名称: RuBisCO / タイプ: protein_or_peptide / ID: 1 / Name.synonym: RuBisCO / コピー数: 1 / 集合状態: Hexadecamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Spinacia oleracea (ホウレンソウ) / 別称: Spinach / 組織: Stroma / 細胞: Plant leaves / Organelle: Chloroplast / 細胞中の位置: Stroma Spinacia oleracea (ホウレンソウ) / 別称: Spinach / 組織: Stroma / 細胞: Plant leaves / Organelle: Chloroplast / 細胞中の位置: Stroma |
| 分子量 | 実験値: 534 KDa / 理論値: 534 KDa |
| 組換発現 | 生物種:  Spinacia oleracea (ホウレンソウ) Spinacia oleracea (ホウレンソウ) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 50 mM Tris-HCl pH 8, 5 mM MgCl2, 1 mM EDTA |
| グリッド | 詳細: Quantifoil grids 2 um holes |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: OTHER / 詳細: Vitrification instrument: Vitrobot / 手法: Blot for 1 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 110 / 平均電子線量: 15 e/Å2 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 51159 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Side entry liquid nitrogen-cooled cryo specimen holder. 試料ホルダーモデル: OTHER |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | The particles were selected interactively at the computer terminal. |
|---|---|
| CTF補正 | 詳細: CTF correction of each particle |
| 最終 再構成 | 想定した対称性 - 点群: D4 (2回x4回 2面回転対称) アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 12.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPARX / 詳細: D4 symmetry applied to final reconstruction / 使用した粒子像数: 29000 |
| 最終 角度割当 | 詳細: SPIDER:theta 45 degrees, phi 45 degrees |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: SITUS |
| 詳細 | Protocol: Rigid Body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: R-factor |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















