+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Virus-like Particle based on PVY coat protein with dC40 deletion with stacked-ring architecture | ||||||||||||
 マップデータ マップデータ | final VLPdC40:r sharp symmetric cryoEM map (C8) after Phenix density modification | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | stacked-ring / dC40 / VLP / Potyvirus / PVY / VIRUS LIKE PARTICLE | ||||||||||||
| 機能・相同性 | Potyvirus coat protein / Potyvirus coat protein / viral capsid / Genome polyprotein 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Potato virus Y strain NTN (ウイルス) Potato virus Y strain NTN (ウイルス) | ||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||
 データ登録者 データ登録者 | Kavcic L / Kezar A / Podobnik M | ||||||||||||
| 資金援助 |  スロベニア, スロベニア,  チェコ, 3件 チェコ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Commun Chem / 年: 2024 ジャーナル: Commun Chem / 年: 2024タイトル: From structural polymorphism to structural metamorphosis of the coat protein of flexuous filamentous potato virus Y. 著者: Luka Kavčič / Andreja Kežar / Neža Koritnik / Magda Tušek Žnidarič / Tajda Klobučar / Žiga Vičič / Franci Merzel / Ellie Holden / Justin L P Benesch / Marjetka Podobnik /   要旨: The structural diversity and tunability of the capsid proteins (CPs) of various icosahedral and rod-shaped viruses have been well studied and exploited in the development of smart hybrid ...The structural diversity and tunability of the capsid proteins (CPs) of various icosahedral and rod-shaped viruses have been well studied and exploited in the development of smart hybrid nanoparticles. However, the potential of CPs of the wide-spread flexuous filamentous plant viruses remains to be explored. Here, we show that we can control the shape, size, RNA encapsidation ability, symmetry, stability and surface functionalization of nanoparticles through structure-based design of CP from potato virus Y (PVY). We provide high-resolution insight into CP-based self-assemblies, ranging from large polymorphic or monomorphic filaments to smaller annular, cubic or spherical particles. Furthermore, we show that we can prevent CP self-assembly in bacteria by fusion with a cleavable protein, enabling controlled nanoparticle formation in vitro. Understanding the remarkable structural diversity of PVY CP not only provides possibilities for the production of biodegradable nanoparticles, but may also advance future studies of CP's polymorphism in a biological context. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17049.map.gz emd_17049.map.gz | 13.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17049-v30.xml emd-17049-v30.xml emd-17049.xml emd-17049.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_17049.png emd_17049.png | 85.8 KB | ||
| マスクデータ |  emd_17049_msk_1.map emd_17049_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17049.cif.gz emd-17049.cif.gz | 6.4 KB | ||
| その他 |  emd_17049_additional_1.map.gz emd_17049_additional_1.map.gz emd_17049_additional_2.map.gz emd_17049_additional_2.map.gz emd_17049_half_map_1.map.gz emd_17049_half_map_1.map.gz emd_17049_half_map_2.map.gz emd_17049_half_map_2.map.gz | 13.3 MB 75.4 MB 75.7 MB 75.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17049 http://ftp.pdbj.org/pub/emdb/structures/EMD-17049 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17049 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17049 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17049_validation.pdf.gz emd_17049_validation.pdf.gz | 730.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17049_full_validation.pdf.gz emd_17049_full_validation.pdf.gz | 729.8 KB | 表示 | |
| XML形式データ |  emd_17049_validation.xml.gz emd_17049_validation.xml.gz | 13.7 KB | 表示 | |
| CIF形式データ |  emd_17049_validation.cif.gz emd_17049_validation.cif.gz | 16.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17049 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17049 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17049 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17049 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8opdMC  8opaC  8opbC  8opcC  8opeC  8opfC  8opgC  8ophC  8opjC  8opkC  8oplC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17049.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17049.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | final VLPdC40:r sharp symmetric cryoEM map (C8) after Phenix density modification | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.063 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17049_msk_1.map emd_17049_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
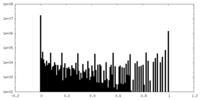
| 密度ヒストグラム |
-追加マップ: VLPdC40:r cryoEM map (C8) after imposing helical symmetry
| ファイル | emd_17049_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | VLPdC40:r cryoEM map (C8) after imposing helical symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
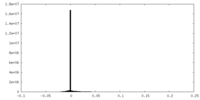
| 密度ヒストグラム |
-追加マップ: VLPdC40:r raw cryoEM map (C8)
| ファイル | emd_17049_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | VLPdC40:r raw cryoEM map (C8) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
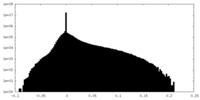
| 密度ヒストグラム |
-ハーフマップ: VLPdC40:r unfiltered half cryoEM map 2
| ファイル | emd_17049_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | VLPdC40:r unfiltered half cryoEM map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
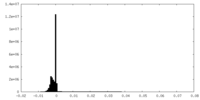
| 密度ヒストグラム |
-ハーフマップ: VLPdC40:r unfiltered half cryoEM map 1
| ファイル | emd_17049_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | VLPdC40:r unfiltered half cryoEM map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Virus-like particle from dC40 coat protein
| 全体 | 名称: Virus-like particle from dC40 coat protein |
|---|---|
| 要素 |
|
-超分子 #1: Virus-like particle from dC40 coat protein
| 超分子 | 名称: Virus-like particle from dC40 coat protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: The sample contains 2 forms of filaments. |
|---|---|
| 由来(天然) | 生物種:  Potato virus Y strain NTN (ウイルス) Potato virus Y strain NTN (ウイルス) |
-分子 #1: Genome polyprotein (Fragment)
| 分子 | 名称: Genome polyprotein (Fragment) / タイプ: protein_or_peptide / ID: 1 詳細: Due to flexibility, structural model could only be built from T41 to K221. コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Potato virus Y strain NTN (ウイルス) / 株: NTN Potato virus Y strain NTN (ウイルス) / 株: NTN |
| 分子量 | 理論値: 25.561 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GNDTIDAGGS TKKDAKQEQG SIQPNLNKEK EKDVNVGTSG THTVPRIKAI TSKMRMPKSK GATVLNLEHL LEYAPQQIDI SNTRATQSQ FDTWYEAVQL AYDIGETEMP TVMNGLMVWC IENGTSPNIN GVWVMMDGDE QVEYPLKPIV ENAKPTLRQI M AHFSDVAE ...文字列: GNDTIDAGGS TKKDAKQEQG SIQPNLNKEK EKDVNVGTSG THTVPRIKAI TSKMRMPKSK GATVLNLEHL LEYAPQQIDI SNTRATQSQ FDTWYEAVQL AYDIGETEMP TVMNGLMVWC IENGTSPNIN GVWVMMDGDE QVEYPLKPIV ENAKPTLRQI M AHFSDVAE AYIEMRNKKE PYMPRYGLVR NLRDGSLARY AFDFYEVTSR TPVRAREAHI QMKAAALKS UniProtKB: Genome polyprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 1.8 mM KH2PO4, 10.1 mM Na2HPO4, 140 mM NaCl, 2.7 mM KCl, pH 7.4 |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / 実像数: 4805 / 平均露光時間: 1.02 sec. / 平均電子線量: 84.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.3 µm / 最小 デフォーカス(公称値): 0.4 µm / 倍率(公称値): 75000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 43.29 Å 想定した対称性 - らせんパラメータ - ΔΦ: 15.04 ° 想定した対称性 - らせんパラメータ - 軸対称性: C8 (8回回転対称) 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF ソフトウェア:
詳細: Final cryoEM map was obtained by Phenix density modification. 使用した粒子像数: 399617 | ||||||
|---|---|---|---|---|---|---|---|
| Segment selection | 選択した数: 2966850 / ソフトウェア - 名称: crYOLO 詳細: CrYOLO filament picking based on pre-trained neural network model | ||||||
| 初期モデル | モデルのタイプ: NONE / 詳細: Ab-initio model in cryoSPARC | ||||||
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー

































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)