+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of subfamily III haloalkane dehalogenase DhmeA from Haloferax mediterranei | |||||||||
 マップデータ マップデータ | Main cryo-EM map of DhmeA enzyme. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | haloalkane dehalogenase / HYDROLASE | |||||||||
| 生物種 |  Haloferax mediterranei (古細菌) Haloferax mediterranei (古細菌) | |||||||||
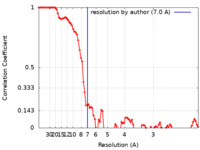
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.0 Å | |||||||||
 データ登録者 データ登録者 | Marek M / Novacek J / Polak M / Chmelova K | |||||||||
| 資金援助 |  チェコ, 1件 チェコ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Protein Sci / 年: 2023 ジャーナル: Protein Sci / 年: 2023タイトル: Multimeric structure of a subfamily III haloalkane dehalogenase-like enzyme solved by combination of cryo-EM and x-ray crystallography. 著者: Klaudia Chmelova / Tadeja Gao / Martin Polak / Andrea Schenkmayerova / Tristan I Croll / Tanvir R Shaikh / Jana Skarupova / Radka Chaloupkova / Kay Diederichs / Randy J Read / Jiri Damborsky ...著者: Klaudia Chmelova / Tadeja Gao / Martin Polak / Andrea Schenkmayerova / Tristan I Croll / Tanvir R Shaikh / Jana Skarupova / Radka Chaloupkova / Kay Diederichs / Randy J Read / Jiri Damborsky / Jiri Novacek / Martin Marek /    要旨: Haloalkane dehalogenase (HLD) enzymes employ an S 2 nucleophilic substitution mechanism to erase halogen substituents in diverse organohalogen compounds. Subfamily I and II HLDs are well- ...Haloalkane dehalogenase (HLD) enzymes employ an S 2 nucleophilic substitution mechanism to erase halogen substituents in diverse organohalogen compounds. Subfamily I and II HLDs are well-characterized enzymes, but the mode and purpose of multimerization of subfamily III HLDs are unknown. Here we probe the structural organization of DhmeA, a subfamily III HLD-like enzyme from the archaeon Haloferax mediterranei, by combining cryo-electron microscopy (cryo-EM) and x-ray crystallography. We show that full-length wild-type DhmeA forms diverse quaternary structures, ranging from small oligomers to large supramolecular ring-like assemblies of various sizes and symmetries. We optimized sample preparation steps, enabling three-dimensional reconstructions of an oligomeric species by single-particle cryo-EM. Moreover, we engineered a crystallizable mutant (DhmeA ) that provided diffraction-quality crystals. The 3.3 Å crystal structure reveals that DhmeA forms a ring-like 20-mer structure with outer and inner diameter of ~200 and ~80 Å, respectively. An enzyme homodimer represents a basic repeating building unit of the crystallographic ring. Three assembly interfaces (dimerization, tetramerization, and multimerization) were identified to form the supramolecular ring that displays a negatively charged exterior, while its interior part harboring catalytic sites is positively charged. Localization and exposure of catalytic machineries suggest a possible processing of large negatively charged macromolecular substrates. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16998.map.gz emd_16998.map.gz | 30.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16998-v30.xml emd-16998-v30.xml emd-16998.xml emd-16998.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16998_fsc.xml emd_16998_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16998.png emd_16998.png | 22.5 KB | ||
| Filedesc metadata |  emd-16998.cif.gz emd-16998.cif.gz | 4.9 KB | ||
| その他 |  emd_16998_half_map_1.map.gz emd_16998_half_map_1.map.gz emd_16998_half_map_2.map.gz emd_16998_half_map_2.map.gz | 59.2 MB 59.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16998 http://ftp.pdbj.org/pub/emdb/structures/EMD-16998 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16998 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16998 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16998_validation.pdf.gz emd_16998_validation.pdf.gz | 671.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16998_full_validation.pdf.gz emd_16998_full_validation.pdf.gz | 670.9 KB | 表示 | |
| XML形式データ |  emd_16998_validation.xml.gz emd_16998_validation.xml.gz | 16.6 KB | 表示 | |
| CIF形式データ |  emd_16998_validation.cif.gz emd_16998_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16998 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16998 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16998 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16998 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16998.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16998.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main cryo-EM map of DhmeA enzyme. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.061 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: First half map.
| ファイル | emd_16998_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | First half map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Second half map.
| ファイル | emd_16998_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Second half map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hexameric form of haloalkane dehalogenase DhmeA from Haloferax me...
| 全体 | 名称: Hexameric form of haloalkane dehalogenase DhmeA from Haloferax mediterranei |
|---|---|
| 要素 |
|
-超分子 #1: Hexameric form of haloalkane dehalogenase DhmeA from Haloferax me...
| 超分子 | 名称: Hexameric form of haloalkane dehalogenase DhmeA from Haloferax mediterranei タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Haloferax mediterranei (古細菌) Haloferax mediterranei (古細菌) |
-分子 #1: A subfamily III haloalkane dehalogenase DhmeA from Haloferax medi...
| 分子 | 名称: A subfamily III haloalkane dehalogenase DhmeA from Haloferax mediterranei タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloferax mediterranei (古細菌) Haloferax mediterranei (古細菌) |
| 配列 | 文字列: MSSASSNARD EVIAAIHEEA DWVDRTVYPF ESRCIGLSSG AVHYIDEGPD DGGRETLLML HGNPTWSFLY RHLVRDLRDE YRCVALDYLG FGLSERPTDF SYRPEDHADV VEEFIDELGL EDVVLVGHDW GGPIGFSYAI DHPENVGGLV VMNTWMWPVS DDKHFSRFSK ...文字列: MSSASSNARD EVIAAIHEEA DWVDRTVYPF ESRCIGLSSG AVHYIDEGPD DGGRETLLML HGNPTWSFLY RHLVRDLRDE YRCVALDYLG FGLSERPTDF SYRPEDHADV VEEFIDELGL EDVVLVGHDW GGPIGFSYAI DHPENVGGLV VMNTWMWPVS DDKHFSRFSK LLGGRIGREL CERYDLFTRV IMPMGFADRS RFTESAREQY RAANRGDRTG TGIFPQAILG SRAWLSSLWE QRDNIADIPA RIIWGMEDSA FRPAELRTFE ALFEDSSTVR LYGVGHYVPE EFGSDLVPLV R EFLEEVLE VLFQ |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: FEI FALCON II (4k x 4k) #0 - 平均電子線量: 49.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #1 - 平均電子線量: 76.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)