+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1696 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Outer Dynein arms with a Microtubule Doublet in the absence of nucleotides | |||||||||
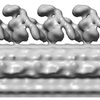
 マップデータ マップデータ | This is an image of outer dynein arms on a microtubule doublet in flagella | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Dynein / flagella / tubulin / microtubule / motility | |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 37.0 Å | |||||||||
 データ登録者 データ登録者 | Movassagh T / Bui KH / Sakakibara H / Oiwa K / Ishikawa T | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2010 ジャーナル: Nat Struct Mol Biol / 年: 2010タイトル: Nucleotide-induced global conformational changes of flagellar dynein arms revealed by in situ analysis. 著者: Tandis Movassagh / Khanh Huy Bui / Hitoshi Sakakibara / Kazuhiro Oiwa / Takashi Ishikawa /  要旨: Outer and inner dynein arms generate force for the flagellar/ciliary bending motion. Although nucleotide-induced structural change of dynein heavy chains (the ATP-driven motor) was proven in vitro, ...Outer and inner dynein arms generate force for the flagellar/ciliary bending motion. Although nucleotide-induced structural change of dynein heavy chains (the ATP-driven motor) was proven in vitro, our lack of knowledge in situ has precluded an understanding of the bending mechanism. Here we reveal nucleotide-induced global structural changes of the outer and inner dynein arms of Chlamydomonas reinhardtii flagella in situ using electron cryotomography. The ATPase domains of the dynein heavy chains move toward the distal end, and the N-terminal tail bends sharply during product release. This motion could drive the adjacent microtubule to cause a sliding motion. In contrast to in vitro results, in the presence of nucleotides, outer dynein arms coexist as clusters of apo or nucleotide-bound forms in situ. This implies a cooperative switching, which may be related to the mechanism of bending. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1696.map.gz emd_1696.map.gz | 2.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1696-v30.xml emd-1696-v30.xml emd-1696.xml emd-1696.xml | 9.3 KB 9.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1696apo.jpg 1696apo.jpg | 72.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1696 http://ftp.pdbj.org/pub/emdb/structures/EMD-1696 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1696 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1696 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1696_validation.pdf.gz emd_1696_validation.pdf.gz | 181.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1696_full_validation.pdf.gz emd_1696_full_validation.pdf.gz | 180.4 KB | 表示 | |
| XML形式データ |  emd_1696_validation.xml.gz emd_1696_validation.xml.gz | 4.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1696 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1696 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1696 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1696 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1696.map.gz / 形式: CCP4 / 大きさ: 3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1696.map.gz / 形式: CCP4 / 大きさ: 3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is an image of outer dynein arms on a microtubule doublet in flagella | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6.85 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Outer dynein arm, microtubule doublet
| 全体 | 名称: Outer dynein arm, microtubule doublet |
|---|---|
| 要素 |
|
-超分子 #1000: Outer dynein arm, microtubule doublet
| 超分子 | 名称: Outer dynein arm, microtubule doublet / タイプ: sample / ID: 1000 / 詳細: The sample was flagella in situ 集合状態: One heterotrimer binds to one microtubule doublet Number unique components: 1 |
|---|
-超分子 #1: Flagella
| 超分子 | 名称: Flagella / タイプ: organelle_or_cellular_component / ID: 1 / Name.synonym: Outer dynein arm and Microtubule doublet / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  別称: Green algae / 細胞: Chlamydomonas reinhardtii / Organelle: Flagella / 細胞中の位置: Axoneme |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 詳細: 30 mM HEPES (pH 7.4), 5 mM MgSO4, 1 mM DTT, 1 mM EGTA, 50 mM Potassium Acetate, 0.5% (w/v) Polyethylene Glycol (MW 20,000) |
|---|---|
| グリッド | 詳細: 300 mesh lacey carbon grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: OTHER / 詳細: Vitrification instrument: Vitrobot / 手法: Blot for 3 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 平均: 100 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF エネルギーフィルター - エネルギー下限: 10.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC GATAN / デジタル化 - サンプリング間隔: 6.85 µm / 実像数: 1200 / 平均電子線量: 30 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt series - Axis1 - Min angle: -60 ° / Tilt series - Axis1 - Max angle: 60 ° |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Average number of projections used in the 3D reconstructions: 6000. Average number of class averages: 1. |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 37.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称:  IMOD IMOD |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)