+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SA11 Rotavirus Non-tripsinized Triple Layered Particle | |||||||||
 マップデータ マップデータ | Reuploaded main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Rotavirus / dsRNA virus / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral intermediate capsid / host cell endoplasmic reticulum lumen / T=13 icosahedral viral capsid / T=2 icosahedral viral capsid / viral inner capsid / viral outer capsid / viral nucleocapsid / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / viral envelope ...viral intermediate capsid / host cell endoplasmic reticulum lumen / T=13 icosahedral viral capsid / T=2 icosahedral viral capsid / viral inner capsid / viral outer capsid / viral nucleocapsid / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / viral envelope / structural molecule activity / RNA binding / metal ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Rotavirus (ウイルス) / Rotavirus (ウイルス) /  Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) | |||||||||
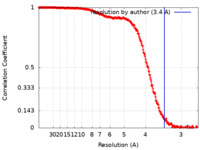
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Asensio-Cob D / Perez-Mata C / Gomez-Blanco J / Vargas J / Rodriguez JM / Luque D | |||||||||
| 資金援助 |  スペイン, 1件 スペイン, 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2025 ジャーナル: PLoS Pathog / 年: 2025タイトル: Structural determinants of rotavirus proteolytic activation. 著者: Dunia Asensio-Cob / Carlos P Mata / Josue Gomez-Blanco / Javier Vargas / Javier M Rodriguez / Daniel Luque /    要旨: The infectivity of rotavirus (RV), the leading cause of childhood diarrhea, hinges on the activation of viral particles through the proteolysis of the spike protein by trypsin-like proteases in the ...The infectivity of rotavirus (RV), the leading cause of childhood diarrhea, hinges on the activation of viral particles through the proteolysis of the spike protein by trypsin-like proteases in the host intestinal lumen. In order to determine the structural basis of trypsin activation, we have used cryogenic electron microscopy (cryo-EM) and advanced image processing methods to compare uncleaved and cleaved RV particles. We find that the conformation of the non-proteolyzed spike is constrained by the position of loops that surround its structure, linking the lectin domains of the spike head to its body. The proteolysis of these loops removes this structural constraint, thereby enabling the spike to undergo the necessary conformational changes required for cell membrane penetration. Thus, these loops function as regulatory elements to ensure that the spike protein is activated precisely when and where it is needed to facilitate a successful infection. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16954.map.gz emd_16954.map.gz | 1.5 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16954-v30.xml emd-16954-v30.xml emd-16954.xml emd-16954.xml | 24.2 KB 24.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16954_fsc.xml emd_16954_fsc.xml | 36.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16954.png emd_16954.png | 191.5 KB | ||
| Filedesc metadata |  emd-16954.cif.gz emd-16954.cif.gz | 7.6 KB | ||
| その他 |  emd_16954_half_map_1.map.gz emd_16954_half_map_1.map.gz emd_16954_half_map_2.map.gz emd_16954_half_map_2.map.gz | 1.6 GB 1.5 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16954 http://ftp.pdbj.org/pub/emdb/structures/EMD-16954 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16954 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16954 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16954.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16954.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reuploaded main map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_16954_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16954_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rotavirus A
| 全体 | 名称:  Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
|---|---|
| 要素 |
|
-超分子 #1: Rotavirus A
| 超分子 | 名称: Rotavirus A / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 / 詳細: Rotavirus SA11 Non-trypsinized TLP / NCBI-ID: 28875 / 生物種: Rotavirus A / Sci species strain: SA11 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Chlorocebus aethiops (ミドリザル) Chlorocebus aethiops (ミドリザル) |
| 分子量 | 理論値: 92 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: TLP / 直径: 1000.0 Å |
-分子 #1: Inner capsid protein VP2
| 分子 | 名称: Inner capsid protein VP2 / タイプ: protein_or_peptide / ID: 1 / 詳細: Inner core protein / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rotavirus (ウイルス) Rotavirus (ウイルス) |
| 分子量 | 理論値: 102.853406 KDa |
| 組換発現 | 生物種:  Chlorocebus aethiops (ミドリザル) Chlorocebus aethiops (ミドリザル) |
| 配列 | 文字列: MAYRKRGARR ETNLKQDERM QEKEDSKNIN NDSPKSQLSE KVLSKKEEII TDNQEEVKIS DEVKKSNKEE SKQLLEVLKT KEEHQKEVQ YEILQKTIPT FEPKESILKK LEDIKPEQAK KQTKLFRIFE PKQLPIYRAN GERELRNRWY WKLKRDTLPD G DYDVREYF ...文字列: MAYRKRGARR ETNLKQDERM QEKEDSKNIN NDSPKSQLSE KVLSKKEEII TDNQEEVKIS DEVKKSNKEE SKQLLEVLKT KEEHQKEVQ YEILQKTIPT FEPKESILKK LEDIKPEQAK KQTKLFRIFE PKQLPIYRAN GERELRNRWY WKLKRDTLPD G DYDVREYF LNLYDQVLME MPDYLLLKDM AVENKNSRDA GKVVDSETAA ICDAIFQDEE TEGAVRRFIA EMRQRVQADR NV VNYPSIL HPIDHAFNEY FLQHQLVEPL NNDIIFNYIP ERIRNDVNYI LNMDRNLPST ARYIRPNLLQ DRLNLHDNFE SLW DTITTS NYILARSVVP DLKELVSTEA QIQKMSQDLQ LEALTIQSET QFLTGINSQA ANDCFKTLIA AMLSQRTMSL DFVT TNYMS LISGMWLLTV IPNDMFIRES LVACQLAIIN TIVYPAFGMQ RMHYRNGDPQ TPFQIAEQQI QNFQVANWLH FVNYN QFRQ VVIDGVLNQV LNDNIRNGHV VNQLMEALMQ LSRQQFPTMP VDYKRSIQRG ILLLSNRLGQ LVDLTRLLSY NYETLM ACI TMNMQHVQTL TTEKLQLTSV TSLCMLIGNA TVIPSPQTLF HYYNVNVNFH SNYNERINDA VAIITAANRL NLYQKKM KS IVEDFLKRLQ IFDVARVPDD QMYRLRDRLR LLPVEIRRLD IFNLIAMNME QIERASDKIA QGVIIAYRDM QLERDEMY G YVNIARNLDG FQQINLEELM RSGDYAQITN MLLNNQPVAL VGALPFITDS SVISLIAKLD ATVFAQIVKL RKVDTLKPI LYKINSDSND FYLVANYDWI PTSTTKVYKQ VPQQFDFRAS MHMLTSNLTF TVYSDLLAFV SADTVEPINA VAFDNMRIMN EL UniProtKB: Inner capsid protein VP2 |
-分子 #2: Outer capsid glycoprotein VP7
| 分子 | 名称: Outer capsid glycoprotein VP7 / タイプ: protein_or_peptide / ID: 2 / 詳細: Outer capsid glycoprotein VP7 / コピー数: 13 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rotavirus (ウイルス) Rotavirus (ウイルス) |
| 分子量 | 理論値: 37.222602 KDa |
| 組換発現 | 生物種:  Chlorocebus aethiops (ミドリザル) Chlorocebus aethiops (ミドリザル) |
| 配列 | 文字列: MYGIEYTTVL TFLISIILLN YILKSLTRIM DFIIYRFLFI IVILSPFLRA QNYGINLPIT GSMDTAYANS TQEETFLTST LCLYYPTEA ATEINDNSWK DTLSQLFLTK GWPTGSVYFK EYTNIASFSV DPQLYCDYNV VLMKYDATLQ LDMSELADLI L NEWLCNPM ...文字列: MYGIEYTTVL TFLISIILLN YILKSLTRIM DFIIYRFLFI IVILSPFLRA QNYGINLPIT GSMDTAYANS TQEETFLTST LCLYYPTEA ATEINDNSWK DTLSQLFLTK GWPTGSVYFK EYTNIASFSV DPQLYCDYNV VLMKYDATLQ LDMSELADLI L NEWLCNPM DITLYYYQQT DEANKWISMG SSCTIKVCPL NTQTLGIGCL TTDATTFEEV ATAEKLVITD VVDGVNHKLD VT TATCTIR NCKKLGPREN VAVIQVGGSD ILDITADPTT APQTERMMRI NWKKWWQVFY TVVDYVDQII QVMSKRSRSL NSA AFYYRV UniProtKB: Outer capsid glycoprotein VP7 |
-分子 #3: Intermediate capsid protein VP6
| 分子 | 名称: Intermediate capsid protein VP6 / タイプ: protein_or_peptide / ID: 3 / 詳細: Intermediate capsid protein VP6 / コピー数: 13 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rotavirus (ウイルス) Rotavirus (ウイルス) |
| 分子量 | 理論値: 44.910738 KDa |
| 組換発現 | 生物種:  Chlorocebus aethiops (ミドリザル) Chlorocebus aethiops (ミドリザル) |
| 配列 | 文字列: MDVLYSLSKT LKDARDKIVE GTLYSNVSDL IQQFNQMIIT MNGNEFQTGG IGNLPIRNWN FNFGLLGTTL LNLDANYVET ARNTIDYFV DFVDNVCMDE MVRESQRNGI APQSDSLRKL SAIKFKRINF DNSSEYIENW NLQNRRQRTG FTFHKPNIFP Y SASFTLNR ...文字列: MDVLYSLSKT LKDARDKIVE GTLYSNVSDL IQQFNQMIIT MNGNEFQTGG IGNLPIRNWN FNFGLLGTTL LNLDANYVET ARNTIDYFV DFVDNVCMDE MVRESQRNGI APQSDSLRKL SAIKFKRINF DNSSEYIENW NLQNRRQRTG FTFHKPNIFP Y SASFTLNR SQPAHDNLMG TMWLNAGSEI QVAGFDYSCA INAPANIQQF EHIVPLRRVL TTATITLLPD AERFSFPRVI NS ADGATTW FFNPVILRPN NVEVEFLLNG QIINTYQARF GTIVARNFDT IRLSFQLMRP PNMTPAVAVL FPNAQPFEHH ATV GLTLRI ESAVCESVLA DASETLLANV TSVRQEYAIP VGPVFPPGMN WTDLITNYSP SREDNLQRVF TVASIRSMLI K UniProtKB: Intermediate capsid protein VP6 |
-分子 #4: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 4 / コピー数: 26 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 10 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 5 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER/RHODIUM / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 295 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / 実像数: 1465 / 平均電子線量: 42.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)