+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 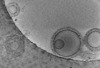 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Tomogram of GBP1 coatomers assembled on brain polar lipid-derived small unilamellar vesicles. | |||||||||
 マップデータ マップデータ | Electron cryotomogram of GBP1 coatomers on BPLE-derived SUVs. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | GBP1 / cryo-ET / liposome / coatomer / IMMUNE SYSTEM | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Kuhm TI / Jakobi AJ | |||||||||
| 資金援助 | European Union,  オランダ, 2件 オランダ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2025 ジャーナル: Nat Struct Mol Biol / 年: 2025タイトル: Structural basis of antimicrobial membrane coat assembly by human GBP1. 著者: Tanja Kuhm / Clémence Taisne / Cecilia de Agrela Pinto / Luca Gross / Evdokia A Giannopoulou / Stefan T Huber / Els Pardon / Jan Steyaert / Sander J Tans / Arjen J Jakobi /   要旨: Guanylate-binding proteins (GBPs) are interferon-inducible guanosine triphosphate hydrolases (GTPases) mediating host defense against intracellular pathogens. Their antimicrobial activity hinges on ...Guanylate-binding proteins (GBPs) are interferon-inducible guanosine triphosphate hydrolases (GTPases) mediating host defense against intracellular pathogens. Their antimicrobial activity hinges on their ability to self-associate and coat pathogen-associated compartments or cytosolic bacteria. Coat formation depends on GTPase activity but how nucleotide binding and hydrolysis prime coat formation remains unclear. Here, we report the cryo-electron microscopy structure of the full-length human GBP1 dimer in its guanine nucleotide-bound state and describe the molecular ultrastructure of the GBP1 coat on liposomes and bacterial lipopolysaccharide membranes. Conformational changes of the middle and GTPase effector domains expose the isoprenylated C terminus for membrane association. The α-helical middle domains form a parallel, crossover arrangement essential for coat formation and position the extended effector domain for intercalation into the lipopolysaccharide layer of gram-negative membranes. Nucleotide binding and hydrolysis create oligomeric scaffolds with contractile abilities that promote membrane extrusion and fragmentation. Our data offer a structural and mechanistic framework for understanding GBP1 effector functions in intracellular immunity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16815.map.gz emd_16815.map.gz | 1 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16815-v30.xml emd-16815-v30.xml emd-16815.xml emd-16815.xml | 10.6 KB 10.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16815.png emd_16815.png | 223.9 KB | ||
| マスクデータ |  emd_16815_msk_1.map emd_16815_msk_1.map emd_16815_msk_2.map emd_16815_msk_2.map | 1.2 GB 1.2 GB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16815.cif.gz emd-16815.cif.gz | 4.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16815 http://ftp.pdbj.org/pub/emdb/structures/EMD-16815 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16815 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16815 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16815_validation.pdf.gz emd_16815_validation.pdf.gz | 433.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16815_full_validation.pdf.gz emd_16815_full_validation.pdf.gz | 433.5 KB | 表示 | |
| XML形式データ |  emd_16815_validation.xml.gz emd_16815_validation.xml.gz | 5 KB | 表示 | |
| CIF形式データ |  emd_16815_validation.cif.gz emd_16815_validation.cif.gz | 5.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16815 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16815 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16815 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16815 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16815.map.gz / 形式: CCP4 / 大きさ: 1.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16815.map.gz / 形式: CCP4 / 大きさ: 1.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
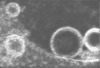
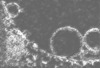
| 注釈 | Electron cryotomogram of GBP1 coatomers on BPLE-derived SUVs. | ||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6.15 Å | ||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16815_msk_1.map emd_16815_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
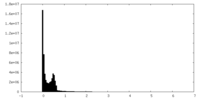
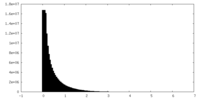
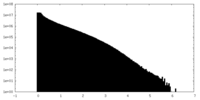
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_16815_msk_2.map emd_16815_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
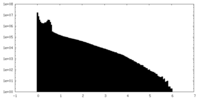
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Membrane-assembled coatomer formed by GDP-AlF3-stabilised GBP1 di...
| 全体 | 名称: Membrane-assembled coatomer formed by GDP-AlF3-stabilised GBP1 dimers on brain polar lipid-derived SUVs. |
|---|---|
| 要素 |
|
-超分子 #1: Membrane-assembled coatomer formed by GDP-AlF3-stabilised GBP1 di...
| 超分子 | 名称: Membrane-assembled coatomer formed by GDP-AlF3-stabilised GBP1 dimers on brain polar lipid-derived SUVs. タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 98 % / チャンバー内温度: 20 K / 装置: LEICA EM GP / 詳細: Blotted for 4 seconds from the carbon side.. |
| 詳細 | Membrane-assembled coatomer formed by GDP-AlF3-stabilised GBP1 dimers on brain polar lipid-derived SUVs. |
| 切片作成 | その他: NO SECTIONING |
| 位置合わせマーカー | Manufacturer: CMC Utrecht / 直径: 10 nm |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 61 / 平均露光時間: 3.0 sec. / 平均電子線量: 1.64 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 12000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 4.1 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 5.0 µm |
| 試料ステージ | 試料ホルダーモデル: JEOL 3200FSC CRYOHOLDER / ホルダー冷却材: NITROGEN |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: BACK PROJECTION / 解像度の算出法: OTHER / ソフトウェア - 名称:  IMOD / 使用した粒子像数: 61 IMOD / 使用した粒子像数: 61 |
|---|
 ムービー
ムービー コントローラー
コントローラー






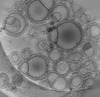

 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)