+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | RNA polymerase bound to purified in vitro transcribed regulatory RNA putL - inactive, open clamp state | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | RNA polymerase / transcriptional pausing / transcription termination / regulatory RNA / TRANSCRIPTION | |||||||||||||||
| 生物種 |  | |||||||||||||||
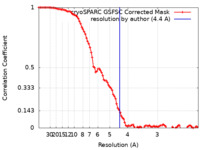
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | |||||||||||||||
 データ登録者 データ登録者 | Dey S / Weixlbaumer A | |||||||||||||||
| 資金援助 | European Union,  フランス, 4件 フランス, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2022 ジャーナル: Mol Cell / 年: 2022タイトル: Structural insights into RNA-mediated transcription regulation in bacteria. 著者: Sanjay Dey / Claire Batisse / Jinal Shukla / Michael W Webster / Maria Takacs / Charlotte Saint-André / Albert Weixlbaumer /  要旨: RNA can regulate its own synthesis without auxiliary proteins. For example, U-rich RNA sequences signal RNA polymerase (RNAP) to pause transcription and are required for transcript release at ...RNA can regulate its own synthesis without auxiliary proteins. For example, U-rich RNA sequences signal RNA polymerase (RNAP) to pause transcription and are required for transcript release at intrinsic terminators in all kingdoms of life. In contrast, the regulatory RNA putL suppresses pausing and termination in cis. However, how nascent RNA modulates its own synthesis remains largely unknown. We present cryo-EM reconstructions of RNAP captured during transcription of putL variants or an unrelated sequence at a U-rich pause site. Our results suggest how putL suppresses pausing and promotes its synthesis. We demonstrate that transcribing a U-rich sequence, a ubiquitous trigger of intrinsic termination, promotes widening of the RNAP nucleic-acid-binding channel. Widening destabilizes RNAP interactions with DNA and RNA to facilitate transcript dissociation reminiscent of intrinsic transcription termination. Surprisingly, RNAP remains bound to DNA after transcript release. Our results provide the structural framework to understand RNA-mediated intrinsic transcription termination. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15613.map.gz emd_15613.map.gz | 79.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15613-v30.xml emd-15613-v30.xml emd-15613.xml emd-15613.xml | 16.4 KB 16.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15613_fsc.xml emd_15613_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15613.png emd_15613.png | 41.1 KB | ||
| Filedesc metadata |  emd-15613.cif.gz emd-15613.cif.gz | 4.6 KB | ||
| その他 |  emd_15613_additional_1.map.gz emd_15613_additional_1.map.gz emd_15613_half_map_1.map.gz emd_15613_half_map_1.map.gz emd_15613_half_map_2.map.gz emd_15613_half_map_2.map.gz | 41.7 MB 77.7 MB 77.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15613 http://ftp.pdbj.org/pub/emdb/structures/EMD-15613 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15613 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15613 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15613_validation.pdf.gz emd_15613_validation.pdf.gz | 852.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15613_full_validation.pdf.gz emd_15613_full_validation.pdf.gz | 851.7 KB | 表示 | |
| XML形式データ |  emd_15613_validation.xml.gz emd_15613_validation.xml.gz | 17.6 KB | 表示 | |
| CIF形式データ |  emd_15613_validation.cif.gz emd_15613_validation.cif.gz | 22.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15613 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15613 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15613 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15613 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15613.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15613.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_15613_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15613_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15613_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia coli RNA polymerase and in vitro transcribed and puri...
| 全体 | 名称: Escherichia coli RNA polymerase and in vitro transcribed and purified putL RNA complex - inactive, open clamp state |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia coli RNA polymerase and in vitro transcribed and puri...
| 超分子 | 名称: Escherichia coli RNA polymerase and in vitro transcribed and purified putL RNA complex - inactive, open clamp state タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 435 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 12 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM Tris-glutamate pH 8.0, 50 mM K-glutamate, 10 mM Mg-glutamate, 0.001 mM ZnCl2, 2mM DTT |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GOLD / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV 詳細: Quantifoil UltrAuFoil R1.2/1.3 300 mesh holey gold grids were plasma cleaned on a Model 1070 (Fischione Instruments) for 30 sec at 70% power and with an 80% Argon and 20% Oxygen mixture prior ...詳細: Quantifoil UltrAuFoil R1.2/1.3 300 mesh holey gold grids were plasma cleaned on a Model 1070 (Fischione Instruments) for 30 sec at 70% power and with an 80% Argon and 20% Oxygen mixture prior to the application of 0.004 ml of sample. Grids were plunge frozen into liquid ethane using a Vitrobot mark IV (FEI) with 95% chamber humidity at 283K.. |
| 詳細 | The complex for cryo-EM analysis was prepared in vitro by mixing E. coli RNAP with DNA oligonucleotides and in vitro transcribed and purified putL RNA to mimic an RNAP EC halted at the U-rich pause (G93). The complex was prepared in 20 mM Tris-glutamate pH 8.0, 50 mM K-glutamate, 10 mM Mg-glutamate, 0.001 mM ZnCl2, and 2mM DTT. The final complex was purified on a Superose 6 Increase 3.2/300 gel filtration column equilibrated in Glutamate buffer. Samples were concentrated to 10-12 mg/mL using an Amicon Ultra 0.5mL centrifugal filter unit (30 KDa MWCO). Before grid freezing, 8 mM of CHAPSO was added to freshly prepared sample to overcome preferred particle orientation. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 平均露光時間: 2.2 sec. / 平均電子線量: 52.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)