+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryoEM structure of the catalytically inactive EndoS from S. pyogenes in complex with the Fc region of immunoglobulin G1 | |||||||||
 マップデータ マップデータ | Main map cryosparc refine | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Endoglycosidase S / EndoS / endo-b-N-acetylglucosaminidase / Fc region / antibody / immunoglobulin G1 / Streptococcus pyogenes / N-glycans / HYDROLASE | |||||||||
| 機能・相同性 | : / Endo-beta-N-acetylglucosaminidase F2, Ig-like domain / Glycosyl hydrolases family 18 (GH18) active site / Glycosyl hydrolases family 18 (GH18) active site signature. / hydrolase activity, hydrolyzing O-glycosyl compounds / Leucine-rich repeat domain superfamily / Glycoside hydrolase superfamily / carbohydrate metabolic process / Endo-beta-N-acetylglucosaminidase F2 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Streptococcus pyogenes (化膿レンサ球菌) / Streptococcus pyogenes (化膿レンサ球菌) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
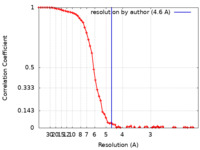
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.6 Å | |||||||||
 データ登録者 データ登録者 | Trastoy B / Cifuente JO / Du JJ / Sundberg EJ / Guerin ME | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Mechanism of antibody-specific deglycosylation and immune evasion by Streptococcal IgG-specific endoglycosidases. 著者: Beatriz Trastoy / Jonathan J Du / Javier O Cifuente / Lorena Rudolph / Mikel García-Alija / Erik H Klontz / Daniel Deredge / Nazneen Sultana / Chau G Huynh / Maria W Flowers / Chao Li / ...著者: Beatriz Trastoy / Jonathan J Du / Javier O Cifuente / Lorena Rudolph / Mikel García-Alija / Erik H Klontz / Daniel Deredge / Nazneen Sultana / Chau G Huynh / Maria W Flowers / Chao Li / Diego E Sastre / Lai-Xi Wang / Francisco Corzana / Alvaro Mallagaray / Eric J Sundberg / Marcelo E Guerin /    要旨: Bacterial pathogens have evolved intricate mechanisms to evade the human immune system, including the production of immunomodulatory enzymes. Streptococcus pyogenes serotypes secrete two multi- ...Bacterial pathogens have evolved intricate mechanisms to evade the human immune system, including the production of immunomodulatory enzymes. Streptococcus pyogenes serotypes secrete two multi-modular endo-β-N-acetylglucosaminidases, EndoS and EndoS2, that specifically deglycosylate the conserved N-glycan at Asn297 on IgG Fc, disabling antibody-mediated effector functions. Amongst thousands of known carbohydrate-active enzymes, EndoS and EndoS2 represent just a handful of enzymes that are specific to the protein portion of the glycoprotein substrate, not just the glycan component. Here, we present the cryoEM structure of EndoS in complex with the IgG1 Fc fragment. In combination with small-angle X-ray scattering, alanine scanning mutagenesis, hydrolytic activity measurements, enzyme kinetics, nuclear magnetic resonance and molecular dynamics analyses, we establish the mechanisms of recognition and specific deglycosylation of IgG antibodies by EndoS and EndoS2. Our results provide a rational basis from which to engineer novel enzymes with antibody and glycan selectivity for clinical and biotechnological applications. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
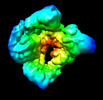
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15205.map.gz emd_15205.map.gz | 10.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15205-v30.xml emd-15205-v30.xml emd-15205.xml emd-15205.xml | 24 KB 24 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15205_fsc.xml emd_15205_fsc.xml | 8.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15205.png emd_15205.png | 92.3 KB | ||
| マスクデータ |  emd_15205_msk_1.map emd_15205_msk_1.map | 22.2 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15205.cif.gz emd-15205.cif.gz | 8 KB | ||
| その他 |  emd_15205_additional_1.map.gz emd_15205_additional_1.map.gz emd_15205_half_map_1.map.gz emd_15205_half_map_1.map.gz emd_15205_half_map_2.map.gz emd_15205_half_map_2.map.gz | 20.9 MB 20.6 MB 20.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15205 http://ftp.pdbj.org/pub/emdb/structures/EMD-15205 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15205 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15205 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15205_validation.pdf.gz emd_15205_validation.pdf.gz | 719 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15205_full_validation.pdf.gz emd_15205_full_validation.pdf.gz | 718.5 KB | 表示 | |
| XML形式データ |  emd_15205_validation.xml.gz emd_15205_validation.xml.gz | 12 KB | 表示 | |
| CIF形式データ |  emd_15205_validation.cif.gz emd_15205_validation.cif.gz | 16.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15205 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15205 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15205 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15205 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8a64MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15205.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15205.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map cryosparc refine | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.052 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15205_msk_1.map emd_15205_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
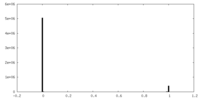
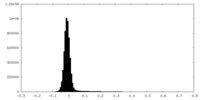
| 密度ヒストグラム |
-追加マップ: Main map cryosparc refine
| ファイル | emd_15205_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map cryosparc refine | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
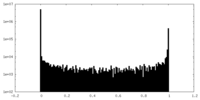
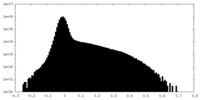
| 密度ヒストグラム |
-ハーフマップ: Half map cryosparc refine
| ファイル | emd_15205_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map cryosparc refine | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
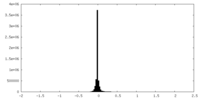
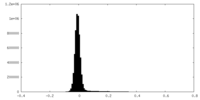
| 密度ヒストグラム |
-ハーフマップ: Half map cryosparc refine
| ファイル | emd_15205_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map cryosparc refine | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
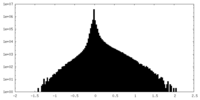
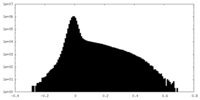
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of the EndoS from S. pyogenes and the Fc region of Immuno...
| 全体 | 名称: Complex of the EndoS from S. pyogenes and the Fc region of Immunoglobulin G |
|---|---|
| 要素 |
|
-超分子 #1: Complex of the EndoS from S. pyogenes and the Fc region of Immuno...
| 超分子 | 名称: Complex of the EndoS from S. pyogenes and the Fc region of Immunoglobulin G タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 150 KDa |
-分子 #1: Endo-beta-N-acetylglucosaminidase F2
| 分子 | 名称: Endo-beta-N-acetylglucosaminidase F2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 108.297039 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EEKTVQVQKG LPSIDSLHYL SENSKKEFKE ELSKAGQESQ KVKEILAKAQ QADKQAQELA KMKIPEKIPM KPLHGPLYGG YFRTWHDKT SDPTEKDKVN SMGELPKEVD LAFIFHDWTK DYSLFWKELA TKHVPKLNKQ GTRVIRTIPW RFLAGGDNSG I AEDTSKYP ...文字列: EEKTVQVQKG LPSIDSLHYL SENSKKEFKE ELSKAGQESQ KVKEILAKAQ QADKQAQELA KMKIPEKIPM KPLHGPLYGG YFRTWHDKT SDPTEKDKVN SMGELPKEVD LAFIFHDWTK DYSLFWKELA TKHVPKLNKQ GTRVIRTIPW RFLAGGDNSG I AEDTSKYP NTPEGNKALA KAIVDEYVYK YNLDGLDVDV AHDSIPKVDK KEDTAGVERS IQVFEEIGKL IGPKGVDKSR LF IMDSTYM ADKNPLIERG APYINLLLVQ VYGSQGEKGG WEPVSNRPEK TMEERWQGYS KYIRPEQYMI GFSFYEENAQ EGN LWYDIN SRKDEDKANG INTDITGTRA ERYARWQPKT GGVKGGIFSY AIDRDGVAHQ PKKYAKQKEF KDATDNIFHS DYSV SKALK TVMLKDKSYD LIDEKDFPDK ALREAVMAQV GTRKGDLERF NGTLRLDNPA IQSLEGLNKF KKLAQLDLIG LSRIT KLDR SVLPANMKPG KDTLETVLET YKKDNKEEPA TIPPVSLKVS GLTGLKELDL SGFDRETLAG LDAATLTSLE KVDISG NKL DLAPGTENRQ IFDTMLSTIS NHVGSNEQTV KFDKQKPTGH YPDTYGKTSL RLPVANEKVD LQSQLLFGTV TNQGTLI NS EADYKAYQNH KIAGRSFVDS NYHYNNFKVS YENYTVKVTD STLGTTTDKT LATDKEETYK VDFFSPADKT KAVHTAKV I VGDEKTMMVN LAEGATVIGG SADPVNARKV FDGQLGSETD NISLGWDSKQ SIIFKLKEDG LIKHWRFFND SARNPETTN KPIQEASLQI FNIKDYNLDN LLENPNKFDD EKYWITVDTY SAQGERATAF SNTLNNITSK YWRVVFDTKG DRYSSPVVPE LQILGYPLP NADTIMKTVT TAKELSQQKD KFSQKMLDEL KIKEMALETS LNSKIFDVTA INANAGVLKD CIEKRQLLKK L UniProtKB: Endo-beta-N-acetylglucosaminidase F2 |
-分子 #2: Immunoglobulin gamma-1 heavy chain
| 分子 | 名称: Immunoglobulin gamma-1 heavy chain / タイプ: protein_or_peptide / ID: 2 詳細: TCPPCPAPELLGGP SVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNS TYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDEL ...詳細: TCPPCPAPELLGGP SVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNS TYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDEL TKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQ QGNVFSCSVMHEALHNHYTQKSLSLSPGK コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 49.386637 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLVQSGGG VVQPGRSLRL SCAASGFTFS RYTIHWVRQA PGKGLEWVAV MSYNGNNKHY ADSVNGRFTI SRNDSKNTLY LNMNSLRPE DTAVYYCARI RDTAMFFAHW GQGTLVTVSS ASTKGPSVFP LAPSSKSTSG GTAALGCLVK DYFPEPVTVS W NSGALTSG ...文字列: QVQLVQSGGG VVQPGRSLRL SCAASGFTFS RYTIHWVRQA PGKGLEWVAV MSYNGNNKHY ADSVNGRFTI SRNDSKNTLY LNMNSLRPE DTAVYYCARI RDTAMFFAHW GQGTLVTVSS ASTKGPSVFP LAPSSKSTSG GTAALGCLVK DYFPEPVTVS W NSGALTSG VHTFPAVLQS SGLYSLSSVV TVPSSSLGTQ TYICNVNHKP SNTKVDKKVE PKSCDKTHTC PPCPAPELLG GP SVFLFPP KPKDTLMISR TPEVTCVVVD VSHEDPEVKF NWYVDGVEVH NAKTKPREEQ YNSTYRVVSV LTVLHQDWLN GKE YKCKVS NKALPAPIEK TISKAKGQPR EPQVYTLPPS RDELTKNQVS LTCLVKGFYP SDIAVEWESN GQPENNYKTT PPVL DSDGS FFLYSKLTVD KSRWQQGNVF SCSVMHEALH NHYTQKSLSL SPGK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: Standard PBS |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK II / 詳細: single blot. |
| 詳細 | Complex stabilized by glutaraldehyde crosslinking prepared by ultracentrifugation in a fixation glycerol gradient (Grafix) and size exclusion chromatography purified at approximately 0.2 mg/mL |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-60 / 撮影したグリッド数: 1 / 実像数: 5546 / 平均電子線量: 59.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.3 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 詳細 | Initial PDB models were rigid-body refined for individual chains. Later, the model was real-space refined removing side chains that couldn't be assigned. | ||||||||
| 精密化 | プロトコル: OTHER | ||||||||
| 得られたモデル |  PDB-8a64: |
 ムービー
ムービー コントローラー
コントローラー



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)