+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of Chaetomium thermophilum INO80 | ||||||||||||||||||
 マップデータ マップデータ | main map | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | chromatin remodeler / INO80 / Actin-related protein / DNA binding protein | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||||||||
| 生物種 |  Thermochaetoides thermophila (菌類) Thermochaetoides thermophila (菌類) | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Kunert F / Metzner FJ / Eustermann S / Jung J / Woike S / Schall K / Kostrewa D / Hopfner KP | ||||||||||||||||||
| 資金援助 | European Union,  ドイツ, 5件 ドイツ, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Structural mechanism of extranucleosomal DNA readout by the INO80 complex. 著者: Franziska Kunert / Felix J Metzner / James Jung / Markus Höpfler / Stephan Woike / Kevin Schall / Dirk Kostrewa / Manuela Moldt / Jia-Xuan Chen / Susanne Bantele / Boris Pfander / Sebastian ...著者: Franziska Kunert / Felix J Metzner / James Jung / Markus Höpfler / Stephan Woike / Kevin Schall / Dirk Kostrewa / Manuela Moldt / Jia-Xuan Chen / Susanne Bantele / Boris Pfander / Sebastian Eustermann / Karl-Peter Hopfner /  要旨: The nucleosomal landscape of chromatin depends on the concerted action of chromatin remodelers. The INO80 remodeler specifically places nucleosomes at the boundary of gene regulatory elements, which ...The nucleosomal landscape of chromatin depends on the concerted action of chromatin remodelers. The INO80 remodeler specifically places nucleosomes at the boundary of gene regulatory elements, which is proposed to be the result of an ATP-dependent nucleosome sliding activity that is regulated by extranucleosomal DNA features. Here, we use cryo-electron microscopy and functional assays to reveal how INO80 binds and is regulated by extranucleosomal DNA. Structures of the regulatory A-module bound to DNA clarify the mechanism of linker DNA binding. The A-module is connected to the motor unit via an HSA/post-HSA lever element to chemomechanically couple the motor and linker DNA sensing. Two notable sites of curved DNA recognition by coordinated action of the four actin/actin-related proteins and the motor suggest how sliding by INO80 can be regulated by extranucleosomal DNA features. Last, the structures clarify the recruitment of YY1/Ies4 subunits and reveal deep architectural similarities between the regulatory modules of INO80 and SWI/SNF complexes. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15165.map.gz emd_15165.map.gz | 5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15165-v30.xml emd-15165-v30.xml emd-15165.xml emd-15165.xml | 22 KB 22 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_15165.png emd_15165.png | 60.3 KB | ||
| Filedesc metadata |  emd-15165.cif.gz emd-15165.cif.gz | 7.1 KB | ||
| その他 |  emd_15165_half_map_1.map.gz emd_15165_half_map_1.map.gz emd_15165_half_map_2.map.gz emd_15165_half_map_2.map.gz | 49.7 MB 49.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15165 http://ftp.pdbj.org/pub/emdb/structures/EMD-15165 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15165 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15165 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15165_validation.pdf.gz emd_15165_validation.pdf.gz | 673.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15165_full_validation.pdf.gz emd_15165_full_validation.pdf.gz | 672.8 KB | 表示 | |
| XML形式データ |  emd_15165_validation.xml.gz emd_15165_validation.xml.gz | 12.1 KB | 表示 | |
| CIF形式データ |  emd_15165_validation.cif.gz emd_15165_validation.cif.gz | 14.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15165 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15165 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15165 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15165 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8a5dMC  8a5aC  8a5oC  8a5pC  8a5qC  8atfC  8av6C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15165.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15165.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.059 Å | ||||||||||||||||||||||||||||||||||||
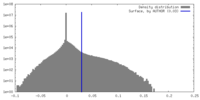
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map A
| ファイル | emd_15165_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
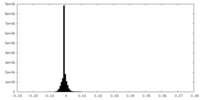
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_15165_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
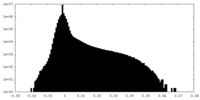
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Chaetomium thermophilum INO80 A-Module (Arp4-Ies4-N-actin-Arp8-In...
| 全体 | 名称: Chaetomium thermophilum INO80 A-Module (Arp4-Ies4-N-actin-Arp8-Ino80HSA) |
|---|---|
| 要素 |
|
-超分子 #1: Chaetomium thermophilum INO80 A-Module (Arp4-Ies4-N-actin-Arp8-In...
| 超分子 | 名称: Chaetomium thermophilum INO80 A-Module (Arp4-Ies4-N-actin-Arp8-Ino80HSA) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila (菌類) Thermochaetoides thermophila (菌類) |
-分子 #1: Ino80 ATPase
| 分子 | 名称: Ino80 ATPase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 Thermochaetoides thermophila (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 37.445441 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: RKRRREKSMA ATLEQQAAAL QKASQAEDEA ERLKYIREAE RANKKVQQTK YILQKGIKGP ARNLPPIEPN LEGGTMATFS AENMEPGKV KGKGRGGRLK KSKEQKQAEK EQAELAQAAI DAGEEPPSKE ETKIRIKLSK AKASAKEEAE KDKENKEPKQ P KEPEKPVE ...文字列: RKRRREKSMA ATLEQQAAAL QKASQAEDEA ERLKYIREAE RANKKVQQTK YILQKGIKGP ARNLPPIEPN LEGGTMATFS AENMEPGKV KGKGRGGRLK KSKEQKQAEK EQAELAQAAI DAGEEPPSKE ETKIRIKLSK AKASAKEEAE KDKENKEPKQ P KEPEKPVE EPKDPLELKF QSKGYNQIYD QIWRDLARKD VSKVFRLATD SYATKASNLK KTAILASKEA KRWQLRTNKG TK DLQARAK RVMRDMMGFW KRNEREERDL RKAAERLELE NARKEEADRE AARQRRKLNF LISQTELYSD YKDDDDKGTD YKD DDDK |
-分子 #2: Actin-related protein 8
| 分子 | 名称: Actin-related protein 8 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 Thermochaetoides thermophila (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 84.139266 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MVGKVSERVL AREGLERTDN GMRQTSWPEV QPINQKNYYT DYMKRDDQIL SLRLQNEANR ERLVKTAQDR DRALNTNTNG EVPLPLDEL QEDAPPAPSA AMDPSKIIVI HPGSQNLRIG FASDALPKTI PMVLGTQYSQ TESEMHEALP RRQFEGRTME Q QYGEEWVK ...文字列: MVGKVSERVL AREGLERTDN GMRQTSWPEV QPINQKNYYT DYMKRDDQIL SLRLQNEANR ERLVKTAQDR DRALNTNTNG EVPLPLDEL QEDAPPAPSA AMDPSKIIVI HPGSQNLRIG FASDALPKTI PMVLGTQYSQ TESEMHEALP RRQFEGRTME Q QYGEEWVK KYQKMCSDLK VTMRANKLKV LPNSKDLVVN FNRRTEPEII SQHNDPLQVE WTNVNKAPED GTTQKVFIGQ QA LRIAEDS SPKYKLWWPI QHGWLNEDDY PTRAHLFDDL EMLLDRALRR ELGLTKKADW KQYSCVIVIP DLYDKRYVEL LLH LCIEFF DLSRVAFIQE SMAATFGAGY TQACVVDVGA QKTSISCVED GLVIEDSRVN LKYGGYDVTE TFIKMMLYDN FPYQ DINLR RRHDFLLAEE LKMKYCTLSQ ANISVQAFDF HLRAPNQPTR KYQFKFYDEV ILAPMGFYDP SIFDNSTKLR GRRKL IDRS YNAYDVDMPD DPTSSAQLAI LAMVQPSLAA PTATASFNGD PTATPSKERT QPFNFLSRPD ATGTPGTSKA PSPAPD GAS TPVPGPYIFG ASSREANGGS PAPSGRNGGT PAPANGTTVL PATSFDNPTS QQPQQRSAKE IAAERDAVLP IAPLDVA IL TSIQHAAKGD EKKVRELVGS IMVVGGGAKI PHFAPFLEEK LKIRRPDLAD RILVSRSARE MDEQVVVWKG ASVFAKLS T NDSWVTNYEY KMLGSRCIYM KLLWHY UniProtKB: Putative actin-related protein |
-分子 #3: Actin
| 分子 | 名称: Actin / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 Thermochaetoides thermophila (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 41.735512 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MEEEVAALVI DNGSGMCKAG FAGDDAPRAV FPSIVGRPRH HGIMIGMGQK DSYVGDEAQS KRGILTLRYP IEHGVVTNWD DMEKIWHHT FYNELRVAPE EHPVLLTEAP INPKSNREKM TQIVFETFNA PAFYVSIQAV LSLYASGRTT GIVLDSGDGV T HVVPIYEG ...文字列: MEEEVAALVI DNGSGMCKAG FAGDDAPRAV FPSIVGRPRH HGIMIGMGQK DSYVGDEAQS KRGILTLRYP IEHGVVTNWD DMEKIWHHT FYNELRVAPE EHPVLLTEAP INPKSNREKM TQIVFETFNA PAFYVSIQAV LSLYASGRTT GIVLDSGDGV T HVVPIYEG FSLPHAIARL DMAGRDLTDY LMKILAERGY TFSTTAEREI VRDIKEKLCY VALDFEQEIQ TAAQSSHLEK SY ELPDGQV ITIGNERFRA PEALFQPSVL GLESGGIHVT TFNSIMKCDV DVRKDLYGNI VMSGGTTMYP GLSDRMQKEI TAL APSSMK VKIIAPPERK YSVWIGGSIL ASLSTFQQMW ISKQEYDESG PSIVHRKCF UniProtKB: Actin |
-分子 #4: Actin related protein 4 (Arp4)
| 分子 | 名称: Actin related protein 4 (Arp4) / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 Thermochaetoides thermophila (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 51.761199 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSQQPLSSSV QPADIYGGDE VSALVLDPGY CNTRAGFAGE EMPKQVIPSF YGHVDGRDLF GDEVIVPRAG FEVRNYMNRD SMVEDWDAA TRVWEYLLVQ RLQPPRPTPA SKNGLNVSDD GDVQMEDSAP NEDALDALEK PLTENPLLMT EAPWNTPKAR E KAIEVVME ...文字列: MSQQPLSSSV QPADIYGGDE VSALVLDPGY CNTRAGFAGE EMPKQVIPSF YGHVDGRDLF GDEVIVPRAG FEVRNYMNRD SMVEDWDAA TRVWEYLLVQ RLQPPRPTPA SKNGLNVSDD GDVQMEDSAP NEDALDALEK PLTENPLLMT EAPWNTPKAR E KAIEVVME NWGTPAFWLS RTPVLAAFAA GKATALVIDV GGANTSVTAI HDGMVLKRSI QRSPAAGVWL SGQIRSMWKS QD PPVNVVP TFMVENKKPV EAGAPPDCRL RNFGFPIHDS FRAFEEERVL TEFKESVVEV WRGPGKYLNP GNEDFAKTQP GRV FEFPDG SNQMWREQRY RVAEGMWDET AAYPSLNPDE AAITKAQTIP ALIKAALDGV DVDLRPNLLG NVVVTGSTSL LNGF NDRLN HELTNMYPGL KIKLHAAGLT SERRFGAWIG GSILASLGTF HQMWISRKEY EENGAGIVEK RCK UniProtKB: Actin-related protein 4 |
-分子 #5: Ies4
| 分子 | 名称: Ies4 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 Thermochaetoides thermophila (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 29.015457 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAPSSKNATP SNKDRRKSGI AAAGNGNAAA SSKIVTLKVT PERLRAIFDP DILMEDAPAS KASPAAANSN TENATESNPN SPSASTPAG QSVMGPPAEG PKKKGVKRGA AALGPNGEPK PRGKPGPKKK QRLEDGTIVE EPSRKLGPKA NQGAINAGLR A LDRSGKPC ...文字列: MAPSSKNATP SNKDRRKSGI AAAGNGNAAA SSKIVTLKVT PERLRAIFDP DILMEDAPAS KASPAAANSN TENATESNPN SPSASTPAG QSVMGPPAEG PKKKGVKRGA AALGPNGEPK PRGKPGPKKK QRLEDGTIVE EPSRKLGPKA NQGAINAGLR A LDRSGKPC RKWTRGTFQM KSFTGVVWEI PRWTAPPRPK PETAAPDDAA ATVAVNGNGA TPASTAADNN SNKENNNPSI AA AKVEDAS SQTAVKSEMS TGSGDIEMQS VAPSIAPSPA PVPIAVA UniProtKB: INO80 complex subunit 4 |
-分子 #6: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 6 / コピー数: 3 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #7: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 7 / コピー数: 3 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 44.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.9 µm / 最小 デフォーカス(公称値): 1.1 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 343000 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)