+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Nup93 in complex with 5-Nup93 inhibitory NB and 15-Nup93 tracking NB | |||||||||
 マップデータ マップデータ | Nup93 in complex with inhibitory Nanobody | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NUP93 / Nuclearporin / inhibitory NB / tracking NB / NUCLEAR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nuclear pore complex assembly / structural constituent of nuclear pore / poly(A)+ mRNA export from nucleus / nuclear pore / nuclear periphery / protein import into nucleus / nuclear membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 | Xenopus laevis, Vicugna pacos /  | |||||||||
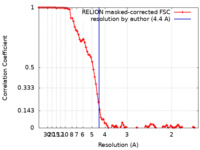
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | |||||||||
 データ登録者 データ登録者 | Fu Z / Guttler T / Colom MS | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2024 ジャーナル: EMBO J / 年: 2024タイトル: A checkpoint function for Nup98 in nuclear pore formation suggested by novel inhibitory nanobodies. 著者: Mireia Solà Colom / Zhenglin Fu / Philip Gunkel / Thomas Güttler / Sergei Trakhanov / Vasundara Srinivasan / Kathrin Gregor / Tino Pleiner / Dirk Görlich /   要旨: Nuclear pore complex (NPC) biogenesis is a still enigmatic example of protein self-assembly. We now introduce several cross-reacting anti-Nup nanobodies for imaging intact nuclear pore complexes from ...Nuclear pore complex (NPC) biogenesis is a still enigmatic example of protein self-assembly. We now introduce several cross-reacting anti-Nup nanobodies for imaging intact nuclear pore complexes from frog to human. We also report a simplified assay that directly tracks postmitotic NPC assembly with added fluorophore-labeled anti-Nup nanobodies. During interphase, NPCs are inserted into a pre-existing nuclear envelope. Monitoring this process is challenging because newly assembled NPCs are indistinguishable from pre-existing ones. We overcame this problem by inserting Xenopus-derived NPCs into human nuclear envelopes and using frog-specific anti-Nup nanobodies for detection. We further asked whether anti-Nup nanobodies could serve as NPC assembly inhibitors. Using a selection strategy against conserved epitopes, we obtained anti-Nup93, Nup98, and Nup155 nanobodies that block Nup-Nup interfaces and arrest NPC assembly. We solved structures of nanobody-target complexes and identified roles for the Nup93 α-solenoid domain in recruiting Nup358 and the Nup214·88·62 complex, as well as for Nup155 and the Nup98 autoproteolytic domain in NPC scaffold assembly. The latter suggests a checkpoint linking pore formation to the assembly of the Nup98-dominated permeability barrier. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14849.map.gz emd_14849.map.gz | 4.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14849-v30.xml emd-14849-v30.xml emd-14849.xml emd-14849.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14849_fsc.xml emd_14849_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14849.png emd_14849.png | 58.3 KB | ||
| Filedesc metadata |  emd-14849.cif.gz emd-14849.cif.gz | 6.7 KB | ||
| その他 |  emd_14849_half_map_1.map.gz emd_14849_half_map_1.map.gz emd_14849_half_map_2.map.gz emd_14849_half_map_2.map.gz | 49.7 MB 49.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14849 http://ftp.pdbj.org/pub/emdb/structures/EMD-14849 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14849 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14849 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14849.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14849.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nup93 in complex with inhibitory Nanobody | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.834 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map of Nup93 in complex with inhibitory Nanobody
| ファイル | emd_14849_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map of Nup93 in complex with inhibitory Nanobody | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map of Nup93 in complex with inhibitory Nanobody
| ファイル | emd_14849_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map of Nup93 in complex with inhibitory Nanobody | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nup93 in complex with 5-Nup93 inhibitory NB and 15-Nup93 tracking NB
| 全体 | 名称: Nup93 in complex with 5-Nup93 inhibitory NB and 15-Nup93 tracking NB |
|---|---|
| 要素 |
|
-超分子 #1: Nup93 in complex with 5-Nup93 inhibitory NB and 15-Nup93 tracking NB
| 超分子 | 名称: Nup93 in complex with 5-Nup93 inhibitory NB and 15-Nup93 tracking NB タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: Xenopus laevis, Vicugna pacos |
| 分子量 | 理論値: 100 KDa |
-分子 #1: Nuclear pore complex protein Nup93
| 分子 | 名称: Nuclear pore complex protein Nup93 / タイプ: protein_or_peptide / ID: 1 / 詳細: Xenopus laevis Nup93(residues 168-820) / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 74.782289 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SESGAPGRSS LDNVEMAYAR QMYMYNEKVV SGHLQPSLVD LCTEAAERLD DKNVSDLWVM VKQMTDVPLI PASDTLKSRC SGQMQMAFV RQALNYLEQS YKNYTLISVF ANLQQAQLGG VPGTYNLVRS FLNIRLPTPI PGLQDGEIEG YPVWALIYYC M RCGDLMAA ...文字列: SESGAPGRSS LDNVEMAYAR QMYMYNEKVV SGHLQPSLVD LCTEAAERLD DKNVSDLWVM VKQMTDVPLI PASDTLKSRC SGQMQMAFV RQALNYLEQS YKNYTLISVF ANLQQAQLGG VPGTYNLVRS FLNIRLPTPI PGLQDGEIEG YPVWALIYYC M RCGDLMAA QQVVNRAQHQ LGDFKNCFQE YIHNKDRRLS PTTENKLRLH YRRAVRASTD PYKRAVYCII GRCDVSDNHS EV ADKTEDY LWLKLSQVCF EDEANSSPQD RLTLPQFQKQ LFEDYGESHF AVNQQPYLYF QVLFLTAQFE AAIAFLFRLE RTR CHAVHV ALALFELKLL LKSTGQSAQL LSQEPGEPQG VRRLNFIRLL MLYTRKFEPT DPREALQYFY FLRNEKDNQG ESMF LRCVS ELVIESREFD MLLGKLEKDG SRKPGAIDKF TRDTKTIINK VASVAENKGL FEEAAKLYDL AKNPDKVLEL TNKLL SPVV SQISAPQSNR ERLKNMALAI AERYKSQGVS AEKSINSTFY LLLDLITFFD EYHAGHIDLS FDVIERLKLV PLSQDS VEE RVAAFRNFSD EIRHNLSEIL LATMNILFTQ YKRLKGSGPT TLGRPQRVQE DKDSVLRSQA RALITFAGMI PYRMSGD TN ARLVQMEVLM N UniProtKB: Nuclear pore complex protein Nup93 |
-分子 #2: xhNup93-Nb4i
| 分子 | 名称: xhNup93-Nb4i / タイプ: protein_or_peptide / ID: 2 / 詳細: 5-Nup93 inhibitory NB / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.811384 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GQVQLVESGG GVVQTGDSLM LSCTVSGHTI DNWAMGWFRQ TPRKQREFVA AIDKRGTGAI YGNSVKGRFT VSRDNAKNMV YLRMNSLKP EDTAVYFCAV DQLNAGLGDV SYDYDYWGQG TQVTVSS |
-分子 #3: xNup93-Nb2t
| 分子 | 名称: xNup93-Nb2t / タイプ: protein_or_peptide / ID: 3 / 詳細: 15-Nup93 tracking NB / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 12.921641 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GDVQLVESGG GLVQPGGSLR LSCVASGFTF SRVGMGWVRQ APGKGLEWVS DINASGGTGY ADSVKGRFAI SRDNAKNTLY LQMNRLKPE DTAVYYCAKM MDTAMIEAGI IKPAGQGTQV TVSS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.04 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Sample volume: 2.0 microliters blotting time: 5 s blot force setting: 6. | ||||||||||||
| 詳細 | The complex was further purified by size exclusion chromatography using a HiLoad 26/60 Superdex 200 column. The purified complex was applied to a glow-discharged grid after being diluted to 1 mg/ml. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 12320 / 平均露光時間: 2.0 sec. / 平均電子線量: 54.0 e/Å2 / 詳細: Counting mode 5 images per hole( beam-image shift) |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)