+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1429 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A lever-arm rotation drives motility of the minus-end-directed kinesin Ncd. | |||||||||
 マップデータ マップデータ | ncd in its ADP-Pi state on helical 15 filament microtubules | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 15.0 Å | |||||||||
 データ登録者 データ登録者 | Endres NF / Yoshioka C / Milligan RA / Vale RD | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2006 ジャーナル: Nature / 年: 2006タイトル: A lever-arm rotation drives motility of the minus-end-directed kinesin Ncd. 著者: Nicholas F Endres / Craig Yoshioka / Ronald A Milligan / Ronald D Vale /  要旨: Kinesins are microtubule-based motor proteins that power intracellular transport. Most kinesin motors, exemplified by Kinesin-1, move towards the microtubule plus end, and the structural changes that ...Kinesins are microtubule-based motor proteins that power intracellular transport. Most kinesin motors, exemplified by Kinesin-1, move towards the microtubule plus end, and the structural changes that govern this directional preference have been described. By contrast, the nature and timing of the structural changes underlying the minus-end-directed motility of Kinesin-14 motors (such as Drosophila Ncd) are less well understood. Using cryo-electron microscopy, here we demonstrate that a coiled-coil mechanical element of microtubule-bound Ncd rotates approximately 70 degrees towards the minus end upon ATP binding. Extending or shortening this coiled coil increases or decreases velocity, respectively, without affecting ATPase activity. An unusual Ncd mutant that lacks directional preference shows unstable nucleotide-dependent conformations of its coiled coil, underscoring the role of this mechanical element in motility. These results show that the force-producing conformational change in Ncd occurs on ATP binding, as in other kinesins, but involves the swing of a lever-arm mechanical element similar to that described for myosins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1429.map.gz emd_1429.map.gz | 485.9 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1429-v30.xml emd-1429-v30.xml emd-1429.xml emd-1429.xml | 8.9 KB 8.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1429.gif 1429.gif | 70.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1429 http://ftp.pdbj.org/pub/emdb/structures/EMD-1429 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1429 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1429 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1429_validation.pdf.gz emd_1429_validation.pdf.gz | 269.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1429_full_validation.pdf.gz emd_1429_full_validation.pdf.gz | 268.8 KB | 表示 | |
| XML形式データ |  emd_1429_validation.xml.gz emd_1429_validation.xml.gz | 4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1429 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1429 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1429 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1429 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1429.map.gz / 形式: CCP4 / 大きさ: 2.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1429.map.gz / 形式: CCP4 / 大きさ: 2.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ncd in its ADP-Pi state on helical 15 filament microtubules | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
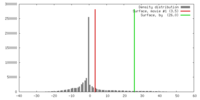
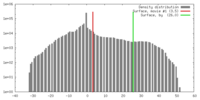
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : non-claret disjunction adp-pi state
| 全体 | 名称: non-claret disjunction adp-pi state |
|---|---|
| 要素 |
|
-超分子 #1000: non-claret disjunction adp-pi state
| 超分子 | 名称: non-claret disjunction adp-pi state / タイプ: sample / ID: 1000 集合状態: Motors bound to 15 protofilament helical microtubules Number unique components: 2 |
|---|
-分子 #1: tubulin
| 分子 | 名称: tubulin / タイプ: protein_or_peptide / ID: 1 / 集合状態: helical 15 protofilaments / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-分子 #2: ncd
| 分子 | 名称: ncd / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 10 mg/mL |
|---|---|
| 緩衝液 | 詳細: 25 mM MOPS (pH 7.25), 100 mM NaCl, 2 mM MgCl2, 1 mM EGTA and 1 mM dithiothreitol, 5mg/ml tubulin, 5mM ADP-AlF4 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 4 K / 装置: OTHER / 詳細: Vitrification instrument: Vitrobot / 手法: 3 sec blot |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 平均電子線量: 10 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 倍率(公称値): 39000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 15.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: phoelix |
|---|
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)