+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1424 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The structure of the prokaryotic cyclic nucleotide-modulated potassium channel MloK1 at 16 A resolution. | |||||||||
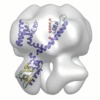
 マップデータ マップデータ | This is the 3D map of MloK1 in 17-A resolution determined by NS-TEM. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Mesorhizobium loti (根粒菌) / synthetic construct (人工物) Mesorhizobium loti (根粒菌) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 16.3 Å | |||||||||
 データ登録者 データ登録者 | Chiu P-L / Pagel M / Evans J / Chou H-T / Gipson B | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2007 ジャーナル: Structure / 年: 2007タイトル: The structure of the prokaryotic cyclic nucleotide-modulated potassium channel MloK1 at 16 A resolution. 著者: Po-Lin Chiu / Matthew D Pagel / James Evans / Hui-Ting Chou / Xiangyan Zeng / Bryant Gipson / Henning Stahlberg / Crina M Nimigean /  要旨: The gating ring of cyclic nucleotide-modulated channels is proposed to be either a two-fold symmetric dimer of dimers or a four-fold symmetric tetramer based on high-resolution structure data of ...The gating ring of cyclic nucleotide-modulated channels is proposed to be either a two-fold symmetric dimer of dimers or a four-fold symmetric tetramer based on high-resolution structure data of soluble cyclic nucleotide-binding domains and functional data on intact channels. We addressed this controversy by obtaining structural data on an intact, full-length, cyclic nucleotide-modulated potassium channel, MloK1, from Mesorhizobium loti, which also features a putative voltage-sensor. We present here the 3D single-particle structure by transmission electron microscopy and the projection map of membrane-reconstituted 2D crystals of MloK1 in the presence of cAMP. Our data show a four-fold symmetric arrangement of the CNBDs, separated by discrete gaps. A homology model for full-length MloK1 suggests a vertical orientation for the CNBDs. The 2D crystal packing in the membrane-embedded state is compatible with the S1-S4 domains in the vertical "up" state. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1424.map.gz emd_1424.map.gz | 1.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1424-v30.xml emd-1424-v30.xml emd-1424.xml emd-1424.xml | 10.4 KB 10.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1424.gif 1424.gif | 67.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1424 http://ftp.pdbj.org/pub/emdb/structures/EMD-1424 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1424 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1424 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1424_validation.pdf.gz emd_1424_validation.pdf.gz | 208.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1424_full_validation.pdf.gz emd_1424_full_validation.pdf.gz | 207.7 KB | 表示 | |
| XML形式データ |  emd_1424_validation.xml.gz emd_1424_validation.xml.gz | 4.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1424 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1424 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1424 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1424 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1424.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1424.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is the 3D map of MloK1 in 17-A resolution determined by NS-TEM. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Full-length prokaryotic potassium channel MloK1
| 全体 | 名称: Full-length prokaryotic potassium channel MloK1 |
|---|---|
| 要素 |
|
-超分子 #1000: Full-length prokaryotic potassium channel MloK1
| 超分子 | 名称: Full-length prokaryotic potassium channel MloK1 / タイプ: sample / ID: 1000 / 集合状態: One tetramer with ligand binding / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 155.2 KDa / 理論値: 152.5 KDa / 手法: Calculation by the protein sequence |
-分子 #1: Prokaryotic potassium channel
| 分子 | 名称: Prokaryotic potassium channel / タイプ: protein_or_peptide / ID: 1 / Name.synonym: MloK1 / 詳細: Each subunit of the MloK1 / コピー数: 1 / 集合状態: Tetramer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Mesorhizobium loti (根粒菌) / 株: MloK1 / 別称: Soil bacteria / 組織: Prokaryote / Organelle: Cell membrane / 細胞中の位置: Cell membrane Mesorhizobium loti (根粒菌) / 株: MloK1 / 別称: Soil bacteria / 組織: Prokaryote / Organelle: Cell membrane / 細胞中の位置: Cell membrane |
| 分子量 | 実験値: 37.74 KDa / 理論値: 38.42 KDa |
| 組換発現 | 生物種:  |
-分子 #2: Cyclic nucleotide
| 分子 | 名称: Cyclic nucleotide / タイプ: ligand / ID: 2 / Name.synonym: cAMP 詳細: The ligand for cyclic nucleotide-modulated channel, MloK1 組換発現: No |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 実験値: 369.2 Da / 理論値: 369.2 Da |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 詳細: 100 mM KCl, 20 mM Tris-Cl, 5 mM DM, 200 uM cAMP |
| 染色 | タイプ: NEGATIVE 詳細: Grids with absorbed proteins deeply stained with 2% uranyl formate |
| グリッド | 詳細: 400 mesh copper grid |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2100F |
|---|---|
| 温度 | 平均: 298.15 K |
| 日付 | 2005年10月27日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F415 (4k x 4k) デジタル化 - サンプリング間隔: 10 µm / 実像数: 34 / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 60000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Side-entry room temperature holder / 試料ホルダーモデル: HOME BUILD |
- 画像解析
画像解析
| CTF補正 | 詳細: CTF correction of each particle |
|---|---|
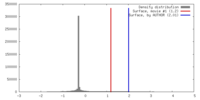
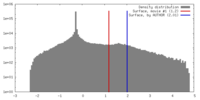
| 最終 再構成 | 想定した対称性 - 点群: C4 (4回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.3 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER, EMAN / 使用した粒子像数: 14921 |
| 最終 角度割当 | 詳細: Refinement with MRA method built in EMAN package |
| 最終 2次元分類 | クラス数: 190 |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)