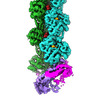
登録情報 データベース : EMDB / ID : EMD-13343タイトル Structure of capping protein bound to the barbed end of a cytoplasmic actin filament Locally filtered map with global B-factor 複合体 : Complex between capping protein and the barbed end of cytoplasmic actin filamentsタンパク質・ペプチド : Isoform 2 of F-actin-capping protein subunit betaタンパク質・ペプチド : F-actin-capping protein subunit alpha-1タンパク質・ペプチド : Actin, cytoplasmic 1タンパク質・ペプチド : Phalloidinリガンド : ADENOSINE-5'-DIPHOSPHATEリガンド : MAGNESIUM IONリガンド : PHOSPHATE ION機能・相同性 分子機能 ドメイン・相同性 構成要素
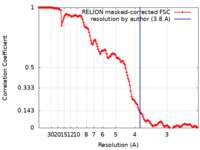
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Mus musculus (ハツカネズミ) / Bovine (ウシ) / synthetic construct (人工物) 手法 / / 解像度 : 3.8 Å Funk J / Merino F / Schacks M / Rottner K / Raunser S / Bieling P 資金援助 Organization Grant number 国 Max Planck Society
ジャーナル : Nat Commun / 年 : 2021タイトル : A barbed end interference mechanism reveals how capping protein promotes nucleation in branched actin networks.著者 : Johanna Funk / Felipe Merino / Matthias Schaks / Klemens Rottner / Stefan Raunser / Peter Bieling / 要旨 : Heterodimeric capping protein (CP/CapZ) is an essential factor for the assembly of branched actin networks, which push against cellular membranes to drive a large variety of cellular processes. Aside ... Heterodimeric capping protein (CP/CapZ) is an essential factor for the assembly of branched actin networks, which push against cellular membranes to drive a large variety of cellular processes. Aside from terminating filament growth, CP potentiates the nucleation of actin filaments by the Arp2/3 complex in branched actin networks through an unclear mechanism. Here, we combine structural biology with in vitro reconstitution to demonstrate that CP not only terminates filament elongation, but indirectly stimulates the activity of Arp2/3 activating nucleation promoting factors (NPFs) by preventing their association to filament barbed ends. Key to this function is one of CP's C-terminal "tentacle" extensions, which sterically masks the main interaction site of the terminal actin protomer. Deletion of the β tentacle only modestly impairs capping. However, in the context of a growing branched actin network, its removal potently inhibits nucleation promoting factors by tethering them to capped filament ends. End tethering of NPFs prevents their loading with actin monomers required for activation of the Arp2/3 complex and thus strongly inhibits branched network assembly both in cells and reconstituted motility assays. Our results mechanistically explain how CP couples two opposed processes-capping and nucleation-in branched actin network assembly. 履歴 登録 2021年8月9日 - ヘッダ(付随情報) 公開 2021年9月1日 - マップ公開 2021年9月1日 - 更新 2021年10月6日 - 現状 2021年10月6日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報

 データ登録者
データ登録者 ドイツ, 1件
ドイツ, 1件  引用
引用 ジャーナル: Nat Commun / 年: 2021
ジャーナル: Nat Commun / 年: 2021
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_13343.map.gz
emd_13343.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-13343-v30.xml
emd-13343-v30.xml emd-13343.xml
emd-13343.xml EMDBヘッダ
EMDBヘッダ emd_13343_fsc.xml
emd_13343_fsc.xml FSCデータファイル
FSCデータファイル emd_13343.png
emd_13343.png emd_13343_msk_1.map
emd_13343_msk_1.map マスクマップ
マスクマップ emd_13343_additional_1.map.gz
emd_13343_additional_1.map.gz emd_13343_additional_2.map.gz
emd_13343_additional_2.map.gz emd_13343_half_map_1.map.gz
emd_13343_half_map_1.map.gz emd_13343_half_map_2.map.gz
emd_13343_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-13343
http://ftp.pdbj.org/pub/emdb/structures/EMD-13343 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13343
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13343 emd_13343_validation.pdf.gz
emd_13343_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_13343_full_validation.pdf.gz
emd_13343_full_validation.pdf.gz emd_13343_validation.xml.gz
emd_13343_validation.xml.gz emd_13343_validation.cif.gz
emd_13343_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13343
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13343 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13343
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13343 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_13343.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_13343.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_13343_msk_1.map
emd_13343_msk_1.map 試料の構成要素
試料の構成要素






 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)