+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | RuvAB branch migration motor complexed to the Holliday junction - RuvB AAA+ state s0-A [t2 dataset] | |||||||||
 マップデータ マップデータ | RuvB AAA state s0-A [t2 dataset] | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA recombination / DNA repair / branch migration / Holliday junction / helicase / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Holliday junction resolvase complex / four-way junction helicase activity / four-way junction DNA binding / DNA recombination / DNA helicase / DNA repair / ATP hydrolysis activity / ATP binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) / synthetic construct (人工物) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) / synthetic construct (人工物) | |||||||||
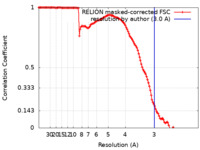
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Wald J / Marlovits TC | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: Mechanism of AAA+ ATPase-mediated RuvAB-Holliday junction branch migration. 著者: Jiri Wald / Dirk Fahrenkamp / Nikolaus Goessweiner-Mohr / Wolfgang Lugmayr / Luciano Ciccarelli / Oliver Vesper / Thomas C Marlovits /    要旨: The Holliday junction is a key intermediate formed during DNA recombination across all kingdoms of life. In bacteria, the Holliday junction is processed by two homo-hexameric AAA+ ATPase RuvB motors, ...The Holliday junction is a key intermediate formed during DNA recombination across all kingdoms of life. In bacteria, the Holliday junction is processed by two homo-hexameric AAA+ ATPase RuvB motors, which assemble together with the RuvA-Holliday junction complex to energize the strand-exchange reaction. Despite its importance for chromosome maintenance, the structure and mechanism by which this complex facilitates branch migration are unknown. Here, using time-resolved cryo-electron microscopy, we obtained structures of the ATP-hydrolysing RuvAB complex in seven distinct conformational states, captured during assembly and processing of a Holliday junction. Five structures together resolve the complete nucleotide cycle and reveal the spatiotemporal relationship between ATP hydrolysis, nucleotide exchange and context-specific conformational changes in RuvB. Coordinated motions in a converter formed by DNA-disengaged RuvB subunits stimulate hydrolysis and nucleotide exchange. Immobilization of the converter enables RuvB to convert the ATP-contained energy into a lever motion, which generates the pulling force driving the branch migration. We show that RuvB motors rotate together with the DNA substrate, which, together with a progressing nucleotide cycle, forms the mechanistic basis for DNA recombination by continuous branch migration. Together, our data decipher the molecular principles of homologous recombination by the RuvAB complex, elucidate discrete and sequential transition-state intermediates for chemo-mechanical coupling of hexameric AAA+ motors and provide a blueprint for the design of state-specific compounds targeting AAA+ motors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13300.map.gz emd_13300.map.gz | 9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13300-v30.xml emd-13300-v30.xml emd-13300.xml emd-13300.xml | 14.6 KB 14.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13300_fsc.xml emd_13300_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13300.png emd_13300.png | 95.5 KB | ||
| Filedesc metadata |  emd-13300.cif.gz emd-13300.cif.gz | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13300 http://ftp.pdbj.org/pub/emdb/structures/EMD-13300 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13300 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13300 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13300_validation.pdf.gz emd_13300_validation.pdf.gz | 364.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13300_full_validation.pdf.gz emd_13300_full_validation.pdf.gz | 364.4 KB | 表示 | |
| XML形式データ |  emd_13300_validation.xml.gz emd_13300_validation.xml.gz | 12.8 KB | 表示 | |
| CIF形式データ |  emd_13300_validation.cif.gz emd_13300_validation.cif.gz | 17.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13300 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13300 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13300 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13300 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7pbrMC  7pblC  7pbmC  7pbnC  7pboC  7pbpC  7pbqC  7pbsC  7pbtC  7pbuC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13300.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13300.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RuvB AAA state s0-A [t2 dataset] | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : RuvAB branch migration motor complexed to the Holliday junction -...
| 全体 | 名称: RuvAB branch migration motor complexed to the Holliday junction - RuvB AAA+ state s0-A [t2 dataset] |
|---|---|
| 要素 |
|
-超分子 #1: RuvAB branch migration motor complexed to the Holliday junction -...
| 超分子 | 名称: RuvAB branch migration motor complexed to the Holliday junction - RuvB AAA+ state s0-A [t2 dataset] タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 / 詳細: RuvB helicase |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 220 KDa |
-分子 #1: Holliday junction ATP-dependent DNA helicase RuvB
| 分子 | 名称: Holliday junction ATP-dependent DNA helicase RuvB / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 35.447508 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TLRPQYFKEY IGQDKVKDQL KIFIEAAKLR DEALDHTLLF GPPGLGKTTM AFVIANEMGV NLKQTSGPAI EKAGDLVAIL NDLEPGDIL FIDEIHRMPM AVEEVLYSAM EDYYIDIMIG AGETSRSVHL DLPPFTLVGA TTRAGMLSNP LRARFGINGH M EYYELPDL ...文字列: TLRPQYFKEY IGQDKVKDQL KIFIEAAKLR DEALDHTLLF GPPGLGKTTM AFVIANEMGV NLKQTSGPAI EKAGDLVAIL NDLEPGDIL FIDEIHRMPM AVEEVLYSAM EDYYIDIMIG AGETSRSVHL DLPPFTLVGA TTRAGMLSNP LRARFGINGH M EYYELPDL TEIVERTSEI FEMTITPEAA LELARRSRGT PRIANRLLKR VRDYAQIMGD GVIDDKIADQ ALTMLDVDHE GL DYVDQKI LRTMIEMYGG GPVGLGTLSV NIAEERETVE DMYEPYLIQK GFIMRTRTGR VATAKAYEHM GYDYTRDN UniProtKB: Holliday junction branch migration complex subunit RuvB |
-分子 #2: random DNA
| 分子 | 名称: random DNA / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 4.643037 KDa |
| 配列 | 文字列: (DG)(DA)(DA)(DC)(DC)(DT)(DT)(DC)(DG)(DA) (DG)(DG)(DA)(DA)(DG) |
-分子 #3: random DNA
| 分子 | 名称: random DNA / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 4.535946 KDa |
| 配列 | 文字列: (DC)(DT)(DT)(DC)(DC)(DT)(DC)(DG)(DA)(DA) (DG)(DG)(DT)(DT)(DC) |
-分子 #4: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 4 / コピー数: 4 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 2 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 0.15 |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | in-vitro reconstituted freshly before vitrification |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 実像数: 30083 / 平均露光時間: 5.0 sec. / 平均電子線量: 30.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-7pbr: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)