+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13157 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM density of caspase-3 cleaved rXKR9 in complex with a sybody at 4.3A | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | small membrane protein / in complex with sybody / apoptotic lipid scrambling / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 | XK-related protein / : / XK-related protein / membrane / plasma membrane / XK-related protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
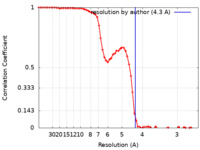
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.3 Å | |||||||||
 データ登録者 データ登録者 | Straub MS / Sawicka M | |||||||||
| 資金援助 |  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: Cryo-EM structures of the caspase-activated protein XKR9 involved in apoptotic lipid scrambling. 著者: Monique S Straub / Carolina Alvadia / Marta Sawicka / Raimund Dutzler /  要旨: The exposure of the negatively charged lipid phosphatidylserine on the cell surface, catalyzed by lipid scramblases, is an important signal for the clearance of apoptotic cells by macrophages. The ...The exposure of the negatively charged lipid phosphatidylserine on the cell surface, catalyzed by lipid scramblases, is an important signal for the clearance of apoptotic cells by macrophages. The protein XKR9 is a member of a conserved family that has been associated with apoptotic lipid scrambling. Here, we describe structures of full-length and caspase-treated XKR9 from in complex with a synthetic nanobody determined by cryo-electron microscopy. The 43 kDa monomeric membrane protein can be divided into two structurally related repeats, each containing four membrane-spanning segments and a helix that is partly inserted into the lipid bilayer. In the full-length protein, the C-terminus interacts with a hydrophobic pocket located at the intracellular side acting as an inhibitor of protein function. Cleavage by caspase-3 at a specific site releases 16 residues of the C-terminus, thus making the pocket accessible to the cytoplasm. Collectively, the work has revealed the unknown architecture of the XKR family and has provided initial insight into its activation by caspases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13157.map.gz emd_13157.map.gz | 20.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13157-v30.xml emd-13157-v30.xml emd-13157.xml emd-13157.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13157_fsc.xml emd_13157_fsc.xml | 8.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13157.png emd_13157.png | 348.4 KB | ||
| マスクデータ |  emd_13157_msk_1.map emd_13157_msk_1.map | 22.2 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13157.cif.gz emd-13157.cif.gz | 6.1 KB | ||
| その他 |  emd_13157_half_map_1.map.gz emd_13157_half_map_1.map.gz emd_13157_half_map_2.map.gz emd_13157_half_map_2.map.gz | 20.5 MB 20.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13157 http://ftp.pdbj.org/pub/emdb/structures/EMD-13157 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13157 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13157 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13157_validation.pdf.gz emd_13157_validation.pdf.gz | 663.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13157_full_validation.pdf.gz emd_13157_full_validation.pdf.gz | 662.7 KB | 表示 | |
| XML形式データ |  emd_13157_validation.xml.gz emd_13157_validation.xml.gz | 12 KB | 表示 | |
| CIF形式データ |  emd_13157_validation.cif.gz emd_13157_validation.cif.gz | 16.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13157 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13157 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13157 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13157 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13157.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13157.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.302 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13157_msk_1.map emd_13157_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_13157_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13157_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : caspase-3 cleaved rXKR9 with sybody
| 全体 | 名称: caspase-3 cleaved rXKR9 with sybody |
|---|---|
| 要素 |
|
-超分子 #1: caspase-3 cleaved rXKR9 with sybody
| 超分子 | 名称: caspase-3 cleaved rXKR9 with sybody / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 17 KDa |
-超分子 #2: rXKR9
| 超分子 | 名称: rXKR9 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Sybody
| 超分子 | 名称: Sybody / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: XK-related protein
| 分子 | 名称: XK-related protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 43.563059 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKYTICNFMM SVLGIIIYVT DLVADIVLTV RYFYDGQYVF GVLTLSFVLC GTLIVHCFSY SWLKDDLKKA GGENEHYFLL LHCLQGGVF TRYWFVLRTG YHVVFKHSHR TSNFMEEQTD PHKEAIDMAT DLSMLRLFET YLEGCPQLIL QLYAFLERGQ A NFSQYMVI ...文字列: MKYTICNFMM SVLGIIIYVT DLVADIVLTV RYFYDGQYVF GVLTLSFVLC GTLIVHCFSY SWLKDDLKKA GGENEHYFLL LHCLQGGVF TRYWFVLRTG YHVVFKHSHR TSNFMEEQTD PHKEAIDMAT DLSMLRLFET YLEGCPQLIL QLYAFLERGQ A NFSQYMVI MVSCCAISWS TVDYQIALRK SLPDKNLLRG FWPKLTYLFY KLFTLLSWML SVVLLLFVDV RTVLLLLLFL WT VGFIWAF INHTQFCNSL SMEFLYRLVV GFILVFTFFN IKGQNTKCPM SCYYTVRVLG TLGILTVFWI YPLSIFNSDY FIP ISATIV LSLLFGIIFL GVYYGTYHPN INAGTQHDEP DGKAPQRDCR IRYFLMDA UniProtKB: XK-related protein |
-分子 #2: Sybody
| 分子 | 名称: Sybody / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.675293 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SQVQLVESGG GSVQAGGSLR LSCAASGNIA DIYYLGWFRQ APGKEREGVA ALITYNGRTY YADSVKGRFT VSLDNAKNTV YLQMNSLKP EDTALYYCAA AYNGLIAAPL KVTRYWYWGQ GTQVTVS |
-分子 #3: DIUNDECYL PHOSPHATIDYL CHOLINE
| 分子 | 名称: DIUNDECYL PHOSPHATIDYL CHOLINE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: PLC |
|---|---|
| 分子量 | 理論値: 622.834 Da |
| Chemical component information |  ChemComp-PLC: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | ||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | This sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 14929 / 平均電子線量: 70.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7p16: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)