+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13071 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The structure of MutS bound to two molecules of ADP-Vanadate | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA mismatch repair protein / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報adenine/cytosine mispair binding / MutS complex / mismatch repair complex / regulation of DNA recombination / mismatched DNA binding / DNA binding, bending / ATP-dependent DNA damage sensor activity / mismatch repair / ADP binding / damaged DNA binding ...adenine/cytosine mispair binding / MutS complex / mismatch repair complex / regulation of DNA recombination / mismatched DNA binding / DNA binding, bending / ATP-dependent DNA damage sensor activity / mismatch repair / ADP binding / damaged DNA binding / DNA damage response / ATP hydrolysis activity / ATP binding / identical protein binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Lamers MH / Borsellini A | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Cryogenic electron microscopy structures reveal how ATP and DNA binding in MutS coordinates sequential steps of DNA mismatch repair. 著者: Alessandro Borsellini / Vladislav Kunetsky / Peter Friedhoff / Meindert H Lamers /   要旨: DNA mismatch repair detects and corrects mismatches introduced during DNA replication. The protein MutS scans for mismatches and coordinates the repair cascade. During this process, MutS undergoes ...DNA mismatch repair detects and corrects mismatches introduced during DNA replication. The protein MutS scans for mismatches and coordinates the repair cascade. During this process, MutS undergoes multiple conformational changes in response to ATP binding, hydrolysis and release, but how ATP induces the various MutS conformations is incompletely understood. Here we present four cryogenic electron microscopy structures of Escherichia coli MutS at sequential stages of the ATP hydrolysis cycle that reveal how ATP binding and hydrolysis induce closing and opening of the MutS dimer, respectively. Biophysical analysis demonstrates how DNA binding modulates the ATPase cycle by prevention of hydrolysis during scanning and mismatch binding, while preventing ADP release in the sliding clamp state. Nucleotide release is achieved when MutS encounters single-stranded DNA that is produced during removal of the daughter strand. The combination of ATP binding and hydrolysis and its modulation by DNA enables MutS to adopt the different conformations needed to coordinate the sequential steps of the mismatch repair cascade. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13071.map.gz emd_13071.map.gz | 1.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13071-v30.xml emd-13071-v30.xml emd-13071.xml emd-13071.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13071_fsc.xml emd_13071_fsc.xml | 6.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13071.png emd_13071.png | 171.4 KB | ||
| Filedesc metadata |  emd-13071.cif.gz emd-13071.cif.gz | 6.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13071 http://ftp.pdbj.org/pub/emdb/structures/EMD-13071 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13071 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13071 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13071_validation.pdf.gz emd_13071_validation.pdf.gz | 462.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13071_full_validation.pdf.gz emd_13071_full_validation.pdf.gz | 461.8 KB | 表示 | |
| XML形式データ |  emd_13071_validation.xml.gz emd_13071_validation.xml.gz | 8.1 KB | 表示 | |
| CIF形式データ |  emd_13071_validation.cif.gz emd_13071_validation.cif.gz | 10.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13071 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13071 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13071 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13071 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13071.map.gz / 形式: CCP4 / 大きさ: 3.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13071.map.gz / 形式: CCP4 / 大きさ: 3.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.32 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : DNA mismatch repair protein MutS
| 全体 | 名称: DNA mismatch repair protein MutS |
|---|---|
| 要素 |
|
-超分子 #1: DNA mismatch repair protein MutS
| 超分子 | 名称: DNA mismatch repair protein MutS / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: MutS bound to two molecules of AMPPNP |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 190 KDa |
-分子 #1: DNA mismatch repair protein MutS
| 分子 | 名称: DNA mismatch repair protein MutS / タイプ: protein_or_peptide / ID: 1 詳細: ADP Vanadate ATP hydrolysis transition state analogue. コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 90.433234 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHMSAI ENFDAHTPMM QQYLRLKAQH PEILLFYRMG DFYELFYDDA KRASQLLDIS LTKRGASAGE PIPMAGIPYH AVENYLAKL VNQGESVAIC EQIGDPATSK GPVERKVVRI VTPGTISDEA LLQERQDNLL AAIWQDSKGF GYATLDISSG R FRLSEPAD ...文字列: HHHHHHMSAI ENFDAHTPMM QQYLRLKAQH PEILLFYRMG DFYELFYDDA KRASQLLDIS LTKRGASAGE PIPMAGIPYH AVENYLAKL VNQGESVAIC EQIGDPATSK GPVERKVVRI VTPGTISDEA LLQERQDNLL AAIWQDSKGF GYATLDISSG R FRLSEPAD RETMAAELQR TNPAELLYAE DFAEMSLIEG RRGLRRRPLW EFEIDTARQQ LNLQFGTRDL VGFGVENAPR GL CAAGCLL QYAKDTQRTT LPHIRSITME REQDSIIMDA ATRRNLEITQ NLAGGAENTL ASVLDCTVTP MGSRMLKRWL HMP VRDTRV LLERQQTIGA LQDFTAGLQP VLRQVGDLER ILARLALRTA RPRDLARMRH AFQQLPELRA QLETVDSAPV QALR EKMGE FAELRDLLER AIIDTPPVLV RDGGVIASGY NEELDEWRAL ADGATDYLER LEVRERERTG LDTLKVGFNA VHGYY IQIS RGQSHLAPIN YMRRQTLKNA ERYIIPELKE YEDKVLTSKG KALALEKQLY EELFDLLLPH LEALQQSASA LAELDV LVN LAERAYTLNY TCPTFIDKPG IRITEGRHPV VEQVLNEPFI ANPLNLSPQR RMLIITGPNM GGKSTYMRQT ALIALMA YI GSYVPAQKVE IGPIDRIFTR VGAADDLASG RSTFMVEMTE TANILHNATE YSLVLMDEIG RGTSTYDGLS LAWACAEN L ANKIKALTLF ATHYFELTQL PEKMEGVANV HLDALEHGDT IAFMHSVQDG AASKSYGLAV AALAGVPKEV IKRARQKLR ELESIS UniProtKB: DNA mismatch repair protein MutS |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 2 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #4: VANADATE ION
| 分子 | 名称: VANADATE ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: VO4 |
|---|---|
| 分子量 | 理論値: 114.939 Da |
| Chemical component information | 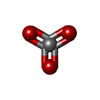 ChemComp-VN3: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.95 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.0004 kPa | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 76 % / チャンバー内温度: 4 K / 装置: LEICA PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-50 / 実像数: 4835 / 平均電子線量: 57.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)























