+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12988 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | rancRNA_18 processing and classification | ||||||||||||
 マップデータ マップデータ | postrpocessed map 60s all particles | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  | ||||||||||||
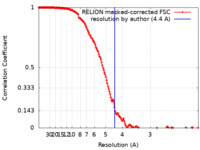
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | ||||||||||||
 データ登録者 データ登録者 | Zuber B / Iacovache I | ||||||||||||
| 資金援助 |  スイス, 3件 スイス, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: RNA Biol / 年: 2021 ジャーナル: RNA Biol / 年: 2021タイトル: A small ribosome-associated ncRNA globally inhibits translation by restricting ribosome dynamics. 著者: Julia Reuther / Lukas Schneider / Ioan Iacovache / Andreas Pircher / Walid H Gharib / Benoît Zuber / Norbert Polacek /  要旨: Ribosome-associated non-coding RNAs (rancRNAs) have been recognized as an emerging class of regulatory molecules capable of fine-tuning translation in all domains of life. RancRNAs are ideally suited ...Ribosome-associated non-coding RNAs (rancRNAs) have been recognized as an emerging class of regulatory molecules capable of fine-tuning translation in all domains of life. RancRNAs are ideally suited for allowing a swift response to changing environments and are therefore considered pivotal during the first wave of stress adaptation. Previously, we identified an mRNA-derived 18 nucleotides long rancRNA (rancRNA_18) in that rapidly downregulates protein synthesis during hyperosmotic stress. However, the molecular mechanism of action remained enigmatic. Here, we combine biochemical, genetic, transcriptome-wide and structural evidence, thus revealing rancRNA_18 as global translation inhibitor by targeting the E-site region of the large ribosomal subunit. Ribosomes carrying rancRNA_18 possess decreased affinity for A-site tRNA and impaired structural dynamics. Cumulatively, these discoveries reveal the mode of action of a rancRNA involved in modulating protein biosynthesis at a thus far unequalled precision. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12988.map.gz emd_12988.map.gz | 27.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12988-v30.xml emd-12988-v30.xml emd-12988.xml emd-12988.xml | 19.6 KB 19.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12988_fsc.xml emd_12988_fsc.xml | 16 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12988.png emd_12988.png | 68.3 KB | ||
| その他 |  emd_12988_additional_1.map.gz emd_12988_additional_1.map.gz emd_12988_additional_2.map.gz emd_12988_additional_2.map.gz emd_12988_additional_3.map.gz emd_12988_additional_3.map.gz emd_12988_additional_4.map.gz emd_12988_additional_4.map.gz | 274.5 MB 273.6 MB 273.8 MB 273.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12988 http://ftp.pdbj.org/pub/emdb/structures/EMD-12988 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12988 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12988 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12988_validation.pdf.gz emd_12988_validation.pdf.gz | 350.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12988_full_validation.pdf.gz emd_12988_full_validation.pdf.gz | 350.2 KB | 表示 | |
| XML形式データ |  emd_12988_validation.xml.gz emd_12988_validation.xml.gz | 15.1 KB | 表示 | |
| CIF形式データ |  emd_12988_validation.cif.gz emd_12988_validation.cif.gz | 20.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12988 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12988 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12988 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12988 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12988.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12988.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | postrpocessed map 60s all particles | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.052 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: refinement of Class 4 after 2D classification
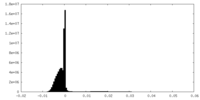
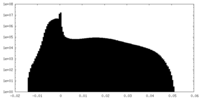
| ファイル | emd_12988_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | refinement of Class 4 after 2D classification | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
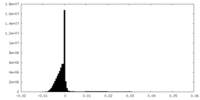
| 密度ヒストグラム |
-追加マップ: refinement 80S all particles
| ファイル | emd_12988_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | refinement 80S all particles | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
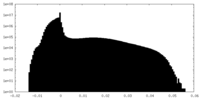
| 密度ヒストグラム |
-追加マップ: refinement of Class 7 after 2D classification
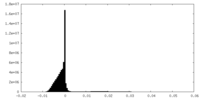
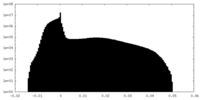
| ファイル | emd_12988_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | refinement of Class 7 after 2D classification | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
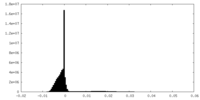
| 密度ヒストグラム |
-追加マップ: refinement of Class 5 after 2D classification
| ファイル | emd_12988_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | refinement of Class 5 after 2D classification | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
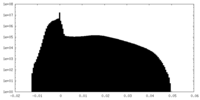
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : rancRNA_18 treated sample - cryoEM analysis and classification
| 全体 | 名称: rancRNA_18 treated sample - cryoEM analysis and classification |
|---|---|
| 要素 |
|
-超分子 #1: rancRNA_18 treated sample - cryoEM analysis and classification
| 超分子 | 名称: rancRNA_18 treated sample - cryoEM analysis and classification タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 0.2 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均電子線量: 70.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.25 mm / 倍率(公称値): 100000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)