+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of undecameric SlyB from Escherichia coli K12 | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | OUTER MEMBRANE CHAPERONE / 2TM GLYCINE ZIPPER / OUTER MEMBRANE LIPOPROTEIN SLYB / LPS-LP BINDING PROTEIN / MEMBRANE PROTEIN | |||||||||||||||
| 機能・相同性 | : / Glycine zipper 2TM domain / Glycine zipper 2TM domain / outer membrane / Prokaryotic membrane lipoprotein lipid attachment site profile. / Outer membrane lipoprotein slyB 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  | |||||||||||||||
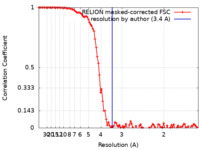
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||||||||
 データ登録者 データ登録者 | Nguyen VS / Remaut H | |||||||||||||||
| 資金援助 |  ベルギー, 4件 ベルギー, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: SlyB encapsulates outer membrane proteins in stress-induced lipid nanodomains 著者: Janssens A / Nguyen VS / Cecil AJ / Van der Verren SE / Timmerman E / Deghelt M / Pak AJ / Collet JF / Impens F / Remaut H | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12950.map.gz emd_12950.map.gz | 85.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12950-v30.xml emd-12950-v30.xml emd-12950.xml emd-12950.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12950_fsc.xml emd_12950_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12950.png emd_12950.png | 174.4 KB | ||
| Filedesc metadata |  emd-12950.cif.gz emd-12950.cif.gz | 6.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12950 http://ftp.pdbj.org/pub/emdb/structures/EMD-12950 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12950 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12950 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12950_validation.pdf.gz emd_12950_validation.pdf.gz | 584.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12950_full_validation.pdf.gz emd_12950_full_validation.pdf.gz | 583.6 KB | 表示 | |
| XML形式データ |  emd_12950_validation.xml.gz emd_12950_validation.xml.gz | 11.6 KB | 表示 | |
| CIF形式データ |  emd_12950_validation.cif.gz emd_12950_validation.cif.gz | 15.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12950 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12950 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12950 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12950 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ojgMC  7ojfC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12950.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12950.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.784 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Outer membrane lipoprotein SlyB
| 全体 | 名称: Outer membrane lipoprotein SlyB |
|---|---|
| 要素 |
|
-超分子 #1: Outer membrane lipoprotein SlyB
| 超分子 | 名称: Outer membrane lipoprotein SlyB / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Undecameric SlyB was purified from overexpression of SlyB-TEV-HIS in E. coli using using 2-step Ni-IMAC purification. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 200 KDa |
-分子 #1: Outer membrane lipoprotein slyB
| 分子 | 名称: Outer membrane lipoprotein slyB / タイプ: protein_or_peptide / ID: 1 / コピー数: 11 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 15.613606 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIKRVLVVSM VGLSLVGCVN NDTLSGDVYT ASEAKQVQNV SYGTIVNVRP VQIQGGDDSN VIGAIGGAVL GGFLGNTVGG GTGRSLATA AGAVAGGVAG QGVQSAMNKT QGVELEIRKD DGNTIMVVQK QGNTRFSPGQ RVVLASNGSQ VTVSPR UniProtKB: Outer membrane lipoprotein slyB |
-分子 #2: PALMITIC ACID
| 分子 | 名称: PALMITIC ACID / タイプ: ligand / ID: 2 / コピー数: 33 / 式: PLM |
|---|---|
| 分子量 | 理論値: 256.424 Da |
| Chemical component information |  ChemComp-PLM: |
-分子 #3: GLYCEROL
| 分子 | 名称: GLYCEROL / タイプ: ligand / ID: 3 / コピー数: 11 / 式: GOL |
|---|---|
| 分子量 | 理論値: 92.094 Da |
| Chemical component information |  ChemComp-GOL: |
-分子 #4: (2~{R},4~{R},5~{R},6~{R})-6-[(1~{R})-1,2-bis(oxidanyl)ethyl]-2-[[...
| 分子 | 名称: (2~{R},4~{R},5~{R},6~{R})-6-[(1~{R})-1,2-bis(oxidanyl)ethyl]-2-[[(2~{R},3~{S},4~{R},5~{R},6~{R})-5-[[(~{E},3~{R})-3-dodecanoyloxytetradec-5-enoyl]amino]-6-[[(2~{R},3~{S},4~{R},5~{R},6~{R})-3- ...名称: (2~{R},4~{R},5~{R},6~{R})-6-[(1~{R})-1,2-bis(oxidanyl)ethyl]-2-[[(2~{R},3~{S},4~{R},5~{R},6~{R})-5-[[(~{E},3~{R})-3-dodecanoyloxytetradec-5-enoyl]amino]-6-[[(2~{R},3~{S},4~{R},5~{R},6~{R})-3-oxidanyl-5-[[(~{E},3~{R})-3-oxidanyltetradec-11-enoyl]amino]-4-[(~{E},3~{R})-3-oxidanyltetradec-5-enoyl]oxy-6-phosphonooxy-oxan-2-yl]methoxy]-3-phosphonooxy-4-[(~{E},3~{R})-3-tetradecanoyloxytetradec-7-enoyl]oxy-oxan-2-yl]methoxy]-4,5-bis(oxidanyl)oxane-2-carboxylic acid タイプ: ligand / ID: 4 / コピー数: 11 / 式: L8Z |
|---|---|
| 分子量 | 理論値: 2.010478 KDa |
| Chemical component information | 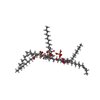 ChemComp-L8Z: |
-分子 #5: 2-(HEXADECANOYLOXY)-1-[(PHOSPHONOOXY)METHYL]ETHYL HEXADECANOATE
| 分子 | 名称: 2-(HEXADECANOYLOXY)-1-[(PHOSPHONOOXY)METHYL]ETHYL HEXADECANOATE タイプ: ligand / ID: 5 / コピー数: 11 / 式: LPP |
|---|---|
| 分子量 | 理論値: 648.891 Da |
| Chemical component information |  ChemComp-LPP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.04 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: GATAN CRYOPLUNGE 3 / 詳細: Back-blotting for 4 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 6352 / 平均露光時間: 3.0 sec. / 平均電子線量: 61.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.55 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 35 |
|---|---|
| 得られたモデル |  PDB-7ojg: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)