+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mouse RNF213, with mixed nucleotides bound | |||||||||||||||
 マップデータ マップデータ | full non-sharpened map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | nucleotide / AAA / RNF213 / E3 ligase / SIGNALING PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報lipid ubiquitination / negative regulation of non-canonical Wnt signaling pathway / lipid droplet formation / xenophagy / Antigen processing: Ubiquitination & Proteasome degradation / sprouting angiogenesis / 転移酵素; アシル基を移すもの; アミノアシル基を移すもの / regulation of lipid metabolic process / protein K63-linked ubiquitination / immune system process ...lipid ubiquitination / negative regulation of non-canonical Wnt signaling pathway / lipid droplet formation / xenophagy / Antigen processing: Ubiquitination & Proteasome degradation / sprouting angiogenesis / 転移酵素; アシル基を移すもの; アミノアシル基を移すもの / regulation of lipid metabolic process / protein K63-linked ubiquitination / immune system process / protein autoubiquitination / lipid droplet / RING-type E3 ubiquitin transferase / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / ubiquitin-protein transferase activity / ubiquitin protein ligase activity / ubiquitin-dependent protein catabolic process / angiogenesis / defense response to bacterium / protein ubiquitination / ATP hydrolysis activity / zinc ion binding / ATP binding / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
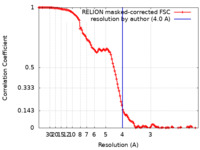
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||||||||
 データ登録者 データ登録者 | Ahel J / Clausen T | |||||||||||||||
| 資金援助 |  オーストリア, オーストリア,  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: E3 ubiquitin ligase RNF213 employs a non-canonical zinc finger active site and is allosterically regulated by ATP 著者: Ahel J / Fletcher AJ / Grabarczyk D / Roitinger E / Deszcz L / Lehner A / Virdee S / Clausen T | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12932.map.gz emd_12932.map.gz | 131.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12932-v30.xml emd-12932-v30.xml emd-12932.xml emd-12932.xml | 34.9 KB 34.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12932_fsc.xml emd_12932_fsc.xml | 12.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12932.png emd_12932.png | 139.4 KB | ||
| マスクデータ |  emd_12932_msk_1.map emd_12932_msk_1.map emd_12932_msk_2.map emd_12932_msk_2.map | 166.4 MB 166.4 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12932.cif.gz emd-12932.cif.gz | 10 KB | ||
| その他 |  emd_12932_additional_1.map.gz emd_12932_additional_1.map.gz emd_12932_additional_2.map.gz emd_12932_additional_2.map.gz emd_12932_additional_3.map.gz emd_12932_additional_3.map.gz emd_12932_additional_4.map.gz emd_12932_additional_4.map.gz emd_12932_additional_5.map.gz emd_12932_additional_5.map.gz emd_12932_half_map_1.map.gz emd_12932_half_map_1.map.gz emd_12932_half_map_2.map.gz emd_12932_half_map_2.map.gz | 152.8 MB 152.5 MB 131.1 MB 131.5 MB 131.5 MB 131.6 MB 131.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12932 http://ftp.pdbj.org/pub/emdb/structures/EMD-12932 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12932 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12932 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12932_validation.pdf.gz emd_12932_validation.pdf.gz | 939.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12932_full_validation.pdf.gz emd_12932_full_validation.pdf.gz | 939.1 KB | 表示 | |
| XML形式データ |  emd_12932_validation.xml.gz emd_12932_validation.xml.gz | 19.8 KB | 表示 | |
| CIF形式データ |  emd_12932_validation.cif.gz emd_12932_validation.cif.gz | 26.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12932 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12932 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12932 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12932 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7oimMC  7oikC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 電子顕微鏡画像生データ |  EMPIAR-10712 (タイトル: Transmission electron micrographs of mouse RNF213 incubated with ATPγS EMPIAR-10712 (タイトル: Transmission electron micrographs of mouse RNF213 incubated with ATPγSData size: 2.3 TB Data #1: unaligned multi-frame micrographs from a dataset collected without grid tilting [micrographs - multiframe] Data #2: unaligned multi-frame micrographs from a dataset collected with a 30 deg grid tilt [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12932.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12932.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full non-sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.053 Å | ||||||||||||||||||||||||||||||||||||
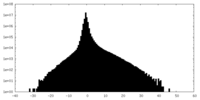
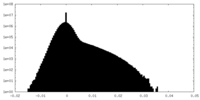
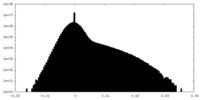
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_12932_msk_1.map emd_12932_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
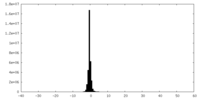
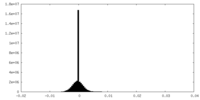
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_12932_msk_2.map emd_12932_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
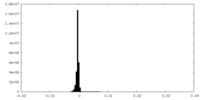
| 密度ヒストグラム |
-追加マップ: full sharpened map
| ファイル | emd_12932_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
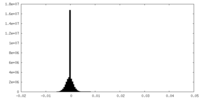
| 密度ヒストグラム |
-追加マップ: sharpened focused map, focused on AAA core
| ファイル | emd_12932_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened focused map, focused on AAA core | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: non-sharpened focused map, focused on AAA core
| ファイル | emd_12932_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | non-sharpened focused map, focused on AAA core | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: non-sharpened focused half map 1, focused on AAA cpre
| ファイル | emd_12932_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | non-sharpened focused half map 1, focused on AAA cpre | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: non-sharpened focused half map 2, focused on AAA cpre
| ファイル | emd_12932_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | non-sharpened focused half map 2, focused on AAA cpre | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: full non-sharpened map, half volume 2
| ファイル | emd_12932_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full non-sharpened map, half volume 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: full non-sharpened map, half volume 1
| ファイル | emd_12932_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full non-sharpened map, half volume 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RNF213 incubated with ATPgS
| 全体 | 名称: RNF213 incubated with ATPgS |
|---|---|
| 要素 |
|
-超分子 #1: RNF213 incubated with ATPgS
| 超分子 | 名称: RNF213 incubated with ATPgS / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 580 KDa |
-分子 #1: E3 ubiquitin-protein ligase RNF213
| 分子 | 名称: E3 ubiquitin-protein ligase RNF213 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RING-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 552.623375 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MASWSHPQFE KGSTSAFNPR DTVTVYFHAI VSRHFGFNPE EHKVYVRGGE GLGQKGWTDA CEMYCTQDLH DLGSLVEGKM DIPRQSLDK PIPYKYVIHR GGSSKDTVEY EFIYEQAQKK GEHVNRCLRV VSTSLGNGDW HQYDDIICMR STGFFQQAKN R ILDSTRKE ...文字列: MASWSHPQFE KGSTSAFNPR DTVTVYFHAI VSRHFGFNPE EHKVYVRGGE GLGQKGWTDA CEMYCTQDLH DLGSLVEGKM DIPRQSLDK PIPYKYVIHR GGSSKDTVEY EFIYEQAQKK GEHVNRCLRV VSTSLGNGDW HQYDDIICMR STGFFQQAKN R ILDSTRKE LLKGKKQAAV VMLDRIFSVL QPWSDINLQS FMTQFLQFYS VVREPMIHDG RARKWTSLQY EEKEVWTNLW EH VKKQMAP FLEGKSGESL PADCPVRSKL TLGLSILFMV EAAEFTVPKK DLDSLCYLLI PSAGSPEALH SDLSPVLRIR QRW RIYLTN LCLRCIDERC DRWLGILPLL HTCMQKSPPK KNSKSQPEDT WAGLEGISFS EFRDKAPTRS QPLQFMQSKM ALLR VDEYL FRSWLSVVPL ESLSSYLENS IDYLSDVPVR VLDCLQGISY RLPGLRKISN QNMKKDVENV FKMLMHLVDI YQHRI FGEN LLQIYLTECL TLHETVCNIT ANHQFFEIPA LSAELICKLL ELSPPGHTDE GLPEKSYEDL VTSTLQEALA TTRNWL RSL FKSRMLSISS AYVRLTYSEE MAVWRRLVEI GFPEKHGWKG SLLGDMEGRL KQEPPRLQIS FFCSSQCRDG GLHDSVS RS FEKCVIEAVS SACQSQTSVL EGLSCQDLQK FGTLLSAVIT KSWPVHNGEP VFDVDEIFKY LLKWPDVRQL FELCGTNE K IIDNITEEGR QLMATAESVF QKVAGELENG TIVVGQLELI LEHQSQFLDI WNLNRRRLPS QEKACDVRSL LKRRRDDLL FLKQEKRYVE SLLRQLGRVK HLVQVDFGNI EIIHSQDLSN KKLNEAVIKL PNSSSYKRET HYCLSPDIRE MASKLDSLKD SHIFQDFWQ ETAESLNTLD KDPRELKVSL PEVLEYLYNP CYDNFYTLYE NLKSGKITFA EVDAIFKDFV DKYDELKNDL K FMCTMNPQ DQKGWISERV GQIKEYHTLH QAVSSAKVIL QVRRALGVTG DFSVLNPLLN FADSFEDFGN EKLDQISPQF IK AKQLLQD ISEPRQRCLE ELARQTELVA WLHKALEDIN ELKVFVDLAS ISAGENDIDV DRVACFHDAV QGYASLLYKM DER TNFSDF MNHLQELWRA LDNDQHLPDK LKDSARNLEW LKTVKESHGS VELSSLSLAT AINSRGVYVI EAPKDGQKIS PDTV LRLLL PDGHGYPEAL RTYSTEELKE LLNKLMLMSG KKDHNSNTEV EKFSEVFSNM QRLVHVFIKL HCAGNMLFRT WTAKV YCCP DGGIFMNFGL ELLSQLTEKG DVIQLLGALC RQMEDFLDNW KTVVAQKRAE HFYLNFYTAE QLVYLSSELR KPRPSE AAL MMLSFIKGKC TVQDLVQATS ACESKADRYC LREVMKKLPQ QLLSEPSLMG KLQVIMMQSL VYMSAFLPHC LDLDALG RC LAHLATMGGT PVERPLPKGL QAGQPNLILC GHSEVLPAAL AIYMQAPRQP LPTFDEVLLC TPATTIEEVE LLLRRCLT S GSQGHKVYSL LFADQLSYEV GCQAEEFFQS LCTRAHREDY QLVILCDAAR EHCYIPSTFS QYKVPLVPQA PLPNIQAYL QSHYQVPKRL LSAATVFRDG LCVGIVTSER AGVGKSLYVN TLHTKLKAKL RDETVPLKII RLTEPHLDEN QVLSALLPFL KEKYQKMPV IFHIDISTSV QTGIPIFLFK LLILQYLMDI NGKIWRRSPG HLYLVEIPQG LSVQPKRSSK LNARAPLFKF L DLFPKVTC RPPKEVIDME LTPERSHTDP AMDPVEFCSE AFQRPYQYLK RFHQQQNLDT FQYEKGSVEG SPEECLQHFL IY CGLINPS WSELRNFAWF LNCQLKDCEA SIFCKSAFTG DTLRGFKNFV VTFMILMARD FATPTLHTSD QSPGRQSVTI GEV VEEDLA PFSLRKRWES EPHPYVFFNG DHMTMTFIGF HLETNNNGYV DAINPSNGKV IKKDVMTKEL FDGLRLQRVP FNID FDNLP RYEKLERLCL ALGIEWPIDP DETYELTTDN MLKILAIEMR FRCGIPVIIM GETGCGKTRL IKFLSDLKRG SVEAE TMKL VKVHGGTTPS MIYSKVKEAE RTAFSNKAQH KLDTILFFDE ANTTEAVSCI KEILCDRTVD GEHLHEDSGL HIIAAC NPY RKHSQEMILR LESAGLGYRV SAEETADRLG SIPLRQLVYR VHALPPSLIP LVWDFGQLND SAEKLYIQQI VQRLVDS VS VNPSETCVIA DVLSASQMFM RKRENECGFV SLRDVERCVK VFRWFHDHSD MLLKELDKFL HESSDSTHTF ERDPVLWS L VMAIGVCYHA SLEEKASYRT AIARCFPKPY NSSRAILDEV THVQDLFLRG APIRTNIARN LALKENVFMM VICIELKIP LFLVGKPGSS KSLAKIIVAD AMQGQAAFSE LFRCLKQVHL VSFQCSPHST PQGIISTFKQ CARFQQGKDL GQYVSVVVLD EVGLAEDSP KMPLKTLHPL LEDGCIEDDP APYKKVGFVG ISNWALDPAK MNRGIFVSRG SPNEKELIES AEGICSSDRL V QDKIRGYF APFAKAYETV CQKQDKEFFG LRDYYSLIKM VFAKAKASKR GLSPQDITHA VLRNFSGKDN IQALSIFTAS LP EARYKEE VSTVELIKQN IYPGPQASSR GLDGAESRYL LVLTRNYVAL QILQQTFFEG QQPEIIFGSS FPQDQEYTQI CRN INRVKI CMETGKMVVL LNLQNLYESL YDALNQYYVY LGGQKYVDLG LGTHRVKCRV HTAFRLIVIE EKDVVYKQFP VPLI NRLEK HYLDMNTVLQ PWQKSIVQEL QQWAHEFADV KADQFIARHK YSPADVFIGY HSDACASVVL QAVERQGCRD LTEEL YRKV SEEARSILLD CATPDAVVRL SGSSLGSFTA KQLSQEYYYA QQHNSFVDFL QAHLRMTHHE CRAVFTEITT FSRLLT GND CDVLASELRG LASKPVVLSL QQYDTEYSFL KDVRSWLTNP GKRKVLVIQA DFDDGTRSAQ LVASAKYTAI NEINKTQ GT KDFVFVYFVT KLSRMGSGTS YVGFHGGLWR SVHIDDLRRS TIMASDVTKL QNVTISQLFK PEDKPEQEEM EIETSQSK E LAEEQMEVED SEEMKKASDP RSCDCSQFLD TTRLVQSCVQ GAVGMLRDQN ESCARNMRRV TILLDLLNED NTRNASFLR ESKMRLHVLL NKQEENQVRS LKEWVTREAA NQDALQEAGT FRHTLWKRVQ DVVTPILASM IAHIDRDGNL ELLAQPDSPA WVQDLWMFI YSDIKFLNIS LVLNNTRSNS EMSFILVQSH MNLLKDAYNA VPFSWRIRDY LEELWVQAQY ITDTEGLSKK F VEIFQKTP LGVFLAQFPV AQQQKLLQSY LKDFLLLTMK VSSREELMFL QMALWSCLRE LQEASGTPDE TYKFPLSLPW VH LAFQHFR TRLQNFSRIL TIHPQVLSSL SQAAEKHSLA GCEMTLDAFA AMACAEMLKG DLLKPSPKAW LQLVKNLSTP LEL VCSEGY LCDSGSMTRS VIQEVRALWN RIFSIALFVE HVLLGTESHI PELSPLVTTY VSLLDKCLEE DSNLKTCRPF VAVM TTLCD CKDKASKKFS RFGIQPCFIC HGDAQDPVCL PCDHVYCLRC IQTWLIPGQM MCPYCLTDLP DKFSPTVSQD HRKAI EKHA QFRHMCNSFF VDLVSTMCFK DNTPPEKSVI DTLLSLLFVQ KELLRDASQK HREHTKSLSP FDDVVDQTPV IRSVLL KLL LKYSFHEVKD YIQNYLTQLE KKAFLTEDKT ELYLLFISCL EDSVHQKTSA GCRNLEQVLR EEGHFLRTYS PGLQGQE PV RIASVEYLQE VARVRLCLDL AADFLSELQE GSELAEDKRR FLKHVEEFCT RVNNDWHRVY LVRKLSSQRG MEFVQSFS K QGHPCQWVFP RKVIAQQKDH VSLMDRYLVH GNEYKAVRDA TAKAVLECKT LDIGNALMAC RSPKPQQTAY LLLALYTEV AALYRSPNGS LHPEAKQLEA VNKFIKESKI LSDPNIRCFA RSLVDNTLPL LKIRSANSIL KGTVTEMAVH VATILLCGHN QILKPLRNL AFYPVNMANA FLPTMPEDLL VHARTWRGLE NVTWYTCPRG HPCSVGECGR PMQESTCLDC GLPVGGLNHT P HEGFSAIR NNEDRTQTGH VLGSPQSSGV AEVSDRGQSP VVFILTRLLT HLAMLVGATH NPQALTVIIK PWVQDPQGFL QQ HIQRDLE QLTKMLGRSA DETIHVVHLI LSSLLRVQSH GVLNFNAELS TKGCRNNWEK HFETLLLREL KHLDKNLPAI NAL ISQDER ISSNPVTKII YGDPATFLPH LPQKSIIHCS KIWSCRRKIT VEYLQHIVEQ KNGKETVPVL WHFLQKEAEL RLVK FLPEI LALQRDLVKQ FQNVSRVEYS SIRGFIHSHS SDGLRKLLHD RITIFLSTWN ALRRSLETNG EIKLPKDYCC SDLDL DAEF EVILPRRQGL GLCGTALVSY LISLHNNMVY TVQKFSNEDN SYSVDISEVA DLHVISYEVE RDLNPLILSN CQYQVQ QGG ETSQEFDLEK IQRQISSRFL QGKPRLTLKG IPTLVYRRDW NYEHLFMDIK NKMAQSSLPN LAISTISGQL QSYSDAC EA LSIIEITLGF LSTAGGDPGM DLNVYIEEVL RMCDQTAQVL KAFSRCQLRH IIALWQFLSA HKSEQRLRLN KELFREID V QYKEELSTQH QRLLGTFLNE AGLDAFLLEL HEMIVLKLKG PRAANSFNPN WSLKDTLVSY METKDSDILS EVESQFPEE ILMSSCISVW KIAATRKWDR QSRGGGHHHH HHHHHH UniProtKB: E3 ubiquitin-protein ligase RNF213 |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #5: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #6: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 6 / コピー数: 1 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 / 構成要素:
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil R2/2 / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||
| 凍結 | 凍結剤: ETHANE / 装置: LEICA PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) #0 - 撮影したグリッド数: 1 / #0 - 実像数: 3055 / #0 - 平均露光時間: 2.0 sec. / #0 - 平均電子線量: 45.0 e/Å2 / #0 - 詳細: grid tilted 30 degrees 1 image per "hole" / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) #1 - 検出モード: COUNTING / #1 - デジタル化 - サイズ - 横: 3838 pixel / #1 - デジタル化 - サイズ - 縦: 3710 pixel / #1 - デジタル化 - 画像ごとのフレーム数: 1-40 / #1 - 撮影したグリッド数: 1 / #1 - 実像数: 5950 / #1 - 平均露光時間: 10.0 sec. / #1 - 平均電子線量: 40.5 e/Å2 / #1 - 詳細: non-tilted dataset 10 images per "hole" |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.5 µm / 最小 デフォーカス(公称値): -1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)