+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12226 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Vps35/Vps29 arch of fungal membrane-assembled retromer:Vps5 (SNX-BAR) complex. | ||||||||||||||||||
 マップデータ マップデータ | LAFTER-filtered map of Vps35/Vps29 arch of fungal retromer:Vps5 assembled on the membrane | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード |  endosomes (エンドソーム) / coat proteins / endosomes (エンドソーム) / coat proteins /  membrane trafficking / cargo-sorting / membrane trafficking / cargo-sorting /  ENDOCYTOSIS (エンドサイトーシス) ENDOCYTOSIS (エンドサイトーシス) | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 retromer, cargo-selective complex / retromer, cargo-selective complex /  retromer complex / retromer complex /  retrograde transport, endosome to Golgi / retrograde transport, endosome to Golgi /  protein transport / protein transport /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) / Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) /  Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) | ||||||||||||||||||
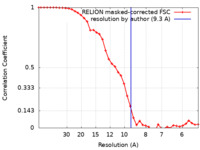
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 9.3 Å クライオ電子顕微鏡法 / 解像度: 9.3 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Leneva N / Kovtun O / Morado DR / Briggs JAG / Owen DJ | ||||||||||||||||||
| 資金援助 |  英国, 5件 英国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: Architecture and mechanism of metazoan retromer:SNX3 tubular coat assembly. 著者: Natalya Leneva / Oleksiy Kovtun / Dustin R Morado / John A G Briggs / David J Owen /  要旨: Retromer is a master regulator of cargo retrieval from endosomes, which is critical for many cellular processes including signaling, immunity, neuroprotection, and virus infection. The retromer core ...Retromer is a master regulator of cargo retrieval from endosomes, which is critical for many cellular processes including signaling, immunity, neuroprotection, and virus infection. The retromer core (VPS26/VPS29/VPS35) is present on cargo-transporting, tubular carriers along with a range of sorting nexins. Here, we elucidate the structural basis of membrane tubulation and coupled cargo recognition by metazoan and fungal retromer coats assembled with the non-Bin1/Amphiphysin/Rvs (BAR) sorting nexin SNX3 using cryo-electron tomography. The retromer core retains its arched, scaffolding structure but changes its mode of membrane recruitment when assembled with different SNX adaptors, allowing cargo recognition at subunit interfaces. Thus, membrane bending and cargo incorporation can be modulated to allow retromer to traffic cargoes along different cellular transport routes. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12226.map.gz emd_12226.map.gz | 4.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12226-v30.xml emd-12226-v30.xml emd-12226.xml emd-12226.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12226_fsc.xml emd_12226_fsc.xml | 3.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12226.png emd_12226.png | 93.7 KB | ||
| マスクデータ |  emd_12226_msk_1.map emd_12226_msk_1.map | 4.3 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12226.cif.gz emd-12226.cif.gz | 6 KB | ||
| その他 |  emd_12226_half_map_1.map.gz emd_12226_half_map_1.map.gz emd_12226_half_map_2.map.gz emd_12226_half_map_2.map.gz | 4.1 MB 4.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12226 http://ftp.pdbj.org/pub/emdb/structures/EMD-12226 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12226 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12226 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12226.map.gz / 形式: CCP4 / 大きさ: 4.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12226.map.gz / 形式: CCP4 / 大きさ: 4.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LAFTER-filtered map of Vps35/Vps29 arch of fungal retromer:Vps5 assembled on the membrane | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12226_msk_1.map emd_12226_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-map2
| ファイル | emd_12226_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-map1
| ファイル | emd_12226_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : arch assembly (Vps35/Vps29) of the fungal retromer:Vps5 complex.
| 全体 | 名称: arch assembly (Vps35/Vps29) of the fungal retromer:Vps5 complex. |
|---|---|
| 要素 |
|
-超分子 #1: arch assembly (Vps35/Vps29) of the fungal retromer:Vps5 complex.
| 超分子 | 名称: arch assembly (Vps35/Vps29) of the fungal retromer:Vps5 complex. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: fungal retromer:Vps5 complex assembled on liposomes. |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) |
-分子 #1: Vacuolar protein sorting-associated protein 35
| 分子 | 名称: Vacuolar protein sorting-associated protein 35 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類)株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 98.566781 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MSTPAPPEEQ ARLLEDALIA VRQQTAMMRK FLDTPGKLMD ALKCCSTLVS ELRTSSLSPK QYYELYMAVF DALRYLSAHL RENHPVNHL ADLYELVQYA GNIIPRLYLM ITVGTAYMSI DGAPVKELMK DMMDMSRGVQ HPVRGLFLRY YLSGQARDYL P TGDSDGPE ...文字列: MSTPAPPEEQ ARLLEDALIA VRQQTAMMRK FLDTPGKLMD ALKCCSTLVS ELRTSSLSPK QYYELYMAVF DALRYLSAHL RENHPVNHL ADLYELVQYA GNIIPRLYLM ITVGTAYMSI DGAPVKELMK DMMDMSRGVQ HPVRGLFLRY YLSGQARDYL P TGDSDGPE GNLQDSINFI LTNFVEMNKL WVRLQHQGHS RERDLRTQER RELQLLVGSN IVRLSQLVDL PTYRDSILGP LL EQIVQCR DILAQEYLLE VITQVFPDEY HLHTLDQFLG AVSRLNPHVN VKAIVIGMMN RLSDYAERES QNEPEEDRAK LEE EALAKL LEKTKLGQNS ELEPQNGDHP DTEVSSTTDS AQAPSTADSD TTAVNGEEEP VRKRRGIPVN VPLYDIFFDQ VQHL VQAQH LPIQDTIALC CSLANLSLNI YPERLDYVDG ILAYALAKVK EHANSADLHS QPAQQSLLSL LQSPLRRYVS IFTAL SLPT YVSLFQAQTY PTRRAIAGEI VRTLLKNQTL ISTPAHLENV LEILKVLIKE GSQPPAGYPG VVQPRARPLE TDETME EQG WLARLVHLIH SDDNDTQFRL LQMTRKAYAE GNERIRTTTP PLITAGLKLA RRFKAREHYD DNWSSQSSSL FKFLHSA IS TLYTRVNGPG VADLCLRLFC SCGQVADMTE FEEVAYEFFA QAFTVYEESI SDSKAQFQAV CVIASALHRT RNFGRENY D TLITKCAQHA SKLLRKPDQC RAVYLASHLW WATPIAARGE TEDTELYRDG KRVLECLQRA LRVADSCMET ATSIELFVE ILDRYVYYFD QRNESVTTKY LNGLIELIHS NLAGNQQDSA SVEASRKHFI QTLEMIQSKE FEGIVVAPK UniProtKB:  Vacuolar protein sorting-associated protein 35 Vacuolar protein sorting-associated protein 35 |
-分子 #2: Vacuolar protein sorting-associated protein 29
| 分子 | 名称: Vacuolar protein sorting-associated protein 29 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類)株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 22.330602 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: SMAFLILVIG NLHIPDRALD IPPKFKKLLS PGKISQTLCL GNLTDRATYD YLRSISPDLK IVRGRMDVEA TSLPLMQVVT HGSLRIGFL EGFTLVSEEP DVLLAEANKL DVDVLCWAGG SHRFECFEYM DKFFVNPGSA TGAFTTDWLA EGEEVVPSFC L MDVQGISL ...文字列: SMAFLILVIG NLHIPDRALD IPPKFKKLLS PGKISQTLCL GNLTDRATYD YLRSISPDLK IVRGRMDVEA TSLPLMQVVT HGSLRIGFL EGFTLVSEEP DVLLAEANKL DVDVLCWAGG SHRFECFEYM DKFFVNPGSA TGAFTTDWLA EGEEVVPSFC L MDVQGISL TLYVYQLRKD ENGTENVAVE KVTYTKPVEP TGAS UniProtKB:  Vacuolar protein sorting-associated protein 29 Vacuolar protein sorting-associated protein 29 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 3.17 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z
Z Y
Y X
X