+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11909 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | N-terminally truncated WzzB from E.coli - C8 symmetry | |||||||||
 マップデータ マップデータ | N-terminally truncated WzzB from E.coli - C8 symmetry | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Polysaccharide chain length determinant N-terminal domain / Chain length determinant protein / : / lipopolysaccharide biosynthetic process / protein tyrosine kinase activity / plasma membrane / Chain length determinant protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||
 データ登録者 データ登録者 | Wiseman B / Nitharwal RG / Hogbom M | |||||||||
| 資金援助 |  スウェーデン, 2件 スウェーデン, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structure of a full-length bacterial polysaccharide co-polymerase. 著者: Benjamin Wiseman / Ram Gopal Nitharwal / Göran Widmalm / Martin Högbom /   要旨: Lipopolysaccharides are important components of the bacterial cell envelope that among other things act as a protective barrier against the environment and toxic molecules such as antibiotics. One of ...Lipopolysaccharides are important components of the bacterial cell envelope that among other things act as a protective barrier against the environment and toxic molecules such as antibiotics. One of the most widely disseminated pathways of polysaccharide biosynthesis is the inner membrane bound Wzy-dependent pathway. Here we present the 3.0 Å structure of the co-polymerase component of this pathway, WzzB from E. coli solved by single-particle cryo-electron microscopy. The overall architecture is octameric and resembles a box jellyfish containing a large bell-shaped periplasmic domain with the 2-helix transmembrane domain from each protomer, positioned 32 Å apart, encircling a large empty transmembrane chamber. This structure also reveals the architecture of the transmembrane domain, including the location of key residues for the Wzz-family of proteins and the Wzy-dependent pathway present in many Gram-negative bacteria, explaining several of the previous biochemical and mutational studies and lays the foundation for future investigations. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11909.map.gz emd_11909.map.gz | 1.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11909-v30.xml emd-11909-v30.xml emd-11909.xml emd-11909.xml | 12.9 KB 12.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
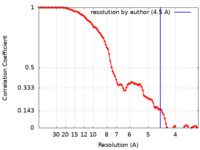
| FSC (解像度算出) |  emd_11909_fsc.xml emd_11909_fsc.xml | 10.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11909.png emd_11909.png | 41.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11909 http://ftp.pdbj.org/pub/emdb/structures/EMD-11909 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11909 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11909 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11909_validation.pdf.gz emd_11909_validation.pdf.gz | 246.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11909_full_validation.pdf.gz emd_11909_full_validation.pdf.gz | 245.4 KB | 表示 | |
| XML形式データ |  emd_11909_validation.xml.gz emd_11909_validation.xml.gz | 11.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11909 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11909 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11909 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11909 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11909.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11909.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | N-terminally truncated WzzB from E.coli - C8 symmetry | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Octameric bacterial polysaccharide co-polymerase complex
| 全体 | 名称: Octameric bacterial polysaccharide co-polymerase complex |
|---|---|
| 要素 |
|
-超分子 #1: Octameric bacterial polysaccharide co-polymerase complex
| 超分子 | 名称: Octameric bacterial polysaccharide co-polymerase complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 300 KDa |
-分子 #1: Chain length determinant protein
| 分子 | 名称: Chain length determinant protein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQIDLIDLLV QLWRGKMTII ISVIVAIALA IGYLAVAKEK WTSTAIITQP DVGQIAGYNN AMNVI YGQA APKVSDLQET LIGRFSSAFS ALAETLDNQE EREKLTIEPS VKNQQLPLTV SYVGQTAEGA QMKLAQYIQQ VDDKVN QEL EKDLKDNIAL GRKNLQDSLR ...文字列: MQIDLIDLLV QLWRGKMTII ISVIVAIALA IGYLAVAKEK WTSTAIITQP DVGQIAGYNN AMNVI YGQA APKVSDLQET LIGRFSSAFS ALAETLDNQE EREKLTIEPS VKNQQLPLTV SYVGQTAEGA QMKLAQYIQQ VDDKVN QEL EKDLKDNIAL GRKNLQDSLR TQEVVAQEQK DLRIRQIQEA LQYANQAQVT KPQIQQTGED ITQDTLFLLG SEALESM IK HEATRPLVFS PNYYQTRQNL LDIESLKVDD LDIHAYRYVM KPMLPIRRDS PKKAITLILA VLLGGMVGAG IVLGRNAL R NYNAKEFRVP GSHHHHHHHH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R2/2 / 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE / 詳細: 40 seconds at 20 mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 2050 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)