[日本語] English
 万見
万見- EMDB-11861: Helical reconstruction of nitrite oxidoreductase from anammox bacteria -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11861 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Helical reconstruction of nitrite oxidoreductase from anammox bacteria | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nitrate reductase / anammoxosome / anaerobic respiration / cell envelope / 3 iron, 4 sulfur cluster binding / 4 iron, 4 sulfur cluster binding / periplasmic space / electron transfer activity / oxidoreductase activity / heme binding ...nitrate reductase / anammoxosome / anaerobic respiration / cell envelope / 3 iron, 4 sulfur cluster binding / 4 iron, 4 sulfur cluster binding / periplasmic space / electron transfer activity / oxidoreductase activity / heme binding / metal ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Candidatus Kuenenia stuttgartiensis (バクテリア) Candidatus Kuenenia stuttgartiensis (バクテリア) | |||||||||
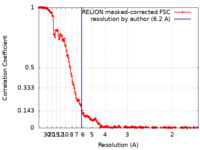
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 6.2 Å | |||||||||
 データ登録者 データ登録者 | Parey K | |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2021 ジャーナル: Nat Microbiol / 年: 2021タイトル: Structural and functional characterization of the intracellular filament-forming nitrite oxidoreductase multiprotein complex. 著者: Tadeo Moreno Chicano / Lea Dietrich / Naomi M de Almeida / Mohd Akram / Elisabeth Hartmann / Franziska Leidreiter / Daniel Leopoldus / Melanie Mueller / Ricardo Sánchez / Guylaine H L ...著者: Tadeo Moreno Chicano / Lea Dietrich / Naomi M de Almeida / Mohd Akram / Elisabeth Hartmann / Franziska Leidreiter / Daniel Leopoldus / Melanie Mueller / Ricardo Sánchez / Guylaine H L Nuijten / Joachim Reimann / Kerstin-Anikó Seifert / Ilme Schlichting / Laura van Niftrik / Mike S M Jetten / Andreas Dietl / Boran Kartal / Kristian Parey / Thomas R M Barends /   要旨: Nitrate is an abundant nutrient and electron acceptor throughout Earth's biosphere. Virtually all nitrate in nature is produced by the oxidation of nitrite by the nitrite oxidoreductase (NXR) ...Nitrate is an abundant nutrient and electron acceptor throughout Earth's biosphere. Virtually all nitrate in nature is produced by the oxidation of nitrite by the nitrite oxidoreductase (NXR) multiprotein complex. NXR is a crucial enzyme in the global biological nitrogen cycle, and is found in nitrite-oxidizing bacteria (including comammox organisms), which generate the bulk of the nitrate in the environment, and in anaerobic ammonium-oxidizing (anammox) bacteria which produce half of the dinitrogen gas in our atmosphere. However, despite its central role in biology and decades of intense study, no structural information on NXR is available. Here, we present a structural and biochemical analysis of the NXR from the anammox bacterium Kuenenia stuttgartiensis, integrating X-ray crystallography, cryo-electron tomography, helical reconstruction cryo-electron microscopy, interaction and reconstitution studies and enzyme kinetics. We find that NXR catalyses both nitrite oxidation and nitrate reduction, and show that in the cell, NXR is arranged in tubules several hundred nanometres long. We reveal the tubule architecture and show that tubule formation is induced by a previously unidentified, haem-containing subunit, NXR-T. The results also reveal unexpected features in the active site of the enzyme, an unusual cofactor coordination in the protein's electron transport chain, and elucidate the electron transfer pathways within the complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11861.map.gz emd_11861.map.gz | 75.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11861-v30.xml emd-11861-v30.xml emd-11861.xml emd-11861.xml | 22.3 KB 22.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11861_fsc.xml emd_11861_fsc.xml | 17.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11861.png emd_11861.png | 224.3 KB | ||
| マスクデータ |  emd_11861_msk_1.map emd_11861_msk_1.map | 421.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_11861_additional_1.map.gz emd_11861_additional_1.map.gz emd_11861_half_map_1.map.gz emd_11861_half_map_1.map.gz emd_11861_half_map_2.map.gz emd_11861_half_map_2.map.gz | 393.7 MB 337.4 MB 337.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11861 http://ftp.pdbj.org/pub/emdb/structures/EMD-11861 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11861 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11861 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11861_validation.pdf.gz emd_11861_validation.pdf.gz | 503.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11861_full_validation.pdf.gz emd_11861_full_validation.pdf.gz | 502.2 KB | 表示 | |
| XML形式データ |  emd_11861_validation.xml.gz emd_11861_validation.xml.gz | 21.5 KB | 表示 | |
| CIF形式データ |  emd_11861_validation.cif.gz emd_11861_validation.cif.gz | 26.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11861 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11861 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11861 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11861 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11861.map.gz / 形式: CCP4 / 大きさ: 81.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11861.map.gz / 形式: CCP4 / 大きさ: 81.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.837 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11861_msk_1.map emd_11861_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_11861_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_11861_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_11861_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nitrite oxidoreductase
| 全体 | 名称: Nitrite oxidoreductase |
|---|---|
| 要素 |
|
-超分子 #1: Nitrite oxidoreductase
| 超分子 | 名称: Nitrite oxidoreductase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Candidatus Kuenenia stuttgartiensis (バクテリア) Candidatus Kuenenia stuttgartiensis (バクテリア) |
| 分子量 | 理論値: 230 kDa/nm |
-分子 #1: Nitrite oxidoreductase subunit A
| 分子 | 名称: Nitrite oxidoreductase subunit A / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号: nitrate reductase |
|---|---|
| 由来(天然) | 生物種:  Candidatus Kuenenia stuttgartiensis (バクテリア) Candidatus Kuenenia stuttgartiensis (バクテリア) |
| 配列 | 文字列: MKLTRRAFLQ VAGATGATLT LAKNAMAFRL LKPAVVVDNP LDTYPDRRWE SVYRDQYQYD RTFTYCCSP NDTHACRIRA FVRNNVMMRV EQNYDHQNYS DLYGNKATRN WNPRMCLKGY T FHRRVYGP YRLRYPLIRK GWKRWADDGF PELTPENKTK YMFDNRGNDE ...文字列: MKLTRRAFLQ VAGATGATLT LAKNAMAFRL LKPAVVVDNP LDTYPDRRWE SVYRDQYQYD RTFTYCCSP NDTHACRIRA FVRNNVMMRV EQNYDHQNYS DLYGNKATRN WNPRMCLKGY T FHRRVYGP YRLRYPLIRK GWKRWADDGF PELTPENKTK YMFDNRGNDE LLRASWDEAF TY ASKGIIH ITKKYSGPEG AQKLIDQGYP KEMVDRMQGA GTRTFKGRGG MGLLGVIGKY GMY RFNNCL AIVDAHNRGV GPDQALGGRN WSNYTWHGDQ APGHPFSHGL QTSDVDMNDV RFSK LLIQT GKNLIENKMP EAHWVTEVME RGGKIVVITP EYSPSAQKAD YWIPIRNNTD TALFL GITK ILIDNKWYDA DYVKKFTDFP LLIRTDTLKR VSPKDIIPNY KLQDISDGPS YHIQGL KDE QREIIGDFVV WDAKSKGPKA ITRDDVGETL VKKGIDPVLE GSFKLKTIDG KEIEVMT LL EMYKIHLRDY DIDSVVSMTN SPKDLIERLA KDIATIKPVA IHYGEGVNHY FHATLMNR S YYLPVMLTGN VGYFGSGSHT WAGNYKAGNF QASKWSGPGF YGWVAEDVFK PNLDPYASA KDLNIKGRAL DEEVAYWNHS ERPLIVNTPK YGRKVFTGKT HMPSPTKVLW FTNVNLINNA KHVYQMLKN VNPNIEQIMS TDIEITGSIE YADFAFPANS WVEFQEFEIT NSCSNPFIQI W GKTGITPV YESKDDVKIL AGMASKLGEL LRDKRFEDNW KFAIEGRASV YINRLLDGST TM KGYTCED ILNGKYGEPG VAMLLFRTYP RHPFWEQVHE SLPFYTPTGR LQAYNDEPEI IEY GENFIV HREGPEATPY LPNAIVSTNP YIRPDDYGIP ENAEYWEDRT VRNIKKSWEE TKKT KNFLW EKGYHFYCVT PKSRHTVHSQ WAVTDWNFIW NNNFGDPYRM DKRMPGVGEH QIHIH PQAA RDLGIEDGDY VYVDANPADR PYEGWKPNDS FYKVSRLMLR AKYNPAYPYN CTMMKH SAW ISSDKTVQAH ETRPDGRALS PSGYQSSFRY GSQQSITRDW SMPMHQLDSL FHKAKIG MK FIFGFEADNH CINTVPKETL VKITKAENGG MGGKGVWDPV KTGYTAGNEN DFMKKFLN G ELIKVDA |
-分子 #2: Nitrite oxidoreductase subunit B
| 分子 | 名称: Nitrite oxidoreductase subunit B / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO / EC番号: nitrate reductase |
|---|---|
| 由来(天然) | 生物種:  Candidatus Kuenenia stuttgartiensis (バクテリア) Candidatus Kuenenia stuttgartiensis (バクテリア) |
| 配列 | 文字列: MTLVHNWHLG RRMEYPYFES RPKHQFAAVF NINRCIACQT CTMACKSTWT FNKGQEFMWW NNVETKPYG GFPQSWDVKT LKLIDSPDNI WYTDDKDKET SQYGTGAPYG TYEGDTIFEV A KKKNINQW AVGYIPEDKE WRSPNFGEDT AKSSNQPGEY STLPEHSRWF ...文字列: MTLVHNWHLG RRMEYPYFES RPKHQFAAVF NINRCIACQT CTMACKSTWT FNKGQEFMWW NNVETKPYG GFPQSWDVKT LKLIDSPDNI WYTDDKDKET SQYGTGAPYG TYEGDTIFEV A KKKNINQW AVGYIPEDKE WRSPNFGEDT AKSSNQPGEY STLPEHSRWF FYLQRICNHC TY PGCLAAC PRKAIYKRKE DGIVLIDQKR CRGYRKCVEQ CPYKKPMYRG LTRVSEKCIA CYP RIEGRD SLTDGRPMET RCMSACVGQI RLQGFLDDNP KNPITWLIRH QKIALPLYPQ FGTE PNIYY IPPRWAPRAY LRQMFGPGVD EAIEKFMVPS RELLAVMSLF RMTQTIVYEY KIEEG PKVF ETEIHGKKFT MYNDTVIGFG EDGKEVVRTT VEEPIHIRPD KHYNSI |
-分子 #3: Nitrite oxidoreductase subunit C
| 分子 | 名称: Nitrite oxidoreductase subunit C / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO / EC番号: nitrate reductase |
|---|---|
| 由来(天然) | 生物種:  Candidatus Kuenenia stuttgartiensis (バクテリア) Candidatus Kuenenia stuttgartiensis (バクテリア) |
| 配列 | 文字列: MKKFYRLLGS SSVALLGCLF LSVALCIAEE AEGVKGVAEE ELTPAKEVLN VKYMQIDVPA HITVGALEG AFKNAEGVQV KLQKQDKAFP NGGGSVNSAE IKAIHDGITI YFQVIWDDAT D NKQAIATQ EFRDGAALMF PLGKITISPE EPFSPRMGDR QKPVNLWHWK ...文字列: MKKFYRLLGS SSVALLGCLF LSVALCIAEE AEGVKGVAEE ELTPAKEVLN VKYMQIDVPA HITVGALEG AFKNAEGVQV KLQKQDKAFP NGGGSVNSAE IKAIHDGITI YFQVIWDDAT D NKQAIATQ EFRDGAALMF PLGKITISPE EPFSPRMGDR QKPVNLWHWK ADWEADLLAT GG IEECPAR YPNMHDDFST NPHSVNYHKG VIQSAAELSG GYAAHNLLSL PRGRAVEDLN AEG FGTLTS QDHQDVDGCS KFENKKWTVV FCRSLNTGDP LDVQFVPGES TYFNMAVWNG DRED RNGQK NISIQWHPLS LERIAWQ |
-分子 #4: Conserved hypothetical (Monoheme) protein
| 分子 | 名称: Conserved hypothetical (Monoheme) protein / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Candidatus Kuenenia stuttgartiensis (バクテリア) Candidatus Kuenenia stuttgartiensis (バクテリア) |
| 配列 | 文字列: MGMIKMKYVV FGVLALIMLG IGNSMFTHKA MGSSGKAAEH EGVIEPTQEL MKDIEKRLTN MLSGILENN LKYVANEAGA VVDQSYKISE FFFPFDPKKN EWFERAGIDP KDAGKITKLR E EFALFQSG IVYKALNVRK MAMEGSQEET LKAFADLIEK TCFACHKKNR DWLFDQPGSH GP GR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 70 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 30 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / 実像数: 1753 / 平均露光時間: 2.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -2.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー























 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)