+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11817 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
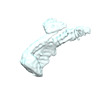
| タイトル | Barbed tail of the human TSC protein complex | |||||||||
 マップデータ マップデータ | Barbed tail of the human TSC protein complex | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 8.2 Å | |||||||||
 データ登録者 データ登録者 | Ramlaul K / Fu W / Wu G / Aylett CHS | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2021 ジャーナル: J Mol Biol / 年: 2021タイトル: Architecture of the Tuberous Sclerosis Protein Complex. 著者: Kailash Ramlaul / Wencheng Fu / Hua Li / Natàlia de Martin Garrido / Lin He / Manjari Trivedi / Wei Cui / Christopher H S Aylett / Geng Wu /   要旨: The Tuberous Sclerosis Complex (TSC) protein complex (TSCC), comprising TSC1, TSC2, and TBC1D7, is widely recognised as a key integration hub for cell growth and intracellular stress signals upstream ...The Tuberous Sclerosis Complex (TSC) protein complex (TSCC), comprising TSC1, TSC2, and TBC1D7, is widely recognised as a key integration hub for cell growth and intracellular stress signals upstream of the mammalian target of rapamycin complex 1 (mTORC1). The TSCC negatively regulates mTORC1 by acting as a GTPase-activating protein (GAP) towards the small GTPase Rheb. Both human TSC1 and TSC2 are important tumour suppressors, and mutations in them underlie the disease tuberous sclerosis. We used single-particle cryo-EM to reveal the organisation and architecture of the complete human TSCC. We show that TSCC forms an elongated scorpion-like structure, consisting of a central "body", with a "pincer" and a "tail" at the respective ends. The "body" is composed of a flexible TSC2 HEAT repeat dimer, along the surface of which runs the TSC1 coiled-coil backbone, breaking the symmetry of the dimer. Each end of the body is structurally distinct, representing the N- and C-termini of TSC1; a "pincer" is formed by the highly flexible N-terminal TSC1 core domains and a barbed "tail" makes up the TSC1 coiled-coil-TBC1D7 junction. The TSC2 GAP domain is found abutting the centre of the body on each side of the dimerisation interface, poised to bind a pair of Rheb molecules at a similar separation to the pair in activated mTORC1. Our architectural dissection reveals the mode of association and topology of the complex, casts light on the recruitment of Rheb to the TSCC, and also hints at functional higher order oligomerisation, which has previously been predicted to be important for Rheb-signalling suppression. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11817.map.gz emd_11817.map.gz | 88.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11817-v30.xml emd-11817-v30.xml emd-11817.xml emd-11817.xml | 23 KB 23 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_11817.png emd_11817.png | 42.6 KB | ||
| その他 |  emd_11817_half_map_1.map.gz emd_11817_half_map_1.map.gz emd_11817_half_map_2.map.gz emd_11817_half_map_2.map.gz | 20.5 MB 20.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11817 http://ftp.pdbj.org/pub/emdb/structures/EMD-11817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11817 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11817_validation.pdf.gz emd_11817_validation.pdf.gz | 256.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11817_full_validation.pdf.gz emd_11817_full_validation.pdf.gz | 255.2 KB | 表示 | |
| XML形式データ |  emd_11817_validation.xml.gz emd_11817_validation.xml.gz | 10.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11817 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11817 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11817 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11817 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11817.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11817.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Barbed tail of the human TSC protein complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map 2
| ファイル | emd_11817_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
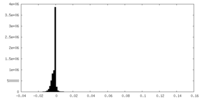
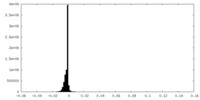
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_11817_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
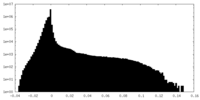
| 投影像・断面図 |
| ||||||||||||
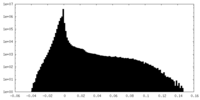
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Barbed tail of the human TSC protein complex
| 全体 | 名称: Barbed tail of the human TSC protein complex |
|---|---|
| 要素 |
|
-超分子 #1: Barbed tail of the human TSC protein complex
| 超分子 | 名称: Barbed tail of the human TSC protein complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 実験値: 700 KDa |
-分子 #1: Hamartin
| 分子 | 名称: Hamartin / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAQQANVGEL LAMLDSPMLG VRDDVTAVFK ENLNSDRGPM LVNTLVDYYL ETSSQPALHI LTTLQEPHD KHLLDRINEY VGKAATRLSI LSLLGHVIRL QPSWKHKLSQ APLLPSLLKC L KMDTDVVV LTTGVLVLIT MLPMIPQSGK QHLLDFFDIF GRLSSWCLKK ...文字列: MAQQANVGEL LAMLDSPMLG VRDDVTAVFK ENLNSDRGPM LVNTLVDYYL ETSSQPALHI LTTLQEPHD KHLLDRINEY VGKAATRLSI LSLLGHVIRL QPSWKHKLSQ APLLPSLLKC L KMDTDVVV LTTGVLVLIT MLPMIPQSGK QHLLDFFDIF GRLSSWCLKK PGHVAEVYLV HL HASVYAL FHRLYGMYPC NFVSFLRSHY SMKENLETFE EVVKPMMEHV RIHPELVTGS KDH ELDPRR WKRLETHDVV IECAKISLDP TEASYEDGYS VSHQISARFP HRSADVTTSP YADT QNSYG CATSTPYSTS RLMLLNMPGQ LPQTLSSPST RLITEPPQAT LWSPSMVCGM TTPPT SPGN VPPDLSHPYS KVFGTTAGGK GTPLGTPATS PPPAPLCHSD DYVHISLPQA TVTPPR KEE RMDSARPCLH RQHHLLNDRG SEEPPGSKGS VTLSDLPGFL GDLASEEDSI EKDKEEA AI SRELSEITTA EAEPVVPRGG FDSPFYRDSL PGSQRKTHSA ASSSQGASVN PEPLHSSL D KLGPDTPKQA FTPIDLPCGS ADESPAGDRE CQTSLETSIF TPSPCKIPPP TRVGFGSGQ PPPYDHLFEV ALPKTAHHFV IRKTEELLKK AKGNTEEDGV PSTSPMEVLD RLIQQGADAH SKELNKLPL PSKSVDWTHF GGSPPSDEIR TLRDQLLLLH NQLLYERFKR QQHALRNRRL L RKVIKAAA LEEHNAAMKD QLKLQEKDIQ MWKVSLQKEQ ARYNQLQEQR DTMVTKLHSQ IR QLQHDRE EFYNQSQELQ TKLEDCRNMI AELRIELKKA NNKVCHTELL LSQVSQKLSN SES VQQQME FLNRQLLVLG EVNELYLEQL QNKHSDTTKE VEMMKAAYRK ELEKNRSHVL QQTQ RLDTS QKRILELESH LAKKDHLLLE QKKYLEDVKL QARGQLQAAE SRYEAQKRIT QVFEL EILD LYGRLEKDGL LKKLEEEKAE AAEAAEERLD CCNDGCSDSM VGHNEEASGH NGETKT PRP SSARGSSGSR GGGGSSSSSS ELSTPEKPPH QRAGPFSSRW ETTMGEASAS IPTTVGS LP SSKSFLGMKA RELFRNKSES QCDEDGMTSS LSESLKTELG KDLGVEAKIP LNLDGPHP S PPTPDSVGQL HIMDYNETHH EHS |
-分子 #2: Tuberin
| 分子 | 名称: Tuberin / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAKPTSKDSG LKEKFKILLG LGTPRPNPRS AEGKQTEFII TAEILRELSM ECGLNNRIRM IGQICEVAK TKKFEEHAVE ALWKAVADLL QPERPLEARH AVLALLKAIV QGQGERLGVL R ALFFKVIK DYPSNEDLHE RLEVFKALTD NGRHITYLEE ELADFVLQWM ...文字列: MAKPTSKDSG LKEKFKILLG LGTPRPNPRS AEGKQTEFII TAEILRELSM ECGLNNRIRM IGQICEVAK TKKFEEHAVE ALWKAVADLL QPERPLEARH AVLALLKAIV QGQGERLGVL R ALFFKVIK DYPSNEDLHE RLEVFKALTD NGRHITYLEE ELADFVLQWM DVGLSSEFLL VL VNLVKFN SCYLDEYIAR MVQMICLLCV RTASSVDIEV SLQVLDAVVC YNCLPAESLP LFI VTLCRT INVKELCEPC WKLMRNLLGT HLGHSAIYNM CHLMEDRAYM EDAPLLRGAV FFVG MALWG AHRLYSLRNS PTSVLPSFYQ AMACPNEVVS YEIVLSITRL IKKYRKELQV VAWDI LLNI IERLLQQLQT LDSPELRTIV HDLLTTVEEL CDQNEFHGSQ ERYFELVERC ADQRPE SSL LNLISYRAQS IHPAKDGWIQ NLQALMERFF RSESRGAVRI KVLDVLSFVL LINRQFY EE ELINSVVISQ LSHIPEDKDH QVRKLATQLL VDLAEGCHTH HFNSLLDIIE KVMARSLS P PPELEERDVA AYSASLEDVK TAVLGLLVIL QTKLYTLPAS HATRVYEMLV SHIQLHYKH SYTLPIASSI RLQAFDFLLL LRADSLHRLG LPNKDGVVRF SPYCVCDYME PERGSEKKTS GPLSPPTGP PGPAPAGPAV RLGSVPYSLL FRVLLQCLKQ ESDWKVLKLV LGRLPESLRY K VLIFTSPC SVDQLCSALC SMLSGPKTLE RLRGAPEGFS RTDLHLAVVP VLTALISYHN YL DKTKQRE MVYCLEQGLI HRCASQCVVA LSICSVEMPD IIIKALPVLV VKLTHISATA SMA VPLLEF LSTLARLPHL YRNFAAEQYA SVFAISLPYT NPSKFNQYIV CLAHHVIAMW FIRC RLPFR KDFVPFITKG LRSNVLLSFD DTPEKDSFRA RSTSLNERPK SLRIARPPKQ GLNNS PPVK EFKESSAAEA FRCRSISVSE HVVRSRIQTS LTSASLGSAD ENSVAQADDS LKNLHL ELT ETCLDMMARY VFSNFTAVPK RSPVGEFLLA GGRTKTWLVG NKLVTVTTSV GTGTRSL LG LDSGELQSGP ESSSSPGVHV RQTKEAPAKL ESQAGQQVSR GARDRVRSMS GGHGLRVG A LDVPASQFLG SATSPGPRTA PAAKPEKASA GTRVPVQEKT NLAAYVPLLT QGWAEILVR RPTGNTSWLM SLENPLSPFS SDINNMPLQE LSNALMAAER FKEHRDTALY KSLSVPAAST AKPPPLPRS NTVASFSSLY QSSCQGQLHR SVSWADSAVV MEEGSPGEVP VLVEPPGLED V EAALGMDR RTDAYSRSSS VSSQEEKSLH AEELVGRGIP IERVVSSEGG RPSVDLSFQP SQ PLSKSSS SPELQTLQDI LGDPGDKADV GRLSPEVKAR SQSGTLDGES AAWSASGEDS RGQ PEGPLP SSSPRSPSGL RPRGYTISDS APSRRGKRVE RDALKSRATA SNAEKVPGIN PSFV FLQLY HSPFFGDESN KPILLPNESQ SFERSVQLLD QIPSYDTHKI AVLYVGEGQS NSELA ILSN EHGSYRYTEF LTGLGRLIEL KDCQPDKVYL GGLDVCGEDG QFTYCWHDDI MQAVFH IAT LMPTKDVDKH RCDKKRHLGN DFVSIVYNDS GEDFKLGTIK GQFNFVHVIV TPLDYEC NL VSLQCRKDME GLVDTSVAKI VSDRNLPFVA RQMALHANMA SQVHHSRSNP TDIYPSKW I ARLRHIKRLR QRICEEAAYS NPSLPLVHPP SHSKAPAQTP AEPTPGYEVG QRKRLISSV EDFTEFV |
-分子 #3: TBC1D7
| 分子 | 名称: TBC1D7 / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MTEDSQRNFR SVYYEKVGFR GVEEKKSLEI LLKDDRLDTE KLCTFSQRFP LPSMYRALVW KVLLGILPP HHESHAKVMM YRKEQYLDVL HALKVVRFVS DATPQAEVYL RMYQLESGKL P RSPSFPLE PDDEVFLAIA KAMEEMVEDS VDCYWITRRF VNQLNTKYRD ...文字列: MTEDSQRNFR SVYYEKVGFR GVEEKKSLEI LLKDDRLDTE KLCTFSQRFP LPSMYRALVW KVLLGILPP HHESHAKVMM YRKEQYLDVL HALKVVRFVS DATPQAEVYL RMYQLESGKL P RSPSFPLE PDDEVFLAIA KAMEEMVEDS VDCYWITRRF VNQLNTKYRD SLPQLPKAFE QY LNLEDGR LLTHLRMCSA APKLPYDLWF KRCFAGCLPE SSLQRVWDKV VSGSCKILVF VAV EILLTF KIKVMALNSA EKITKFLENI PQDSSDAIVS KAIDLWHKHC GTPVHSS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 詳細: 25 mM K-HEPES, pH 7.6, 175 mM KCl or 150 mM LiCl, 1 mM TCEP, and 0.5 uM EDTA |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - #0 - Film type ID: 1 / 支持フィルム - #0 - 材質: CARBON / 支持フィルム - #0 - トポロジー: HOLEY ARRAY / 支持フィルム - #1 - Film type ID: 2 / 支持フィルム - #1 - 材質: GRAPHENE OXIDE / 支持フィルム - #1 - トポロジー: CONTINUOUS / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 96 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV / 詳細: 2s blot. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 70 eV |
| 詳細 | Data collected at eBIC |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 2 / 実像数: 5387 / 平均電子線量: 52.3 e/Å2 / 詳細: Dose weighting |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 37000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)