+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1151 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron cryotomography of the E. coli pyruvate and 2-oxoglutarate dehydrogenase complexes. | |||||||||
 マップデータ マップデータ | Volume of an extracted E. coli pyruvate dehydrogenase multienzyme complex particle from a dual-tilt tomogram. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Pyruvate dehydrogenase E1 component / Dihydrolipoamide acetyltransferase pyruvate dehydrogenase complex / Dihydrolipoamide dehydrogenase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 55.0 Å | |||||||||
 データ登録者 データ登録者 | Murphy GE / Jensen GJ | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2005 ジャーナル: Structure / 年: 2005タイトル: Electron cryotomography of the E. coli pyruvate and 2-oxoglutarate dehydrogenase complexes. 著者: Gavin E Murphy / Grant J Jensen /  要旨: The E. coli pyruvate and 2-oxoglutarate dehydrogenases are two closely related, large complexes that exemplify a growing number of multiprotein "machines" whose domains have been studied extensively ...The E. coli pyruvate and 2-oxoglutarate dehydrogenases are two closely related, large complexes that exemplify a growing number of multiprotein "machines" whose domains have been studied extensively and modeled in atomic detail, but whose quaternary structures have remained unclear for lack of an effective imaging technology. Here, electron cryotomography was used to show that the E1 and E3 subunits of these complexes are flexibly tethered approximately 11 nm away from the E2 core. This result demonstrates unambiguously that electron cryotomography can reveal the relative positions of features as small as 80 kDa in individual complexes, elucidating quaternary structure and conformational flexibility. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1151.map.gz emd_1151.map.gz | 224.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1151-v30.xml emd-1151-v30.xml emd-1151.xml emd-1151.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1151.gif 1151.gif | 21.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1151 http://ftp.pdbj.org/pub/emdb/structures/EMD-1151 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1151 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1151 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1151_validation.pdf.gz emd_1151_validation.pdf.gz | 246.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1151_full_validation.pdf.gz emd_1151_full_validation.pdf.gz | 245.7 KB | 表示 | |
| XML形式データ |  emd_1151_validation.xml.gz emd_1151_validation.xml.gz | 4.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1151 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1151 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1151 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1151 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1151.map.gz / 形式: CCP4 / 大きさ: 489.3 KB / タイプ: IMAGE STORED AS SIGNED BYTE ダウンロード / ファイル: emd_1151.map.gz / 形式: CCP4 / 大きさ: 489.3 KB / タイプ: IMAGE STORED AS SIGNED BYTE | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Volume of an extracted E. coli pyruvate dehydrogenase multienzyme complex particle from a dual-tilt tomogram. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 8.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
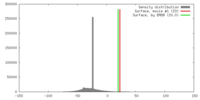
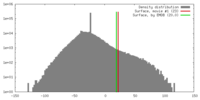
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Unengineered E. coli Pyruvate Dehydrogenase Multienzyme Complex
| 全体 | 名称: Unengineered E. coli Pyruvate Dehydrogenase Multienzyme Complex |
|---|---|
| 要素 |
|
-超分子 #1000: Unengineered E. coli Pyruvate Dehydrogenase Multienzyme Complex
| 超分子 | 名称: Unengineered E. coli Pyruvate Dehydrogenase Multienzyme Complex タイプ: sample / ID: 1000 詳細: The sample was thawed from storage at -80 degrees Celcius before being loaded onto the grid. 集合状態: Up to 24 pyruvate dehydrogenase E1p and dihydrolipoamide dehydrogenase E3 dimers together bind to the 24 dihydrolipoamide acetyltransferase E2p octahedral core. Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 5.6 MDa |
-分子 #1: E2p octahedral core
| 分子 | 名称: E2p octahedral core / タイプ: protein_or_peptide / ID: 1 / Name.synonym: dihydrolipoamide acetyltransferase 詳細: 24 arranged as cube; See Wagenknecht, T. et al., JSB 109:70-77 (1992). コピー数: 24 / 集合状態: 24mer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 1.6 MDa |
| 組換発現 | 生物種:  |
| 配列 | InterPro: Dihydrolipoamide acetyltransferase pyruvate dehydrogenase complex |
-分子 #2: E1p
| 分子 | 名称: E1p / タイプ: protein_or_peptide / ID: 2 / Name.synonym: pyruvate dehydrogenase 詳細: Up to 24 of these; See Wagenknecht, T. et al., JSB 109:70-77 (1992). コピー数: 2 / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 200 KDa |
| 組換発現 | 生物種:  |
| 配列 | InterPro: Pyruvate dehydrogenase E1 component |
-分子 #3: E3
| 分子 | 名称: E3 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: dihydrolipoamide dehydrogenase 詳細: Up to 24 of these; See Wagenknecht, T. et al., JSB 109:70-77 (1992). コピー数: 2 / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 100 KDa |
| 組換発現 | 生物種:  |
| 配列 | InterPro: Dihydrolipoamide dehydrogenase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7 / 詳細: 20 mM Potassium Phosphate |
| グリッド | 詳細: R 1.5/1.3 Quantifoil |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: OTHER 詳細: Vitrification instrument: Vitrobot. Sample was at 22 C in air before plunging. 手法: Blot for 3.5 seconds with an offset of -3 before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 温度 | 最低: 82 K / 最高: 82 K / 平均: 82 K |
| アライメント法 | Legacy - Electron beam tilt params: 0 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF 3000 エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 日付 | 2004年8月24日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 1000 (2k x 2k) 平均電子線量: 110 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 36600 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 10.0 µm / 最小 デフォーカス(公称値): 10.0 µm / 倍率(公称値): 27500 |
| 試料ステージ | 試料ホルダー: FEI Polara / 試料ホルダーモデル: GATAN HELIUM / Tilt series - Axis1 - Min angle: -63 ° / Tilt series - Axis1 - Max angle: 66 ° |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Dual-axis tilt series, with 44 sections on one axis, and 45 on the other. Average number of tilts used in the 3D reconstructions: 89. Average tomographic tilt angle increment: 3. |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 55.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称:  IMOD IMOD詳細: The individual, orthogonal tomograms were filtered at their first CTF zero and then merged. |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)