+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11287 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human mitochondrial ribosome in complex with mRNA, A/A tRNA and P/P tRNA, local-masked aligned on SSU head. | |||||||||
 マップデータ マップデータ | Sharpened, local-resolution filtered and masked. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
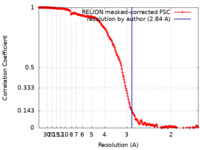
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.84 Å | |||||||||
 データ登録者 データ登録者 | Andrell A / Itoh Y / Amunts A | |||||||||
| 資金援助 |  スウェーデン, 1件 スウェーデン, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2021 ジャーナル: Science / 年: 2021タイトル: Mechanism of membrane-tethered mitochondrial protein synthesis. 著者: Yuzuru Itoh / Juni Andréll / Austin Choi / Uwe Richter / Priyanka Maiti / Robert B Best / Antoni Barrientos / Brendan J Battersby / Alexey Amunts /     要旨: Mitochondrial ribosomes (mitoribosomes) are tethered to the mitochondrial inner membrane to facilitate the cotranslational membrane insertion of the synthesized proteins. We report cryo-electron ...Mitochondrial ribosomes (mitoribosomes) are tethered to the mitochondrial inner membrane to facilitate the cotranslational membrane insertion of the synthesized proteins. We report cryo-electron microscopy structures of human mitoribosomes with nascent polypeptide, bound to the insertase oxidase assembly 1-like (OXA1L) through three distinct contact sites. OXA1L binding is correlated with a series of conformational changes in the mitoribosomal large subunit that catalyze the delivery of newly synthesized polypeptides. The mechanism relies on the folding of mL45 inside the exit tunnel, forming two specific constriction sites that would limit helix formation of the nascent chain. A gap is formed between the exit and the membrane, making the newly synthesized proteins accessible. Our data elucidate the basis by which mitoribosomes interact with the OXA1L insertase to couple protein synthesis and membrane delivery. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11287.map.gz emd_11287.map.gz | 14.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11287-v30.xml emd-11287-v30.xml emd-11287.xml emd-11287.xml | 37.7 KB 37.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11287_fsc.xml emd_11287_fsc.xml | 19.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11287.png emd_11287.png | 72.4 KB | ||
| マスクデータ |  emd_11287_msk_1.map emd_11287_msk_1.map | 600.7 MB |  マスクマップ マスクマップ | |
| その他 |  emd_11287_half_map_1.map.gz emd_11287_half_map_1.map.gz emd_11287_half_map_2.map.gz emd_11287_half_map_2.map.gz | 486.3 MB 486.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11287 http://ftp.pdbj.org/pub/emdb/structures/EMD-11287 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11287 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11287 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11287_validation.pdf.gz emd_11287_validation.pdf.gz | 399.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11287_full_validation.pdf.gz emd_11287_full_validation.pdf.gz | 398.5 KB | 表示 | |
| XML形式データ |  emd_11287_validation.xml.gz emd_11287_validation.xml.gz | 496 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11287 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11287 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11287 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11287 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11287.map.gz / 形式: CCP4 / 大きさ: 39.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11287.map.gz / 形式: CCP4 / 大きさ: 39.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened, local-resolution filtered and masked. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11287_msk_1.map emd_11287_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_11287_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_11287_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ribosome with mRNA and tRNAs
| 全体 | 名称: Ribosome with mRNA and tRNAs |
|---|---|
| 要素 |
|
-超分子 #1: Ribosome with mRNA and tRNAs
| 超分子 | 名称: Ribosome with mRNA and tRNAs / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#89 詳細: Mitochondrial ribosome in complex with mRNA, A/A tRNA and P/P tRNA |
|---|---|
| 分子量 | 理論値: 3 MDa |
-超分子 #2: SSU body
| 超分子 | 名称: SSU body / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#53 / 詳細: The body part of the small subunit of ribosome |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 3.0 nm / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-20 / 撮影したグリッド数: 2 / 実像数: 19655 / 平均露光時間: 4.0 sec. / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.6 µm / 最小 デフォーカス(補正後): 0.2 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)