+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1120 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Conservation of the capsid structure in tailed dsDNA bacteriophages: the pseudoatomic structure of phi29. | |||||||||
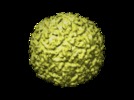
 マップデータ マップデータ | This map is a 3D cryo-EM reconstruction of a fiberless, isometric variant of bacteriophage phi29. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Bacillus phage phi29 (ファージ) Bacillus phage phi29 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.9 Å | |||||||||
 データ登録者 データ登録者 | Morais MC / Choi KH / Koti JS / Chipman PR / Anderson DL / Rossmann MG | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2005 ジャーナル: Mol Cell / 年: 2005タイトル: Conservation of the capsid structure in tailed dsDNA bacteriophages: the pseudoatomic structure of phi29. 著者: Marc C Morais / Kyung H Choi / Jaya S Koti / Paul R Chipman / Dwight L Anderson / Michael G Rossmann /  要旨: Bacteriophage phi29 is one of the smallest and simplest known dsDNA phages, making it amenable to structural investigations. The three-dimensional structure of a fiberless, isometric variant has been ...Bacteriophage phi29 is one of the smallest and simplest known dsDNA phages, making it amenable to structural investigations. The three-dimensional structure of a fiberless, isometric variant has been determined to 7.9 A resolution by cryo-electron microscopy (cryo-EM), allowing the identification of alpha helices and beta sheets. Their arrangement indicates that the folds of the phi29 and bacteriophage HK97 capsid proteins are similar except for an additional immunoglobulin-like domain of the phi29 protein. An atomic model that incorporates these two domains fits well into the cryo-EM density of the T = 3, fiberless isometric phi29 particle, and cryo-EM structures of fibered isometric and fiberless prolate prohead phi29 particles at resolutions of 8.7 A and 12.7 A, respectively. Thus, phi29 joins the growing number of phages that utilize the HK97 capsid structure, suggesting that this protein fold may be as prevalent in capsids of dsDNA phages as the jelly roll fold is in eukaryotic viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1120.map.gz emd_1120.map.gz | 4.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1120-v30.xml emd-1120-v30.xml emd-1120.xml emd-1120.xml | 10.1 KB 10.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1120.gif 1120.gif | 21 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1120 http://ftp.pdbj.org/pub/emdb/structures/EMD-1120 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1120 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1120 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1120_validation.pdf.gz emd_1120_validation.pdf.gz | 404.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1120_full_validation.pdf.gz emd_1120_full_validation.pdf.gz | 404.4 KB | 表示 | |
| XML形式データ |  emd_1120_validation.xml.gz emd_1120_validation.xml.gz | 5.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1120 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1120 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1120 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1120 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1120.map.gz / 形式: CCP4 / 大きさ: 82.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1120.map.gz / 形式: CCP4 / 大きさ: 82.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This map is a 3D cryo-EM reconstruction of a fiberless, isometric variant of bacteriophage phi29. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.84 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : phi29 fiberless isometric particle
| 全体 | 名称: phi29 fiberless isometric particle |
|---|---|
| 要素 |
|
-超分子 #1000: phi29 fiberless isometric particle
| 超分子 | 名称: phi29 fiberless isometric particle / タイプ: sample / ID: 1000 / 集合状態: particle forms an T3 icosahedron / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 8.97 MDa |
-超分子 #1: Bacillus phage phi29
| 超分子 | 名称: Bacillus phage phi29 / タイプ: virus / ID: 1 / Name.synonym: phi29 / NCBI-ID: 10756 / 生物種: Bacillus phage phi29 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes / Syn species name: phi29 |
|---|---|
| 宿主 | 生物種:  |
| 分子量 | 実験値: 8.97 MDa |
| ウイルス殻 | Shell ID: 1 / 直径: 425 Å / T番号(三角分割数): 3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 詳細: 25 mM Tris-HCl 5 mM MgCl2 50 mM NaCl 5 mM sodium azide |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 温度 | 平均: 90.0 K |
| アライメント法 | Legacy - Electron beam tilt params: 0 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 15 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.7 µm / 倍率(公称値): 38000 |
| 試料ステージ | 試料ホルダー: Side entry liquid nitrogen-cooled cryo specimen holder 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt angle min: 0 / Tilt angle max: 0 |
- 画像解析
画像解析
| CTF補正 | 詳細: CTF correction included phase and amplitude correction for whole micrographs |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 7.9 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMPFT, POR, P3DR / 使用した粒子像数: 5922 |
-原子モデル構築 1
| ソフトウェア | 名称: SITUS and EMFIT |
|---|---|
| 詳細 | Protocol: Rigid Body. The fitted molecule consists of two domains, an HK97-like domain and a bacterial immunoglobulin group 2-like domain (BIG2). The two domains were fitted separately. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: correlation of laplacian filtered data for SITUS, and for EMFIT, target maximizes positive density around an atom, minimizes negative density, and minimizes clashes ...当てはまり具合の基準: correlation of laplacian filtered data for SITUS, and for EMFIT, target maximizes positive density around an atom, minimizes negative density, and minimizes clashes with neighboring olecules |
| 得られたモデル |  PDB-1yxn: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)