+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10889 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Vip3Bc1 tetramer in processed, activated state | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Toxin / Vip3 / Bt toxin | |||||||||
| 機能・相同性 | Vegetative insecticide protein 3 / Vegetative insecticide protein 3A N terminal / Vegetative insecticidal protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
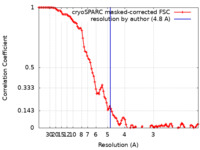
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.8 Å | |||||||||
 データ登録者 データ登録者 | Thompson RF / Byrne MJ | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Cryo-EM structures of an insecticidal Bt toxin reveal its mechanism of action on the membrane. 著者: Matthew J Byrne / Matthew G Iadanza / Marcos Arribas Perez / Daniel P Maskell / Rachel M George / Emma L Hesketh / Paul A Beales / Marc D Zack / Colin Berry / Rebecca F Thompson /   要旨: Insect pests are a major cause of crop losses worldwide, with an estimated economic cost of $470 billion annually. Biotechnological tools have been introduced to control such insects without the need ...Insect pests are a major cause of crop losses worldwide, with an estimated economic cost of $470 billion annually. Biotechnological tools have been introduced to control such insects without the need for chemical pesticides; for instance, the development of transgenic plants harbouring genes encoding insecticidal proteins. The Vip3 (vegetative insecticidal protein 3) family proteins from Bacillus thuringiensis convey toxicity to species within the Lepidoptera, and have wide potential applications in commercial agriculture. Vip3 proteins are proposed to exert their insecticidal activity through pore formation, though to date there is no mechanistic description of how this occurs on the membrane. Here we present cryo-EM structures of a Vip3 family toxin in both inactive and activated forms in conjunction with structural and functional data on toxin-membrane interactions. Together these data demonstrate that activated Vip3Bc1 complex is able to insert into membranes in a highly efficient manner, indicating that receptor binding is the likely driver of Vip3 specificity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10889.map.gz emd_10889.map.gz | 96.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10889-v30.xml emd-10889-v30.xml emd-10889.xml emd-10889.xml | 10.7 KB 10.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10889_fsc.xml emd_10889_fsc.xml | 11.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10889.png emd_10889.png | 131.5 KB | ||
| Filedesc metadata |  emd-10889.cif.gz emd-10889.cif.gz | 5.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10889 http://ftp.pdbj.org/pub/emdb/structures/EMD-10889 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10889 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10889 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10889_validation.pdf.gz emd_10889_validation.pdf.gz | 189.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10889_full_validation.pdf.gz emd_10889_full_validation.pdf.gz | 189.5 KB | 表示 | |
| XML形式データ |  emd_10889_validation.xml.gz emd_10889_validation.xml.gz | 503 B | 表示 | |
| CIF形式データ |  emd_10889_validation.cif.gz emd_10889_validation.cif.gz | 373 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10889 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10889 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10889 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10889 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10889.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10889.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.065 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Tetrameric assembly of activated, processed Vip3Bc1
| 全体 | 名称: Tetrameric assembly of activated, processed Vip3Bc1 |
|---|---|
| 要素 |
|
-超分子 #1: Tetrameric assembly of activated, processed Vip3Bc1
| 超分子 | 名称: Tetrameric assembly of activated, processed Vip3Bc1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Vegetative insecticidal protein
| 分子 | 名称: Vegetative insecticidal protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 91.287148 KDa |
| 組換発現 | 生物種:  Pseudomonas fluorescens (蛍光菌) Pseudomonas fluorescens (蛍光菌) |
| 配列 | 文字列: MVQKWMQRMI IVDNNKLNVR ALPSFIDYFN GIYGFATGIK DIMGMIFKTD TGGSNLTLDE ILKNQNLLND ISGKLDGING DLGDLIAQG NLNSELAKEL LKISNEQNQM LNHVNAQLNA INSTLNIYLP KITSMLNEVM KQNHVLSLQI EFLSKQLQEI S DKLDIINL ...文字列: MVQKWMQRMI IVDNNKLNVR ALPSFIDYFN GIYGFATGIK DIMGMIFKTD TGGSNLTLDE ILKNQNLLND ISGKLDGING DLGDLIAQG NLNSELAKEL LKISNEQNQM LNHVNAQLNA INSTLNIYLP KITSMLNEVM KQNHVLSLQI EFLSKQLQEI S DKLDIINL NVLINSTLTE ITPAYQRIKY VNEKFDELTS TVEKNPKSYQ DNVTKEVIEN LNELTELAKS VTKNDMDSFE FY LQTFHDV MTGNNLFGRS ALKTASELIT KENVTTRGSE IGKVYNFLIV LTSLQAKAFL TLTACRKLLG LTDIDYTQIM NHH IDGQKR EFRINILPTL SNNFSNPSYS KNRGSDIDDP IVVLEAAPGY ALIGFEILND PLPILKGYQA RLKPNYQVDR ESMS ETIYG DIHKLFCPKQ LEQKYYIKDI EFPEGYVITK IVFEKRLNQL GYEVTANFYD PSTGSIDLNK VKVESWKEKS CEEDS CEDE YSIIKAETDG IYMPLGVVSE TFLTPIYGFG LTVDEKNQKI TLTGKSYLRE SLLETDLLNN ETYLIASPDG YISSIV ENW NITSDNTGSW RANNNNAFVD KADTIKGSSS LYTHKDGEFS QFIGNKLKPK TNYVIQYVIK GRPAIYLKNN KDTLFED TK NNFSDFQTVT KKFNSGVNPS EIYFLFKNQS EYEAWGNNFI ILEIKSLEFL PQMLKPEDWI PSGNVQMKDG GRLEILGD G YFKQFIKLEN DSTYHLRLSV KGTGRVSIID ESKYLLFVNV KDEDLTRVIK NTSSKGECFI ALEGTYVENS STIFSNVSI VKE UniProtKB: Vegetative insecticidal protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: FEI FALCON III (4k x 4k) #0 - 検出モード: INTEGRATING / #0 - 撮影したグリッド数: 1 / #0 - 平均露光時間: 1.5 sec. / #0 - 平均電子線量: 70.8 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: FEI FALCON III (4k x 4k) #1 - 検出モード: INTEGRATING / #1 - 平均電子線量: 42.51 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)