+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10794 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Photorhabdus luminescens TcdA1 in complex with BSA-Lewis X | |||||||||
 マップデータ マップデータ | The main map of Pl-TcdA1 with BSA-LewisX after postprocessing | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Photorhabdus luminescens (バクテリア) / Photorhabdus luminescens (バクテリア) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.0 Å | |||||||||
 データ登録者 データ登録者 | Roderer D / Broecker F / Sitsel O / Kaplonek P / Leidreiter F / Seeberger PH / Raunser S | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Glycan-dependent cell adhesion mechanism of Tc toxins. 著者: Daniel Roderer / Felix Bröcker / Oleg Sitsel / Paulina Kaplonek / Franziska Leidreiter / Peter H Seeberger / Stefan Raunser /   要旨: Toxin complex (Tc) toxins are virulence factors of pathogenic bacteria. Tcs are composed of three subunits: TcA, TcB and TcC. TcA facilitates receptor-toxin interaction and membrane permeation, TcB ...Toxin complex (Tc) toxins are virulence factors of pathogenic bacteria. Tcs are composed of three subunits: TcA, TcB and TcC. TcA facilitates receptor-toxin interaction and membrane permeation, TcB and TcC form a toxin-encapsulating cocoon. While the mechanisms of holotoxin assembly and pore formation have been described, little is known about receptor binding of TcAs. Here, we identify heparins/heparan sulfates and Lewis antigens as receptors for different TcAs from insect and human pathogens. Glycan array screening reveals that all tested TcAs bind negatively charged heparins. Cryo-EM structures of Morganella morganii TcdA4 and Xenorhabdus nematophila XptA1 reveal that heparins/heparan sulfates unexpectedly bind to different regions of the shell domain, including receptor-binding domains. In addition, Photorhabdus luminescens TcdA1 binds to Lewis antigens with micromolar affinity. Here, the glycan interacts with the receptor-binding domain D of the toxin. Our results suggest a glycan dependent association mechanism of Tc toxins on the host cell surface. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10794.map.gz emd_10794.map.gz | 15.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10794-v30.xml emd-10794-v30.xml emd-10794.xml emd-10794.xml | 16.4 KB 16.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10794.png emd_10794.png | 164.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10794 http://ftp.pdbj.org/pub/emdb/structures/EMD-10794 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10794 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10794 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10794_validation.pdf.gz emd_10794_validation.pdf.gz | 217.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10794_full_validation.pdf.gz emd_10794_full_validation.pdf.gz | 217.1 KB | 表示 | |
| XML形式データ |  emd_10794_validation.xml.gz emd_10794_validation.xml.gz | 7.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10794 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10794 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10794 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10794 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10794.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10794.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The main map of Pl-TcdA1 with BSA-LewisX after postprocessing | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
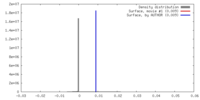
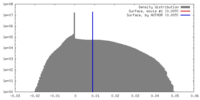
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Photorhabdus luminescens TcdA1 pentamer with one BSA-Lewis X, cro...
| 全体 | 名称: Photorhabdus luminescens TcdA1 pentamer with one BSA-Lewis X, crosslinked with glutaraldehyde |
|---|---|
| 要素 |
|
-超分子 #1: Photorhabdus luminescens TcdA1 pentamer with one BSA-Lewis X, cro...
| 超分子 | 名称: Photorhabdus luminescens TcdA1 pentamer with one BSA-Lewis X, crosslinked with glutaraldehyde タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 1.5 MDa |
-超分子 #2: Photorhabdus luminescens toxin complex subunit TcdA1, Homopentamer
| 超分子 | 名称: Photorhabdus luminescens toxin complex subunit TcdA1, Homopentamer タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Photorhabdus luminescens (バクテリア) Photorhabdus luminescens (バクテリア) |
| 組換発現 | 生物種:  |
-超分子 #3: BSA with Lewis X glycans crosslinked to surface lysines
| 超分子 | 名称: BSA with Lewis X glycans crosslinked to surface lysines タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Photorhabdus luminescens toxin complex subunit TcdA1, Homopentamer
| 分子 | 名称: Photorhabdus luminescens toxin complex subunit TcdA1, Homopentamer タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Photorhabdus luminescens (バクテリア) Photorhabdus luminescens (バクテリア) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNESVKEIPD VLKSQCGFNC LTDISHSSFN EFRQQVSEHL SWSETHDLYH DAQQAQKDNR LYEARILKR ANPQLQNAVH LAILAPNAEL IGYNNQFSGR ASQYVAPGTV SSMFSPAAYL T ELYREARN LHASDSVYYL DTRRPDLKSM ALSQQNMDIE LSTLSLSNEL ...文字列: MNESVKEIPD VLKSQCGFNC LTDISHSSFN EFRQQVSEHL SWSETHDLYH DAQQAQKDNR LYEARILKR ANPQLQNAVH LAILAPNAEL IGYNNQFSGR ASQYVAPGTV SSMFSPAAYL T ELYREARN LHASDSVYYL DTRRPDLKSM ALSQQNMDIE LSTLSLSNEL LLESIKTESK LE NYTKVME MLSTFRPSGA TPYHDAYENV REVIQLQDPG LEQLNASPAI AGLMHQASLL GIN ASISPE LFNILTEEIT EGNAEELYKK NFGNIEPASL AMPEYLKRYY NLSDEELSQF IGKA SNFGQ QEYSNNQLIT PVVNSSDGTV KVYRITREYT TNAYQMDVEL FPFGGENYRL DYKFK NFYN ASYLSIKLND KRELVRTEGA PQVNIEYSAN ITLNTADISQ PFEIGLTRVL PSGSWA YAA AKFTVEEYNQ YSFLLKLNKA IRLSRATELS PTILEGIVRS VNLQLDINTD VLGKVFL TK YYMQRYAIHA ETALILCNAP ISQRSYDNQP SQFDRLFNTP LLNGQYFSTG DEEIDLNS G STGDWRKTIL KRAFNIDDVS LFRLLKITDH DNKDGKIKNN LKNLSNLYIG KLLADIHQL TIDELDLLLI AVGEGKTNLS AISDKQLATL IRKLNTITSW LHTQKWSVFQ LFIMTSTSYN KTLTPEIKN LLDTVYHGLQ GFDKDKADLL HVMAPYIAAT LQLSSENVAH SVLLWADKLQ P GDGAMTAE KFWDWLNTKY TPGSSEAVET QEHIVQYCQA LAQLEMVYHS TGINENAFRL FV TKPEMFG AATGAAPAHD ALSLIMLTRF ADWVNALGEK ASSVLAAFEA NSLTAEQLAD AMN LDANLL LQASIQAQNH QHLPPVTPEN AFSCWTSINT ILQWVNVAQQ LNVAPQGVSA LVGL DYIQS MKETPTYAQW ENAAGVLTAG LNSQQANTLH AFLDESRSAA LSTYYIRQVA KAAAA IKSR DDLYQYLLID NQVSAAIKTT RIAEAIASIQ LYVNRALENV EENANSGVIS RQFFID WDK YNKRYSTWAG VSQLVYYPEN YIDPTMRIGQ TKMMDALLQS VSQSQLNADT VEDAFMS YL TSFEQVANLK VISAYHDNIN NDQGLTYFIG LSETDAGEYY WRSVDHSKFN DGKFAANA W SEWHKIDCPI NPYKSTIRPV IYKSRLYLLW LEQKEITKQT GNSKDGYQTE TDYRYELKL AHIRYDGTWN TPITFDVNKK ISELKLEKNR APGLYCAGYQ GEDTLLVMFY NQQDTLDSYK NASMQGLYI FADMASKDMT PEQSNVYRDN SYQQFDTNNV RRVNNRYAED YEIPSSVSSR K DYGWGDYY LSMVYNGDIP TINYKAASSD LKIYISPKLR IIHNGYEGQK RNQCNLMNKY GK LGDKFIV YTSLGVNPNN SSNKLMFYPV YQYSGNTSGL NQGRLLFHRD TTYPSKVEAW IPG AKRSLT NQNAAIGDDY ATDSLNKPDD LKQYIFMTDS KGTATDVSGP VEINTAISPA KVQI IVKAG GKEQTFTADK DVSIQPSPSF DEMNYQFNAL EIDGSGLNFI NNSASIDVTF TAFAE DGRK LGYESFSIPV TLKVSTDNAL TLHHNENGAQ YMQWQSYRTR LNTLFARQLV ARATTG IDT ILSMETQNIQ EPQLGKGFYA TFVIPPYNLS THGDERWFKL YIKHVVDNNS HIIYSGQ LT DTNINITLFI PLDDVPLNQD YHAKVYMTFK KSPSDGTWWG PHFVRDDKGI VTINPKSI L THFESVNVLN NISSEPMDFS GANSLYFWEL FYYTPMLVAQ RLLHEQNFDE ANRWLKYVW SPSGYIVHGQ IQNYQWNVRP LLEDTSWNSD PLDSVDPDAV AQHDPMHYKV STFMRTLDLL IARGDHAYR QLERDTLNEA KMWYMQALHL LGDKPYLPLS TTWSDPRLDR AADITTQNAH D SAIVALRQ NIPTPAPLSL RSANTLTDLF LPQINEVMMN YWQTLAQRVY NLRHNLSIDG QP LYLPIYA TPADPKALLS AAVATSQGGG KLPESFMSLW RFPHMLENAR GMVSQLTQFG STL QNIIER QDAEALNALL QNQAAELILT NLSIQDKTIE ELDAEKTVLE KSKAGAQSRF DSYG KLYDE NINAGENQAM TLRASAAGLT TAVQASRLAG AAADLVPNIF GFAGGGSRWG AIAEA TGYV MEFSANVMNT EADKISQSET YRRRRQEWEI QRNNAEAELK QIDAQLKSLA VRREAA VLQ KTSLKTQQEQ TQSQLAFLQR KFSNQALYNW LRGRLAAIYF QFYDLAVARC LMAEQAY RW ELNDDSARFI KPGAWQGTYA GLLAGETLML SLAQMEDAHL KRDKRALEVE RTVSLAEV Y AGLPKDNGPF SLAQEIDKLV SQGSGSAGSG NNNLAFGAGT DTKTSLQASV SFADLKIRE DYPASLGKIR RIKQISVTLP ALLGPYQDVQ AILSYGDKAG LANGCEALAV SHGMNDSGQF QLDFNDGKF LPFEGIAIDQ GTLTLSFPNA SMPEKGKQAT MLKTLNDIIL HIRYTIK |
-分子 #2: BSA with Lewis X glycans crosslinked to surface lysines
| 分子 | 名称: BSA with Lewis X glycans crosslinked to surface lysines タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MKWVTFISLL LLFSSAYSRG VFRRDTHKSE IAHRFKDLGE EHFKGLVLIA FSQYLQQCPF DEHVKLVNE LTEFAKTCVA DESHAGCEKS LHTLFGDELC KVASLRETYG DMADCCEKQE P ERNECFLS HKDDSPDLPK LKPDPNTLCD EFKADEKKFW GKYLYEIARR ...文字列: MKWVTFISLL LLFSSAYSRG VFRRDTHKSE IAHRFKDLGE EHFKGLVLIA FSQYLQQCPF DEHVKLVNE LTEFAKTCVA DESHAGCEKS LHTLFGDELC KVASLRETYG DMADCCEKQE P ERNECFLS HKDDSPDLPK LKPDPNTLCD EFKADEKKFW GKYLYEIARR HPYFYAPELL YY ANKYNGV FQECCQAEDK GACLLPKIET MREKVLASSA RQRLRCASIQ KFGERALKAW SVA RLSQKF PKAEFVEVTK LVTDLTKVHK ECCHGDLLEC ADDRADLAKY ICDNQDTISS KLKE CCDKP LLEKSHCIAE VEKDAIPENL PPLTADFAED KDVCKNYQEA KDAFLGSFLY EYSRR HPEY AVSVLLRLAK EYEATLEECC AKDDPHACYS TVFDKLKHLV DEPQNLIKQN CDQFEK LGE YGFQNALIVR YTRKVPQVST PTLVEVSRSL GKVGTRCCTK PESERMPCTE DYLSLIL NR LCVLHEKTPV SEKVTKCCTE SLVNRRPCFS ALTPDETYVP KAFDEKLFTF HADICTLP D TEKQIKKQTA LVELLKHKPK ATEEQLKTVM ENFVAFVDKC CAADDKEACF AVEGPKLVV STQTALA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.06 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2.0 nm / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 293 K / 装置: GATAN CRYOPLUNGE 3 |
| 詳細 | The sample was crosslinked with glutaraldehyde and subsequently purified by SEC |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 実像数: 4992 / 平均電子線量: 100.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)