+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10577 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PKM2 in complex with Compound 6 | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | PKM2 / FBDD / CYTOSOLIC PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pyruvate kinase / pyruvate kinase activity / histone H3T11 kinase activity / programmed cell death / Pyruvate metabolism / positive regulation of cytoplasmic translation / canonical glycolysis / Glycolysis / positive regulation of sprouting angiogenesis / potassium ion binding ...pyruvate kinase / pyruvate kinase activity / histone H3T11 kinase activity / programmed cell death / Pyruvate metabolism / positive regulation of cytoplasmic translation / canonical glycolysis / Glycolysis / positive regulation of sprouting angiogenesis / potassium ion binding / rough endoplasmic reticulum / Regulation of pyruvate metabolism / glycolytic process / non-specific protein-tyrosine kinase / cellular response to insulin stimulus / MHC class II protein complex binding / extracellular vesicle / : / protein tyrosine kinase activity / secretory granule lumen / vesicle / ficolin-1-rich granule lumen / transcription coactivator activity / non-specific serine/threonine protein kinase / cilium / cadherin binding / intracellular membrane-bounded organelle / mRNA binding / Neutrophil degranulation / magnesium ion binding / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / mitochondrion / RNA binding / extracellular exosome / extracellular region / ATP binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Saur M / Hartshorn MJ | |||||||||
 引用 引用 |  ジャーナル: Drug Discov Today / 年: 2020 ジャーナル: Drug Discov Today / 年: 2020タイトル: Fragment-based drug discovery using cryo-EM. 著者: Michael Saur / Michael J Hartshorn / Jing Dong / Judith Reeks / Gabor Bunkoczi / Harren Jhoti / Pamela A Williams /  要旨: Recent advances in electron cryo-microscopy (cryo-EM) structure determination have pushed the resolutions obtainable by the method into the range widely considered to be of utility for drug discovery. ...Recent advances in electron cryo-microscopy (cryo-EM) structure determination have pushed the resolutions obtainable by the method into the range widely considered to be of utility for drug discovery. Here, we review the use of cryo-EM in fragment-based drug discovery (FBDD) based on in-house method development. We demonstrate not only that cryo-EM can reveal details of the molecular interactions between fragments and a protein, but also that the current reproducibility, quality, and throughput are compatible with FBDD. We exemplify this using the test system β-galactosidase (Bgal) and the oncology target pyruvate kinase 2 (PKM2). | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10577.map.gz emd_10577.map.gz | 30.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10577-v30.xml emd-10577-v30.xml emd-10577.xml emd-10577.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10577.png emd_10577.png | 141.5 KB | ||
| Filedesc metadata |  emd-10577.cif.gz emd-10577.cif.gz | 6.1 KB | ||
| その他 |  emd_10577_additional_1.map.gz emd_10577_additional_1.map.gz emd_10577_additional_2.map.gz emd_10577_additional_2.map.gz emd_10577_half_map_1.map.gz emd_10577_half_map_1.map.gz emd_10577_half_map_2.map.gz emd_10577_half_map_2.map.gz | 160.1 MB 3 MB 134 MB 134.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10577 http://ftp.pdbj.org/pub/emdb/structures/EMD-10577 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10577 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10577 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10577_validation.pdf.gz emd_10577_validation.pdf.gz | 817.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10577_full_validation.pdf.gz emd_10577_full_validation.pdf.gz | 817.3 KB | 表示 | |
| XML形式データ |  emd_10577_validation.xml.gz emd_10577_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_10577_validation.cif.gz emd_10577_validation.cif.gz | 13.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10577 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10577 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10577 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10577 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6ttiMC  6tshC  6tskC  6tteC  6ttfC  6tthC  6ttqC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10650 (タイトル: PKM2 in complex with Compound 6 / Data size: 768.0 EMPIAR-10650 (タイトル: PKM2 in complex with Compound 6 / Data size: 768.0 Data #1: Data from EPU (movies have been converted to compressed TIF) [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10577.map.gz / 形式: CCP4 / 大きさ: 38.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10577.map.gz / 形式: CCP4 / 大きさ: 38.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Relion post-process unmasked map from halfmaps which have...
| ファイル | emd_10577_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion post-process unmasked map from halfmaps which have not been resampled after auto-refine. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
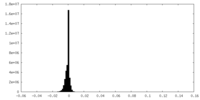
| 密度ヒストグラム |
-追加マップ: None
| ファイル | emd_10577_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
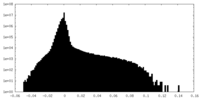
| 密度ヒストグラム |
-ハーフマップ: Relion auto-refine halfmap 1
| ファイル | emd_10577_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion auto-refine halfmap 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
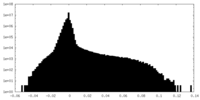
| 密度ヒストグラム |
-ハーフマップ: Relion auto-refine halfmap 2
| ファイル | emd_10577_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion auto-refine halfmap 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pyruvate Kinase M2
| 全体 | 名称: Pyruvate Kinase M2 |
|---|---|
| 要素 |
|
-超分子 #1: Pyruvate Kinase M2
| 超分子 | 名称: Pyruvate Kinase M2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 240 KDa |
-分子 #1: Pyruvate kinase PKM
| 分子 | 名称: Pyruvate kinase PKM / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: pyruvate kinase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 59.83182 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSK PHSEAGTAFI QTQQLHAAMA DTFLEHMCRL DIDSPPITAR NTGIICTIGP ASRSVETLKE MIKSGMNVA RLNFSHGTHE YHAETIKNVR TATESFASDP ILYRPVAVAL DTKGPEIRTG LIKGSGTAEV ELKKGATLKI T LDNAYMEK ...文字列: MGSSHHHHHH SSGLVPRGSK PHSEAGTAFI QTQQLHAAMA DTFLEHMCRL DIDSPPITAR NTGIICTIGP ASRSVETLKE MIKSGMNVA RLNFSHGTHE YHAETIKNVR TATESFASDP ILYRPVAVAL DTKGPEIRTG LIKGSGTAEV ELKKGATLKI T LDNAYMEK CDENILWLDY KNICKVVEVG SKIYVDDGLI SLQVKQKGAD FLVTEVENGG SLGSKKGVNL PGAAVDLPAV SE KDIQDLK FGVEQDVDMV FASFIRKASD VHEVRKVLGE KGKNIKIISK IENHEGVRRF DEILEASDGI MVARGDLGIE IPA EKVFLA QKMMIGRCNR AGKPVICATQ MLESMIKKPR PTRAEGSDVA NAVLDGADCI MLSGETAKGD YPLEAVRMQH LIAR EAEAA IYHLQLFEEL RRLAPITSDP TEATAVGAVE ASFKCCSGAI IVLTKSGRSA HQVARYRPRA PIIAVTRNPQ TARQA HLYR GIFPVLCKDP VQEAWAEDVD LRVNFAMNVG KARGFFKKGD VVIVLTGWRP GSGFTNTMRV VPVP UniProtKB: Pyruvate kinase PKM |
-分子 #2: ~{N}-[3-(trifluoromethyl)-1,2-oxazol-5-yl]ethanamide
| 分子 | 名称: ~{N}-[3-(trifluoromethyl)-1,2-oxazol-5-yl]ethanamide タイプ: ligand / ID: 2 / コピー数: 4 / 式: NXE |
|---|---|
| 分子量 | 理論値: 194.111 Da |
| Chemical component information |  ChemComp-NXE: |
-分子 #3: 1,6-di-O-phosphono-beta-D-fructofuranose
| 分子 | 名称: 1,6-di-O-phosphono-beta-D-fructofuranose / タイプ: ligand / ID: 3 / コピー数: 4 / 式: FBP |
|---|---|
| 分子量 | 理論値: 340.116 Da |
| Chemical component information | 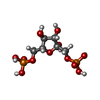 ChemComp-FBP: |
-分子 #4: DIMETHYL SULFOXIDE
| 分子 | 名称: DIMETHYL SULFOXIDE / タイプ: ligand / ID: 4 / コピー数: 4 / 式: DMS |
|---|---|
| 分子量 | 理論値: 78.133 Da |
| Chemical component information |  ChemComp-DMS: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 220 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.16 mg/mL |
|---|---|
| 緩衝液 | pH: 8.2 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 590 / 平均露光時間: 59.98 sec. / 平均電子線量: 65.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)