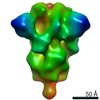
登録情報 データベース : EMDB / ID : EMD-10545タイトル bacterial RNA polymerase rrnTAC delta S4 global state 複合体 : bacterial rRNA transcription anti-termination complex機能・相同性 分子機能 ドメイン・相同性 構成要素
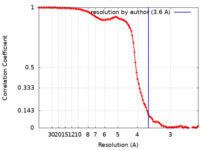
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Escherichia coli (大腸菌)手法 / / 解像度 : 3.6 Å Hilal T / Huang Y / Wahl MC 資金援助 Organization Grant number 国 German Research Foundation (DFG) RTG 2473-1 German Research Foundation (DFG) INST 130/1014-1 FUGG
ジャーナル : Mol Cell / 年 : 2020タイトル : Structure-Based Mechanisms of a Molecular RNA Polymerase/Chaperone Machine Required for Ribosome Biosynthesis.著者 : Yong-Heng Huang / Tarek Hilal / Bernhard Loll / Jörg Bürger / Thorsten Mielke / Christoph Böttcher / Nelly Said / Markus C Wahl / 要旨 : Bacterial ribosomal RNAs are synthesized by a dedicated, conserved transcription-elongation complex that transcribes at high rates, shields RNA polymerase from premature termination, and supports co- ... Bacterial ribosomal RNAs are synthesized by a dedicated, conserved transcription-elongation complex that transcribes at high rates, shields RNA polymerase from premature termination, and supports co-transcriptional RNA folding, modification, processing, and ribosomal subunit assembly by presently unknown mechanisms. We have determined cryo-electron microscopy structures of complete Escherichia coli ribosomal RNA transcription elongation complexes, comprising RNA polymerase; DNA; RNA bearing an N-utilization-site-like anti-termination element; Nus factors A, B, E, and G; inositol mono-phosphatase SuhB; and ribosomal protein S4. Our structures and structure-informed functional analyses show that fast transcription and anti-termination involve suppression of NusA-stabilized pausing, enhancement of NusG-mediated anti-backtracking, sequestration of the NusG C-terminal domain from termination factor ρ, and the ρ blockade. Strikingly, the factors form a composite RNA chaperone around the RNA polymerase RNA-exit tunnel, which supports co-transcriptional RNA folding and annealing of distal RNA regions. Our work reveals a polymerase/chaperone machine required for biosynthesis of functional ribosomes. 履歴 登録 2019年12月16日 - ヘッダ(付随情報) 公開 2020年12月16日 - マップ公開 2020年12月16日 - 更新 2021年4月14日 - 現状 2021年4月14日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 ドイツ, 2件
ドイツ, 2件  引用
引用 ジャーナル: Mol Cell / 年: 2020
ジャーナル: Mol Cell / 年: 2020
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_10545.map.gz
emd_10545.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-10545-v30.xml
emd-10545-v30.xml emd-10545.xml
emd-10545.xml EMDBヘッダ
EMDBヘッダ emd_10545_fsc.xml
emd_10545_fsc.xml FSCデータファイル
FSCデータファイル emd_10545.png
emd_10545.png http://ftp.pdbj.org/pub/emdb/structures/EMD-10545
http://ftp.pdbj.org/pub/emdb/structures/EMD-10545 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10545
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10545 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_10545.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_10545.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素

 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析 ムービー
ムービー コントローラー
コントローラー


























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)