+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10450 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Endoplasmic Reticulum microsome used for Videos in (Martinez-Sanchez et al., Nature Methods) | |||||||||
 マップデータ マップデータ | ER microsome used for Videos (Martinez-Sanchez et al., Nature Methods) | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Martinez-Sanchez A / Lucic V / Pfeffer S / Forster F | |||||||||
| 資金援助 |  スペイン, 1件 スペイン, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2020 ジャーナル: Nat Methods / 年: 2020タイトル: Template-free detection and classification of membrane-bound complexes in cryo-electron tomograms. 著者: Antonio Martinez-Sanchez / Zdravko Kochovski / Ulrike Laugks / Johannes Meyer Zum Alten Borgloh / Saikat Chakraborty / Stefan Pfeffer / Wolfgang Baumeister / Vladan Lučić /  要旨: With faithful sample preservation and direct imaging of fully hydrated biological material, cryo-electron tomography provides an accurate representation of molecular architecture of cells. However, ...With faithful sample preservation and direct imaging of fully hydrated biological material, cryo-electron tomography provides an accurate representation of molecular architecture of cells. However, detection and precise localization of macromolecular complexes within cellular environments is aggravated by the presence of many molecular species and molecular crowding. We developed a template-free image processing procedure for accurate tracing of complex networks of densities in cryo-electron tomograms, a comprehensive and automated detection of heterogeneous membrane-bound complexes and an unsupervised classification (PySeg). Applications to intact cells and isolated endoplasmic reticulum (ER) allowed us to detect and classify small protein complexes. This classification provided sufficiently homogeneous particle sets and initial references to allow subsequent de novo subtomogram averaging. Spatial distribution analysis showed that ER complexes have different localization patterns forming nanodomains. Therefore, this procedure allows a comprehensive detection and structural analysis of complexes in situ. #1:  ジャーナル: Nature Communications / 年: 2015 ジャーナル: Nature Communications / 年: 2015タイトル: Structure of the native Sec61 protein-conducting channel 著者: Pfeffer S / Forster F | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10450.map.gz emd_10450.map.gz | 35 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10450-v30.xml emd-10450-v30.xml emd-10450.xml emd-10450.xml | 12.9 KB 12.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10450.png emd_10450.png | 220.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10450 http://ftp.pdbj.org/pub/emdb/structures/EMD-10450 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10450 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10450 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10450_validation.pdf.gz emd_10450_validation.pdf.gz | 210.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10450_full_validation.pdf.gz emd_10450_full_validation.pdf.gz | 209.9 KB | 表示 | |
| XML形式データ |  emd_10450_validation.xml.gz emd_10450_validation.xml.gz | 4.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10450 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10450 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10450 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10450 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10450.map.gz / 形式: CCP4 / 大きさ: 37.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10450.map.gz / 形式: CCP4 / 大きさ: 37.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
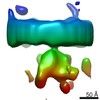
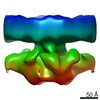
| 注釈 | ER microsome used for Videos (Martinez-Sanchez et al., Nature Methods) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 10.48 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
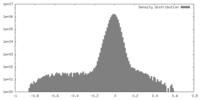
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Representative tomogram of a canine pancreatic rough Endoplasmic ...
| 全体 | 名称: Representative tomogram of a canine pancreatic rough Endoplasmic Reticulum vesicle. |
|---|---|
| 要素 |
|
-超分子 #1: Representative tomogram of a canine pancreatic rough Endoplasmic ...
| 超分子 | 名称: Representative tomogram of a canine pancreatic rough Endoplasmic Reticulum vesicle. タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.00 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 70 % / 装置: FEI VITROBOT MARK IV / 詳細: Blot 3 seconds before plunging.. | ||||||||||||
| 切片作成 | その他: NO SECTIONING | ||||||||||||
| 位置合わせマーカー | Manufacturer: Aurion, Wageningen, The Netherlands / 直径: 10 nm |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 1.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: OTHER / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 3.0 µm / 倍率(公称値): 42000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Reconstruction of a single microsome after two binning steps (from 2.62A/pixel to 10.48A/pixel) |
|---|---|
| 最終 再構成 | ソフトウェア - 名称: PyTom / 使用した粒子像数: 61 |
| CTF補正 | ソフトウェア - 名称: PyTom |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)