+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10286 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cubic Dps-DNA crystal structure in vitro | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
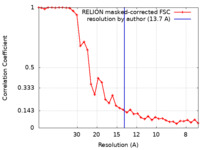
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 13.7 Å | |||||||||
 データ登録者 データ登録者 | Chesnokov YM / Kamyshinsky RA / Vasiliev AL | |||||||||
| 資金援助 |  ロシア, 1件 ロシア, 1件
| |||||||||
 引用 引用 |  ジャーナル: Biomolecules / 年: 2019 ジャーナル: Biomolecules / 年: 2019タイトル: Polymorphic Protective Dps-DNA Co-Crystals by Cryo Electron Tomography and Small Angle X-Ray Scattering. 著者: Roman Kamyshinsky / Yury Chesnokov / Liubov Dadinova / Andrey Mozhaev / Ivan Orlov / Maxim Petoukhov / Anton Orekhov / Eleonora Shtykova / Alexander Vasiliev /  要旨: Rapid increase of intracellular synthesis of specific histone-like Dps protein that binds DNA to protect the genome against deleterious factors leads to in cellulo crystallization-one of the most ...Rapid increase of intracellular synthesis of specific histone-like Dps protein that binds DNA to protect the genome against deleterious factors leads to in cellulo crystallization-one of the most curious processes in the area of life science at the moment. However, the actual structure of the Dps-DNA co-crystals remained uncertain in the details for more than two decades. Cryo-electron tomography and small-angle X-ray scattering revealed polymorphous modifications of the co-crystals depending on the buffer parameters. Two different types of the Dps-DNA co-crystals are formed in vitro: triclinic and cubic. Three-dimensional reconstruction revealed DNA and Dps molecules in cubic co-crystals, and the unit cell parameters of cubic lattice were determined consistently by both methods. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10286.map.gz emd_10286.map.gz | 2.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10286-v30.xml emd-10286-v30.xml emd-10286.xml emd-10286.xml | 19.2 KB 19.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10286_fsc.xml emd_10286_fsc.xml | 3.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10286.png emd_10286.png | 168.1 KB | ||
| マスクデータ |  emd_10286_msk_1.map emd_10286_msk_1.map | 2.8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10286_additional.map.gz emd_10286_additional.map.gz emd_10286_additional_1.map.gz emd_10286_additional_1.map.gz emd_10286_half_map_1.map.gz emd_10286_half_map_1.map.gz emd_10286_half_map_2.map.gz emd_10286_half_map_2.map.gz | 1.9 MB 1.9 MB 1.9 MB 1.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10286 http://ftp.pdbj.org/pub/emdb/structures/EMD-10286 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10286 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10286 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10286_validation.pdf.gz emd_10286_validation.pdf.gz | 398.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10286_full_validation.pdf.gz emd_10286_full_validation.pdf.gz | 397.8 KB | 表示 | |
| XML形式データ |  emd_10286_validation.xml.gz emd_10286_validation.xml.gz | 8.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10286 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10286 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10286 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10286 | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10286.map.gz / 形式: CCP4 / 大きさ: 2.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10286.map.gz / 形式: CCP4 / 大きさ: 2.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10286_msk_1.map emd_10286_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
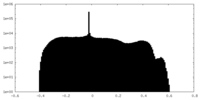
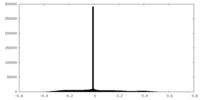
| 密度ヒストグラム |
-追加マップ: Map before any post-processing
| ファイル | emd_10286_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map before any post-processing | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
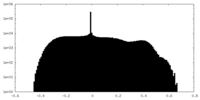
| 密度ヒストグラム |
-追加マップ: Map before any post-processing
| ファイル | emd_10286_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map before any post-processing | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
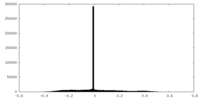
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10286_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10286_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dps-DNA co-crystal
| 全体 | 名称: Dps-DNA co-crystal |
|---|---|
| 要素 |
|
-超分子 #1: Dps-DNA co-crystal
| 超分子 | 名称: Dps-DNA co-crystal / タイプ: complex / ID: 1 / 親要素: 0 詳細: Dps protein and DNA were mixed in the concentrations corresponding to the Dps/DNA ratio 1Dps dodecamer/60 bp of DNA. A ring vector pcDNA-hIRR-GFP 9900 bp was used as DNA sample. |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 224 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 濃度 | 3.1 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 / 構成要素:
| ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV / 詳細: blotting for 1.5 seconds. | ||||||
| 詳細 | weight Dps/DNA ratio = 3/1 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 球面収差補正装置: Cs image corrector (CEOS, Germany) |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / 撮影したグリッド数: 1 / 平均露光時間: 1.0 sec. / 平均電子線量: 1.02 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 37837 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 3.0 µm / 倍率(公称値): 18000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: Cross-correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)