+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0920 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the human CALHM2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | channel / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of microglial cell activation / ATP export / calcium ion import / monoatomic cation channel activity / regulation of synaptic plasticity / positive regulation of apoptotic process / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
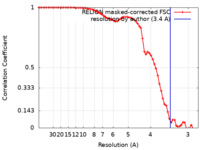
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Demura K / Kusakizako T | |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2020 ジャーナル: Sci Adv / 年: 2020タイトル: Cryo-EM structures of calcium homeostasis modulator channels in diverse oligomeric assemblies. 著者: Kanae Demura / Tsukasa Kusakizako / Wataru Shihoya / Masahiro Hiraizumi / Kengo Nomura / Hiroto Shimada / Keitaro Yamashita / Tomohiro Nishizawa / Akiyuki Taruno / Osamu Nureki /  要旨: Calcium homeostasis modulator (CALHM) family proteins are Ca-regulated adenosine triphosphate (ATP)-release channels involved in neural functions including neurotransmission in gustation. Here, we ...Calcium homeostasis modulator (CALHM) family proteins are Ca-regulated adenosine triphosphate (ATP)-release channels involved in neural functions including neurotransmission in gustation. Here, we present the cryo-electron microscopy (EM) structures of killifish CALHM1, human CALHM2, and CLHM-1 at resolutions of 2.66, 3.4, and 3.6 Å, respectively. The CALHM1 octamer structure reveals that the N-terminal helix forms the constriction site at the channel pore in the open state and modulates the ATP conductance. The CALHM2 undecamer and CLHM-1 nonamer structures show the different oligomeric stoichiometries among CALHM homologs. We further report the cryo-EM structures of the chimeric construct, revealing that the intersubunit interactions at the transmembrane domain (TMD) and the TMD-intracellular domain linker define the oligomeric stoichiometry. These findings advance our understanding of the ATP conduction and oligomerization mechanisms of CALHM channels. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0920.map.gz emd_0920.map.gz | 3.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0920-v30.xml emd-0920-v30.xml emd-0920.xml emd-0920.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0920_fsc.xml emd_0920_fsc.xml | 6.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0920.png emd_0920.png | 141.2 KB | ||
| マスクデータ |  emd_0920_msk_1.map emd_0920_msk_1.map | 26.2 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-0920.cif.gz emd-0920.cif.gz | 5.8 KB | ||
| その他 |  emd_0920_half_map_1.map.gz emd_0920_half_map_1.map.gz emd_0920_half_map_2.map.gz emd_0920_half_map_2.map.gz | 19.9 MB 19.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0920 http://ftp.pdbj.org/pub/emdb/structures/EMD-0920 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0920 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0920 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0920_validation.pdf.gz emd_0920_validation.pdf.gz | 664 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0920_full_validation.pdf.gz emd_0920_full_validation.pdf.gz | 663.5 KB | 表示 | |
| XML形式データ |  emd_0920_validation.xml.gz emd_0920_validation.xml.gz | 13.7 KB | 表示 | |
| CIF形式データ |  emd_0920_validation.cif.gz emd_0920_validation.cif.gz | 17.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0920 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0920 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0920 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0920 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6lmuMC  0919C  0921C  0922C  0923C  6lmtC  6lmvC  6lmwC  6lmxC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10444 (タイトル: Cryo-EM structures of calcium homeostasis modulator (CALHM) channels EMPIAR-10444 (タイトル: Cryo-EM structures of calcium homeostasis modulator (CALHM) channelsData size: 6.8 TB Data #1: Unaligned movies for OlCALHM1 [micrographs - multiframe] Data #2: Unaligned movies for HsCALHM2 [micrographs - multiframe] Data #3: Unaligned movies for CeCLHM-1 [micrographs - multiframe] Data #4: Unaligned movies for OlCALHM1-HsCALHM2 chimera [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0920.map.gz / 形式: CCP4 / 大きさ: 26.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0920.map.gz / 形式: CCP4 / 大きさ: 26.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.36172 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
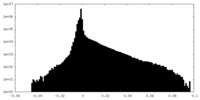
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0920_msk_1.map emd_0920_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
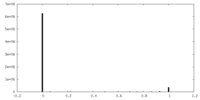
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_0920_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
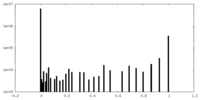
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_0920_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
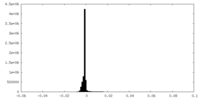
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CALHM2
| 全体 | 名称: CALHM2 |
|---|---|
| 要素 |
|
-超分子 #1: CALHM2
| 超分子 | 名称: CALHM2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Calcium homeostasis modulator protein 2
| 分子 | 名称: Calcium homeostasis modulator protein 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 11 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 37.006367 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAALIAENFR FLSLFFKSKD VMIFNGLVAL GTVGSQELFS VVAFHCPCSP ARNYLYGLAA IGVPALVLFI IGIILNNHTW NLVAECQHR RTKNCSAAPT FLLLSSILGR AAVAPVTWSV ISLLRGEAYV CALSEFVDPS SLTAREEHFP SAHATEILAR F PCKENPDN ...文字列: MAALIAENFR FLSLFFKSKD VMIFNGLVAL GTVGSQELFS VVAFHCPCSP ARNYLYGLAA IGVPALVLFI IGIILNNHTW NLVAECQHR RTKNCSAAPT FLLLSSILGR AAVAPVTWSV ISLLRGEAYV CALSEFVDPS SLTAREEHFP SAHATEILAR F PCKENPDN LSDFREEVSR RLRYESQLFG WLLIGVVAIL VFLTKCLKHY CSPLSYRQEA YWAQYRANED QLFQRTAEVH SR VLAANNV RRFFGFVALN KDDEELIANF PVEGTQPRPQ WNAITGVYLY RENQGLPLYS RLHKWAQGLA GNGAAPDNVE MAL LPSENL YFQ UniProtKB: Calcium homeostasis modulator protein 2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)