+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0724 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | FimA type V pilus from P.gingivalis | ||||||||||||||||||
 マップデータ マップデータ | FimA pilus cryo-EM reconstruction | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | Type V pilus / FimA / Porphyromonas gingivalis / ATCC33277 / CELL ADHESION | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||||||||
| 生物種 |  Porphyromonas gingivalis ATCC 33277 (バクテリア) Porphyromonas gingivalis ATCC 33277 (バクテリア) | ||||||||||||||||||
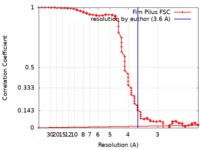
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Shibata S / Shoji M | ||||||||||||||||||
| 資金援助 |  日本, 5件 日本, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2020 ジャーナル: Nat Microbiol / 年: 2020タイトル: Structure of polymerized type V pilin reveals assembly mechanism involving protease-mediated strand exchange. 著者: Satoshi Shibata / Mikio Shoji / Kodai Okada / Hideyuki Matsunami / Melissa M Matthews / Katsumi Imada / Koji Nakayama / Matthias Wolf /  要旨: Bacterial adhesion is a general strategy for host-microbe and microbe-microbe interactions. Adhesive pili are essential for colonization, biofilm formation, virulence and pathogenesis of many ...Bacterial adhesion is a general strategy for host-microbe and microbe-microbe interactions. Adhesive pili are essential for colonization, biofilm formation, virulence and pathogenesis of many environmental and pathogenic bacteria. Members of the class Bacteroidia have unique type V pili, assembled by protease-mediated polymerization. Porphyromonas gingivalis is the main contributor to periodontal disease and its type V pili are a key factor for its virulence. However, the structure of the polymerized pilus and its assembly mechanism are unknown. Here we show structures of polymerized and monomeric states of FimA stalk pilin from P. gingivalis, determined by cryo-electron microscopy and crystallography. The atomic model of assembled FimA shows that the C-terminal strand of a donor subunit is inserted into a groove in the β-sheet of an acceptor subunit after N-terminal cleavage by the protease RgpB. The C terminus of the donor strand is essential for polymerization. We propose that type V pili assemble via a sequential polar assembly mechanism at the cell surface, involving protease-mediated strand exchange, employed by various Gram-negative species belonging to the class Bacteroidia. Our results reveal functional surfaces related to pathogenic properties of polymerized FimA. These insights may facilitate development of antibacterial drugs. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0724.map.gz emd_0724.map.gz | 200.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0724-v30.xml emd-0724-v30.xml emd-0724.xml emd-0724.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0724_fsc.xml emd_0724_fsc.xml | 15.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0724.png emd_0724.png | 43 KB | ||
| Filedesc metadata |  emd-0724.cif.gz emd-0724.cif.gz | 6.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0724 http://ftp.pdbj.org/pub/emdb/structures/EMD-0724 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0724 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0724 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0724_validation.pdf.gz emd_0724_validation.pdf.gz | 811.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0724_full_validation.pdf.gz emd_0724_full_validation.pdf.gz | 810.8 KB | 表示 | |
| XML形式データ |  emd_0724_validation.xml.gz emd_0724_validation.xml.gz | 11 KB | 表示 | |
| CIF形式データ |  emd_0724_validation.cif.gz emd_0724_validation.cif.gz | 15.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0724 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0724 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0724 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0724 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6kmfMC  6jzjC  6jzkC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-11015 (タイトル: Raw data for "Structure of polymerized type V pilin reveals assembly mechanism involving protease-mediated strand exchange" EMPIAR-11015 (タイトル: Raw data for "Structure of polymerized type V pilin reveals assembly mechanism involving protease-mediated strand exchange"Data size: 72.1 Data #1: FimA motion-corrected dose-weighted frame sums [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0724.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0724.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | FimA pilus cryo-EM reconstruction | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Type V pilus (FimA)
| 全体 | 名称: Type V pilus (FimA) |
|---|---|
| 要素 |
|
-超分子 #1: Type V pilus (FimA)
| 超分子 | 名称: Type V pilus (FimA) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Porphyromonas gingivalis ATCC 33277 (バクテリア) Porphyromonas gingivalis ATCC 33277 (バクテリア) |
| 分子量 | 理論値: 6 kDa/nm |
-分子 #1: Major fimbrium subunit FimA type-1
| 分子 | 名称: Major fimbrium subunit FimA type-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Porphyromonas gingivalis ATCC 33277 (バクテリア) Porphyromonas gingivalis ATCC 33277 (バクテリア) |
| 分子量 | 理論値: 36.488805 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AFGVGDDESK VAKLTVMVYN GEQQEAIKSA ENATKVEDIK CSAGQRTLVV MANTGAMELV GKTLAEVKAL TTELTAENQE AAGLIMTAE PKTIVLKAGK NYIGYSGTGE GNHIENDPLK IKRVHARMAF TEIKVQMSAA YDNIYTFVPE KIYGLIAKKQ S NLFGATLV ...文字列: AFGVGDDESK VAKLTVMVYN GEQQEAIKSA ENATKVEDIK CSAGQRTLVV MANTGAMELV GKTLAEVKAL TTELTAENQE AAGLIMTAE PKTIVLKAGK NYIGYSGTGE GNHIENDPLK IKRVHARMAF TEIKVQMSAA YDNIYTFVPE KIYGLIAKKQ S NLFGATLV NADANYLTGS LTTFNGAYTP ANYANVPWLS RNYVAPAADA PQGFYVLEND YSANGGTIHP TILCVYGKLQ KN GADLAGA DLAAAQAANW VDAEGKTYYP VLVNFNSNNY TYDSNYTPKN KIERNHKYDI KLTITGPGTN NPENPITESA HLN VQCTVA EWVLVGQNAT W UniProtKB: Major fimbrium subunit FimA type-1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 構成要素 - 濃度: 20.0 mM / 構成要素 - 式: C4H11NO3 / 構成要素 - 名称: Tris |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 100.1 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 1e-06 kPa / 詳細: Gatan Solarus |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 289 K / 装置: FEI VITROBOT MARK IV / 詳細: 3 second blot, 3.0uL. |
| 詳細 | matured, polymerized state |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 100.0 K |
| アライメント法 | Coma free - Residual tilt: 0.1 mrad |
| 詳細 | nanoprobe, parallel beam illumination |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4000 pixel / デジタル化 - サイズ - 縦: 4000 pixel / デジタル化 - 画像ごとのフレーム数: 1-75 / 撮影したグリッド数: 1 / 実像数: 1153 / 平均露光時間: 60.0 sec. / 平均電子線量: 46.0 e/Å2 / 詳細: frame alignment and dose weighting using motioncor2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 30.0 µm / 倍率(補正後): 125000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.5 µm / 最小 デフォーカス(公称値): -1.5 µm / 倍率(公称値): 92000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)