+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 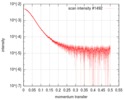 |
|---|---|
 試料 試料 | GON7, the fifth subunit of human KEOPS, bound to the EKC/KEOPS complex subunit, LAGE3
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報EKC/KEOPS complex / tRNA threonylcarbamoyladenosine metabolic process / tRNA threonylcarbamoyladenosine modification / tRNA modification in the nucleus and cytosol / tRNA processing / nuclear body / nucleolus / nucleoplasm / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 |
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
 引用 引用 |  日付: 2019 Dec 日付: 2019 Decタイトル: Defects in t6A tRNA modification due to GON7 and YRDC mutations lead to Galloway-Mowat syndrome 著者: Arrondel C / Missoury S / Snoek R / Patat J / Menara G / Collinet B / Liger D / Durand D / Gribouval O / Boyer O / Buscara L / Martin G / Machuca E / Nevo F / Lescop E / Braun D / Boschat A / ...著者: Arrondel C / Missoury S / Snoek R / Patat J / Menara G / Collinet B / Liger D / Durand D / Gribouval O / Boyer O / Buscara L / Martin G / Machuca E / Nevo F / Lescop E / Braun D / Boschat A / Sanquer S / Guerrera I / Revy P / Parisot M / Masson C / Boddaert N / Charbit M / Decramer S / Novo R / Macher M / Ranchin B / Bacchetta J / Laurent A / Collardeau-Frachon S / van Eerde A / Hildebrandt F / Magen D / Antignac C / van Tilbeurgh H |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: GON7, the fifth subunit of human KEOPS, bound to the EKC/KEOPS complex subunit, LAGE3 試料濃度: 0.7 mg/ml / Entity id: 1764 / 1765 |
|---|---|
| バッファ | 名称: 20 mM MES, 200 mM NaCl, 5 mM β-mercaptoethanol / pH: 6.5 |
| 要素 #1764 | 名称: GON7 / タイプ: protein / 記述: EKC/KEOPS complex subunit GON7 / 分子量: 12.59 / 分子数: 2 / 由来: Homo sapiens / 参照: UniProt: Q9BXV9 配列: MGHHHHHHEN LYFQGELLGE YVGQEGKPQK LRVSCEAPGD GDPFQGLLSG VAQMKDMVTE LFDPLVQGEV QHRVAAAPDE DLDGDDEDDA EDENNIDNRT NFDGPSAKRP KTPS |
| 要素 #1765 | 名称: LAGE3 / タイプ: protein / 記述: EKC/KEOPS complex subunit LAGE3 / 分子量: 16.478 / 分子数: 2 / 由来: Homo sapiens / 参照: UniProt: Q14657 配列: MHHHHHHENL YFQGRDADAD AGGGADGGDG RGGHSCRGGV DTAAAPAGGA PPAHAPGPGR DAASAARGSR MRPHIFTLSV PFPTPLEAEI AHGSLAPDAE PHQRVVGKDL TVSGRILVVR WKAEDCRLLR ISVINFLDQL SLVVRTMQRF GPPVSR |
-実験情報
| ビーム | 設備名称: SOLEIL SWING  / 地域: Saint-Aubin / 国: France / 地域: Saint-Aubin / 国: France  / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 1.8 mm / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 1.8 mm | |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: AVIEX PCCD170170 / タイプ: CCD | |||||||||||||||||||||||||||||||||
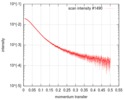
| スキャン | 測定日: 2017年3月19日 / セル温度: 15 °C / 照射時間: 1 sec. / フレーム数: 255 / 単位: 1/A /
| |||||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| |||||||||||||||||||||||||||||||||
| 結果 | コメント: The scattered intensities were displayed on an absolute scale (cm-1) using the scattering of water. SAXS data were normalized to the intensity of the incident beam, averaged and ...コメント: The scattered intensities were displayed on an absolute scale (cm-1) using the scattering of water. SAXS data were normalized to the intensity of the incident beam, averaged and background subtracted using the program US-SOMO. Frames were examined individually and 36 frames were averaged and further processed. The corresponding SEC-elution concentration was approximately 0.7 mg/mL.
|
 ムービー
ムービー コントローラー
コントローラー



 SASDFM8
SASDFM8