+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  |
|---|---|
 試料 試料 | RXR/RAR Heterodimer : N-CoRNID Complex (1:1) with RAR agonist (Am580)
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Transcriptional regulation of granulopoiesis / Carnitine shuttle / Transcriptional regulation of white adipocyte differentiation / Signaling by Retinoic Acid / Sertoli cell fate commitment / positive regulation of binding / NR1H2 & NR1H3 regulate gene expression to control bile acid homeostasis / NR1H3 & NR1H2 regulate gene expression linked to cholesterol transport and efflux / definitive erythrocyte differentiation / SUMOylation of intracellular receptors ...Transcriptional regulation of granulopoiesis / Carnitine shuttle / Transcriptional regulation of white adipocyte differentiation / Signaling by Retinoic Acid / Sertoli cell fate commitment / positive regulation of binding / NR1H2 & NR1H3 regulate gene expression to control bile acid homeostasis / NR1H3 & NR1H2 regulate gene expression linked to cholesterol transport and efflux / definitive erythrocyte differentiation / SUMOylation of intracellular receptors / trachea cartilage development / Recycling of bile acids and salts / Downregulation of SMAD2/3:SMAD4 transcriptional activity / Synthesis of bile acids and bile salts / CD4-positive, CD25-positive, alpha-beta regulatory T cell differentiation / Nuclear Receptor transcription pathway / visceral serous pericardium development / glandular epithelial cell development / Synthesis of bile acids and bile salts via 7alpha-hydroxycholesterol / Synthesis of bile acids and bile salts via 27-hydroxycholesterol / ventricular cardiac muscle cell differentiation / mesenchyme development / HDACs deacetylate histones / Endogenous sterols / Notch-HLH transcription pathway / chondroblast differentiation / positive regulation of translational initiation by iron / embryonic camera-type eye development / MLL4 and MLL3 complexes regulate expression of PPARG target genes in adipogenesis and hepatic steatosis / maternal placenta development / growth plate cartilage development / negative regulation of granulocyte differentiation / protein kinase B binding / angiogenesis involved in coronary vascular morphogenesis / prostate gland development / positive regulation of T-helper 2 cell differentiation / Regulation of lipid metabolism by PPARalpha / retinoic acid-responsive element binding / negative regulation of cartilage development / regulation of hematopoietic progenitor cell differentiation / Cytoprotection by HMOX1 / secretory columnal luminar epithelial cell differentiation involved in prostate glandular acinus development / positive regulation of thyroid hormone receptor signaling pathway / positive regulation of interleukin-13 production / positive regulation of interleukin-5 production / cardiac muscle cell differentiation / nuclear retinoic acid receptor binding / thalamus development / retinoic acid binding / camera-type eye development / outflow tract septum morphogenesis / regulation of thyroid hormone receptor signaling pathway / TGFBR3 expression / positive regulation of vitamin D receptor signaling pathway / response to vitamin A / heterocyclic compound binding / nuclear thyroid hormone receptor binding / limb development / apoptotic cell clearance / ureteric bud development / Signaling by Retinoic Acid / histone deacetylase regulator activity / regulation of myelination / DNA-binding transcription repressor activity / RNA polymerase II intronic transcription regulatory region sequence-specific DNA binding / regulation of branching involved in prostate gland morphogenesis / ventricular cardiac muscle tissue morphogenesis / protein kinase A binding / positive regulation of interleukin-4 production / locomotor rhythm / face development / germ cell development / alpha-actinin binding / negative regulation of type II interferon production / nuclear steroid receptor activity / cellular response to Thyroglobulin triiodothyronine / negative regulation of tumor necrosis factor production / regulation of multicellular organism growth / cellular response to estrogen stimulus / epidermis development / establishment of skin barrier / cardiac muscle cell proliferation / response to retinoic acid / positive regulation of bone mineralization / nuclear retinoid X receptor binding / heart morphogenesis / retinoic acid receptor signaling pathway / hormone-mediated signaling pathway / cellular response to retinoic acid / positive regulation of cell cycle / response to cytokine / transcription repressor complex / positive regulation of neuron differentiation / embryo implantation / negative regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / negative regulation of miRNA transcription / mRNA regulatory element binding translation repressor activity / cholesterol homeostasis / placenta development / RNA polymerase II transcription regulatory region sequence-specific DNA binding 類似検索 - 分子機能 |
| 生物種 | Mouse (Mus musculus) Human (Homo sapiens) |
 引用 引用 |  ジャーナル: Structure / 年: 2019 ジャーナル: Structure / 年: 2019タイトル: Interplay of Protein Disorder in Retinoic Acid Receptor Heterodimer and Its Corepressor Regulates Gene Expression. 著者: Tiago N Cordeiro / Nathalie Sibille / Pierre Germain / Philippe Barthe / Abdelhay Boulahtouf / Fréderic Allemand / Rémy Bailly / Valérie Vivat / Christine Ebel / Alessandro Barducci / ...著者: Tiago N Cordeiro / Nathalie Sibille / Pierre Germain / Philippe Barthe / Abdelhay Boulahtouf / Fréderic Allemand / Rémy Bailly / Valérie Vivat / Christine Ebel / Alessandro Barducci / William Bourguet / Albane le Maire / Pau Bernadó /    要旨: In its unliganded form, the retinoic acid receptor (RAR) in heterodimer with the retinoid X receptor (RXR) exerts a strong repressive activity facilitated by the recruitment of transcriptional ...In its unliganded form, the retinoic acid receptor (RAR) in heterodimer with the retinoid X receptor (RXR) exerts a strong repressive activity facilitated by the recruitment of transcriptional corepressors in the promoter region of target genes. By integrating complementary structural, biophysical, and computational information, we demonstrate that intrinsic disorder is a required feature for the precise regulation of RAR activity. We show that structural dynamics of RAR and RXR H12 regions is an essential mechanism for RAR regulation. Unexpectedly we found that, while mainly disordered, the corepressor N-CoR presents evolutionary conserved structured regions involved in transient intramolecular contacts. In the presence of RXR/RAR, N-CoR exploits its multivalency to form a cooperative multisite complex that displays equilibrium between different conformational states that can be tuned by cognate ligands and receptor mutations. This equilibrium is key to preserving the repressive basal state while allowing the conversion to a transcriptionally active form. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: RXR/RAR Heterodimer : N-CoRNID Complex (1:1) with RAR agonist (Am580) 試料濃度: 0.90-3.50 / Entity id: 1515 / 1536 / 1537 |
|---|---|
| バッファ | 名称: 50mM Tris HCl, 150mM NaCl, 2mM TCEP. / pH: 7.5 |
| 要素 #1515 | 名称: N-CoR-NID / タイプ: protein 記述: Nuclear receptor CoRepressor 1; Nuclear Receptor Interaction Domain (NID) 分子量: 29.139 / 分子数: 1 / 由来: Mouse (Mus musculus) / 参照: UniProt: Q60974 配列: GPHMQVPRTH RLITLADHIC QIITQDFARN QVPSQASTST FQTSPSALSS TPVRTKTSSR YSPESQSQTV LHPRPGPRVS PENLVDKSRG SRPGKSPERS HIPSEPYEPI SPPQGPAVHE KQDSMLLLSQ RGVDPAEQRS DSRSPGSISY LPSFFTKLES TSPMVKSKKQ ...配列: GPHMQVPRTH RLITLADHIC QIITQDFARN QVPSQASTST FQTSPSALSS TPVRTKTSSR YSPESQSQTV LHPRPGPRVS PENLVDKSRG SRPGKSPERS HIPSEPYEPI SPPQGPAVHE KQDSMLLLSQ RGVDPAEQRS DSRSPGSISY LPSFFTKLES TSPMVKSKKQ EIFRKLNSSG GGDSDMAAAQ PGTEIFNLPA VTTSGAVSSR SHSFADPASN LGLEDIIRKA LMGSFDDKVE DHGVVMSHPV GIMPGSASTS VVTSSEARRD E |
| 要素 #1536 | 名称: RXR / タイプ: protein 記述: Retinoid-X receptor alpha (RXR-alpha) Ligand Binding Domain (LBD) 分子量: 26.47 / 分子数: 1 / 由来: Mouse (Mus musculus) / 参照: UniProt: P28700 配列: SANEDMPVEK ILEAELAVEP KTETYVEANM GLNPSSPNDP VTNICQAADK QLFTLVEWAK RIPHFSELPL DDQVILLRAG WNELLIASAS HRSIAVKDGI LLATGLHVHR NSAHSAGVGA IFDRVLTELV SKMRDMQMDK TELGCLRAIV LFNPDSKGLS NPAEVEALRE ...配列: SANEDMPVEK ILEAELAVEP KTETYVEANM GLNPSSPNDP VTNICQAADK QLFTLVEWAK RIPHFSELPL DDQVILLRAG WNELLIASAS HRSIAVKDGI LLATGLHVHR NSAHSAGVGA IFDRVLTELV SKMRDMQMDK TELGCLRAIV LFNPDSKGLS NPAEVEALRE KVYASLEAYC KHKYPEQPGR FAKLLLRLPA LRSIGLKCLE HLFFFKLIGD TPIDTFLMEM LEAPHQAT |
| 要素 #1537 | 名称: RAR / タイプ: protein 記述: Retinoic acid receptor alpha (RAR-alpha) Ligand binding domain (LDB) 分子量: 28.047 / 分子数: 1 / 由来: Human (Homo sapiens) / 参照: UniProt: P10276 配列: GSHESYTLTP EVGELIEKVR KAHQETFPAL CQLGKYTTNN SSEQRVSLDI DLWDKFSELS TKCIIKTVEF AKQLPGFTTL TIADQITLLK AACLDILILR ICTRYTPEQD TMTFSDGLTL NRTQMHNAGF GPLTDLVFAF ANQLLPLEMD DAETGLLSAI CLICGDRQDL ...配列: GSHESYTLTP EVGELIEKVR KAHQETFPAL CQLGKYTTNN SSEQRVSLDI DLWDKFSELS TKCIIKTVEF AKQLPGFTTL TIADQITLLK AACLDILILR ICTRYTPEQD TMTFSDGLTL NRTQMHNAGF GPLTDLVFAF ANQLLPLEMD DAETGLLSAI CLICGDRQDL EQPDRVDMLQ EPLLEALKVY VRKRRPSRPH MFPKMLMKIT DLRSISAKGA ERVITLKMEI PGSMPPLIQE MLENSEGLD |
-実験情報
| ビーム | 設備名称: ESRF BM29 / 地域: Grenoble / 国: France  / 線源: X-ray synchrotron / 波長: 0.099 Å / スペクトロメータ・検出器間距離: 2.9 mm / 線源: X-ray synchrotron / 波長: 0.099 Å / スペクトロメータ・検出器間距離: 2.9 mm | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 1M / タイプ: Dectris / Pixsize x: 172 mm | ||||||||||||||||||||||||||||||||||||
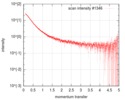
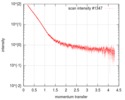
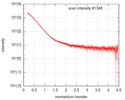
| スキャン | 測定日: 2014年7月23日 / 保管温度: 10 °C / セル温度: 10 °C / 照射時間: 1 sec. / フレーム数: 10 / 単位: 1/nm /
| ||||||||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||||||||||||||||||||
| 結果 | コメント: Note: Am580 = 4-[(5,6,7,8-Tetrahydro-5,5,8,8- tetramethyl-2- naphthalenyl) carboxamido]benzoic acid
|
 ムービー
ムービー コントローラー
コントローラー



 SASDF84
SASDF84