+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDEH2 |
|---|---|
 試料 試料 | Unlabeled nucleoporin NUP49/NSP49 (N49) without denaturant
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nuclear pore central transport channel / telomere tethering at nuclear periphery / nuclear pore cytoplasmic filaments / post-transcriptional tethering of RNA polymerase II gene DNA at nuclear periphery / tRNA export from nucleus / structural constituent of nuclear pore / nuclear localization sequence binding / RNA export from nucleus / nucleocytoplasmic transport / poly(A)+ mRNA export from nucleus ...nuclear pore central transport channel / telomere tethering at nuclear periphery / nuclear pore cytoplasmic filaments / post-transcriptional tethering of RNA polymerase II gene DNA at nuclear periphery / tRNA export from nucleus / structural constituent of nuclear pore / nuclear localization sequence binding / RNA export from nucleus / nucleocytoplasmic transport / poly(A)+ mRNA export from nucleus / ribosomal large subunit export from nucleus / nuclear pore / molecular condensate scaffold activity / protein import into nucleus / nuclear envelope / nuclear membrane / amyloid fibril formation / RNA binding / identical protein binding 類似検索 - 分子機能 |
| 生物種 |  |
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2017 ジャーナル: Proc Natl Acad Sci U S A / 年: 2017タイトル: Decoupling of size and shape fluctuations in heteropolymeric sequences reconciles discrepancies in SAXS vs. FRET measurements. 著者: Gustavo Fuertes / Niccolò Banterle / Kiersten M Ruff / Aritra Chowdhury / Davide Mercadante / Christine Koehler / Michael Kachala / Gemma Estrada Girona / Sigrid Milles / Ankur Mishra / ...著者: Gustavo Fuertes / Niccolò Banterle / Kiersten M Ruff / Aritra Chowdhury / Davide Mercadante / Christine Koehler / Michael Kachala / Gemma Estrada Girona / Sigrid Milles / Ankur Mishra / Patrick R Onck / Frauke Gräter / Santiago Esteban-Martín / Rohit V Pappu / Dmitri I Svergun / Edward A Lemke /     要旨: Unfolded states of proteins and native states of intrinsically disordered proteins (IDPs) populate heterogeneous conformational ensembles in solution. The average sizes of these heterogeneous ...Unfolded states of proteins and native states of intrinsically disordered proteins (IDPs) populate heterogeneous conformational ensembles in solution. The average sizes of these heterogeneous systems, quantified by the radius of gyration ( ), can be measured by small-angle X-ray scattering (SAXS). Another parameter, the mean dye-to-dye distance ( ) for proteins with fluorescently labeled termini, can be estimated using single-molecule Förster resonance energy transfer (smFRET). A number of studies have reported inconsistencies in inferences drawn from the two sets of measurements for the dimensions of unfolded proteins and IDPs in the absence of chemical denaturants. These differences are typically attributed to the influence of fluorescent labels used in smFRET and to the impact of high concentrations and averaging features of SAXS. By measuring the dimensions of a collection of labeled and unlabeled polypeptides using smFRET and SAXS, we directly assessed the contributions of dyes to the experimental values and For chemically denatured proteins we obtain mutual consistency in our inferences based on and , whereas for IDPs under native conditions, we find substantial deviations. Using computations, we show that discrepant inferences are neither due to methodological shortcomings of specific measurements nor due to artifacts of dyes. Instead, our analysis suggests that chemical heterogeneity in heteropolymeric systems leads to a decoupling between and that is amplified in the absence of denaturants. Therefore, joint assessments of and combined with measurements of polymer shapes should provide a consistent and complete picture of the underlying ensembles. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-モデル
| モデル #2246 |  タイプ: atomic / ソフトウェア: (CAMPARI) コメント: p-acetylphenylalanine was replaced with a cysteine. カイ2乗値: 0.956 / P-value: 0.184774  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
|---|---|
| モデル #2247 | 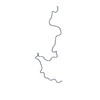 タイプ: atomic / ソフトウェア: (CAMPARI) コメント: p-acetylphenylalanine was replaced with a cysteine. カイ2乗値: 0.956 / P-value: 0.184774  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #2248 | 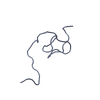 タイプ: atomic / ソフトウェア: (CAMPARI) コメント: p-acetylphenylalanine was replaced with a cysteine. カイ2乗値: 0.956 / P-value: 0.184774  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #2249 |  タイプ: atomic / ソフトウェア: (CAMPARI) コメント: p-acetylphenylalanine was replaced with a cysteine. カイ2乗値: 0.956 / P-value: 0.184774  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #2250 | 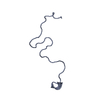 タイプ: atomic / ソフトウェア: (CAMPARI) コメント: p-acetylphenylalanine was replaced with a cysteine. カイ2乗値: 0.956 / P-value: 0.184774  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #2251 | 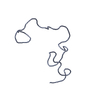 タイプ: atomic / ソフトウェア: (CAMPARI) コメント: p-acetylphenylalanine was replaced with a cysteine. カイ2乗値: 0.956 / P-value: 0.184774  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #2252 |  タイプ: atomic / ソフトウェア: (CAMPARI) コメント: p-acetylphenylalanine was replaced with a cysteine. カイ2乗値: 0.956 / P-value: 0.184774  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #2253 |  タイプ: atomic / ソフトウェア: (CAMPARI) コメント: p-acetylphenylalanine was replaced with a cysteine. カイ2乗値: 0.956 / P-value: 0.184774  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #2254 |  タイプ: atomic / ソフトウェア: (CAMPARI) コメント: p-acetylphenylalanine was replaced with a cysteine. カイ2乗値: 0.956 / P-value: 0.184774  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
- 試料
試料
 試料 試料 | 名称: Unlabeled nucleoporin NUP49/NSP49 (N49) without denaturant 試料濃度: 2.00-10.00 |
|---|---|
| バッファ | 名称: PBS, 10 mM DTT / pH: 7.4 |
| 要素 #1229 | 名称: N49 / タイプ: protein / 記述: Nucleoporin NUP49/NSP49 / 分子量: 3.853 / 分子数: 1 由来: Saccharomyces cerevisiae (strain ATCC 204508 / S288c) 参照: UniProt: Q02199 配列: GCQTSRGLFG NNNTNNINNS SSGMNNASAG LFGSKPUA |
-実験情報
| ビーム | 設備名称: PETRA III EMBL P12 / 地域: Hamburg / 国: Germany  / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 3 mm / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 3 mm | |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 2M | |||||||||||||||||||||||||||||||||
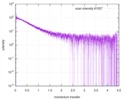
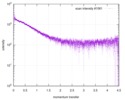
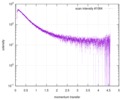
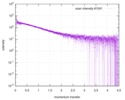
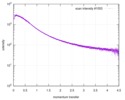
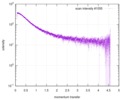
| スキャン | 測定日: 2013年12月8日 / セル温度: 23 °C / 照射時間: 0.05 sec. / フレーム数: 20 / 単位: 1/nm /
| |||||||||||||||||||||||||||||||||
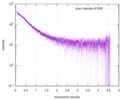
| 距離分布関数 P(R) |
| |||||||||||||||||||||||||||||||||
| 結果 | コメント: The protein contains a penultimate non-canonical amino acid p-acetylphenylalanine (207 Da) that is represented as U (selenocysteine, 168 Da) in the amino acid sequence for the entry. ...コメント: The protein contains a penultimate non-canonical amino acid p-acetylphenylalanine (207 Da) that is represented as U (selenocysteine, 168 Da) in the amino acid sequence for the entry. Therefore, the calculated MW from sequence (MW(expected)) must be adjusted accordingly (ca. 40 Da).
|
 ムービー
ムービー コントローラー
コントローラー


 SASDEH2
SASDEH2