+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 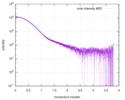 |
|---|---|
 試料 試料 | Bovine serum albumin mixture: averaged and individual data frames (subtracted and unsubtracted test sets)
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to calcium ion starvation / enterobactin binding / negative regulation of mitochondrial depolarization / toxic substance binding / cellular response to starvation / fatty acid binding / pyridoxal phosphate binding / protein-containing complex / extracellular space / DNA binding ...cellular response to calcium ion starvation / enterobactin binding / negative regulation of mitochondrial depolarization / toxic substance binding / cellular response to starvation / fatty acid binding / pyridoxal phosphate binding / protein-containing complex / extracellular space / DNA binding / extracellular region / metal ion binding / cytoplasm 類似検索 - 分子機能 |
| 生物種 |  |
 引用 引用 |  ジャーナル: Biophys J / 年: 2018 ジャーナル: Biophys J / 年: 2018タイトル: Machine Learning Methods for X-Ray Scattering Data Analysis from Biomacromolecular Solutions. 著者: Daniel Franke / Cy M Jeffries / Dmitri I Svergun /  要旨: Small-angle x-ray scattering (SAXS) of biological macromolecules in solutions is a widely employed method in structural biology. SAXS patterns include information about the overall shape and low- ...Small-angle x-ray scattering (SAXS) of biological macromolecules in solutions is a widely employed method in structural biology. SAXS patterns include information about the overall shape and low-resolution structure of dissolved particles. Here, we describe how to transform experimental SAXS patterns to feature vectors and how a simple k-nearest neighbor approach is able to retrieve information on overall particle shape and maximal diameter (D) as well as molecular mass directly from experimental scattering data. Based on this transformation, we develop a rapid multiclass shape-classification ranging from compact, extended, and flat categories to hollow and random-chain-like objects. This classification may be employed, e.g., as a decision block in automated data analysis pipelines. Further, we map protein structures from the Protein Data Bank into the classification space and, in a second step, use this mapping as a data source to obtain accurate estimates for the structural parameters (D, molecular mass) of the macromolecule under study based on the experimental scattering pattern alone, without inverse Fourier transform for D. All methods presented are implemented in a Fortran binary DATCLASS, part of the ATSAS data analysis suite, available on Linux, Mac, and Windows and free for academic use. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: Bovine serum albumin mixture: averaged and individual data frames (subtracted and unsubtracted test sets) 試料濃度: 2.25 mg/ml |
|---|---|
| バッファ | 名称: 50 mM HEPES / 濃度: 50.00 mM / pH: 7.5 |
| 要素 #969 | 名称: BSA / タイプ: protein / 記述: Bovine serum albumin / 分子量: 66.462 / 由来: Bos taurus / 参照: UniProt: P02769 配列: DTHKSEIAHR FKDLGEEHFK GLVLIAFSQY LQQCPFDEHV KLVNELTEFA KTCVADESHA GCEKSLHTLF GDELCKVASL RETYGDMADC CEKQEPERNE CFLSHKDDSP DLPKLKPDPN TLCDEFKADE KKFWGKYLYE IARRHPYFYA PELLYYANKY NGVFQECCQA ...配列: DTHKSEIAHR FKDLGEEHFK GLVLIAFSQY LQQCPFDEHV KLVNELTEFA KTCVADESHA GCEKSLHTLF GDELCKVASL RETYGDMADC CEKQEPERNE CFLSHKDDSP DLPKLKPDPN TLCDEFKADE KKFWGKYLYE IARRHPYFYA PELLYYANKY NGVFQECCQA EDKGACLLPK IETMREKVLT SSARQRLRCA SIQKFGERAL KAWSVARLSQ KFPKAEFVEV TKLVTDLTKV HKECCHGDLL ECADDRADLA KYICDNQDTI SSKLKECCDK PLLEKSHCIA EVEKDAIPEN LPPLTADFAE DKDVCKNYQE AKDAFLGSFL YEYSRRHPEY AVSVLLRLAK EYEATLEECC AKDDPHACYS TVFDKLKHLV DEPQNLIKQN CDQFEKLGEY GFQNALIVRY TRKVPQVSTP TLVEVSRSLG KVGTRCCTKP ESERMPCTED YLSLILNRLC VLHEKTPVSE KVTKCCTESL VNRRPCFSAL TPDETYVPKA FDEKLFTFHA DICTLPDTEK QIKKQTALVE LLKHKPKATE EQLKTVMENF VAFVDKCCAA DDKEACFAVE GPKLVVSTQT ALA |
-実験情報
| ビーム | 設備名称: PETRA III EMBL P12 / 地域: Hamburg / 国: Germany  / 線源: X-ray synchrotron / 波長: 0.124 Å / スペクトロメータ・検出器間距離: 3.1 mm / 線源: X-ray synchrotron / 波長: 0.124 Å / スペクトロメータ・検出器間距離: 3.1 mm | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 2M | ||||||||||||||||||||||||||||||
| スキャン |
| ||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||||||||||||||
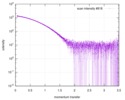
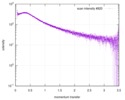
| 結果 | コメント: The data displayed in this entry represents the averaged 1D-SAXS profile obtained from 100 individual SAXS data frames. Included in the full entry zip-archive are each of the individual ...コメント: The data displayed in this entry represents the averaged 1D-SAXS profile obtained from 100 individual SAXS data frames. Included in the full entry zip-archive are each of the individual subtracted SAXS profiles as well as each individual sample and buffer frame (100 in total).
|
 ムービー
ムービー コントローラー
コントローラー



 SASDDN3
SASDDN3