+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 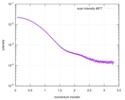 |
|---|---|
 試料 試料 | NBD-MsbA (apo)
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報MsbA transporter complex / lipopolysaccharide floppase activity / lipid translocation / ABC-type lipid A-core oligosaccharide transporter / lipopolysaccharide transport / ATPase-coupled lipid transmembrane transporter activity / ABC-type xenobiotic transporter activity / lipid transport / ATP-binding cassette (ABC) transporter complex / transmembrane transport ...MsbA transporter complex / lipopolysaccharide floppase activity / lipid translocation / ABC-type lipid A-core oligosaccharide transporter / lipopolysaccharide transport / ATPase-coupled lipid transmembrane transporter activity / ABC-type xenobiotic transporter activity / lipid transport / ATP-binding cassette (ABC) transporter complex / transmembrane transport / lipid binding / ATP hydrolysis activity / ATP binding / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 |
| 生物種 |  |
 引用 引用 |  ジャーナル: Structure / 年: 2018 ジャーナル: Structure / 年: 2018タイトル: Conformational States of ABC Transporter MsbA in a Lipid Environment Investigated by Small-Angle Scattering Using Stealth Carrier Nanodiscs. 著者: Inokentijs Josts / Julius Nitsche / Selma Maric / Haydyn D Mertens / Martine Moulin / Michael Haertlein / Sylvain Prevost / Dmitri I Svergun / Sebastian Busch / V Trevor Forsyth / Henning Tidow /     要旨: Structural studies of integral membrane proteins (IMPs) are challenging, as many of them are inactive or insoluble in the absence of a lipid environment. Here, we describe an approach making use of ...Structural studies of integral membrane proteins (IMPs) are challenging, as many of them are inactive or insoluble in the absence of a lipid environment. Here, we describe an approach making use of fractionally deuterium labeled "stealth carrier" nanodiscs that are effectively invisible to low-resolution neutron diffraction and enable structural studies of IMPs in a lipidic native-like solution environment. We illustrate the potential of the method in a joint small-angle neutron scattering (SANS) and X-ray scattering (SAXS) study of the ATP-binding cassette (ABC) transporter protein MsbA solubilized in the stealth nanodiscs. The data allow for a direct observation of the signal from the solubilized protein without contribution from the surrounding lipid nanodisc. Not only the overall shape but also differences between conformational states of MsbA can be reliably detected from the scattering data, demonstrating the sensitivity of the approach and its general applicability to structural studies of IMPs. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: NBD-MsbA (apo) / Contrast: 2.842 / Specific vol: 0.738 / 試料濃度: 18 mg/ml / 濃度測定法: A280 |
|---|---|
| バッファ | 名称: 30 mM Tris, 150 mM NaCl, 0.5 mM TCEP / pH: 7.5 |
| 要素 #1019 | 名称: NBD-MsbA / タイプ: protein 記述: Nucleotide Binding Domain of Lipid A export ATP-binding/permease protein MsbA 分子量: 27.263 / 分子数: 1 / 由来: Escherichia coli (strain K12) / 参照: UniProt: P60752 配列: RATGDVEFRN VTFTYPGRDV PALRNINLKI PAGKTVALVG RSGSGKSTIA SLITRFYDID EGEILMDGHD LREYTLASLR NQVALVSQNV HLFNDTVANN IAYARTEQYS REQIEEAARM AYAMDFINKM DNGLDTVIGE NGVLLSGGQR QRIAIARALL RDSPILILDE ...配列: RATGDVEFRN VTFTYPGRDV PALRNINLKI PAGKTVALVG RSGSGKSTIA SLITRFYDID EGEILMDGHD LREYTLASLR NQVALVSQNV HLFNDTVANN IAYARTEQYS REQIEEAARM AYAMDFINKM DNGLDTVIGE NGVLLSGGQR QRIAIARALL RDSPILILDE ATSALDTESE RAIQAALDEL QKNRTSLVIA HRLSTIEKAD EIVVVEDGVI VERGTHNDLL EHRGVYAQLH KMQFGQ |
-実験情報
| ビーム | 設備名称: PETRA III EMBL P12 / 地域: Hamburg / 国: Germany  / 線源: X-ray synchrotron / 波長: 0.124 Å / スペクトロメータ・検出器間距離: 3.3 mm / 線源: X-ray synchrotron / 波長: 0.124 Å / スペクトロメータ・検出器間距離: 3.3 mm | ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 2M | ||||||||||||||||||||||||||||||||||||||||||
| スキャン |
| ||||||||||||||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||||||||||||||||||||||||||
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー



 SASDDE5
SASDDE5





