+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDD25 |
|---|---|
 試料 試料 | 1:1 Mixture between Protein sex-lethal mutant (Sxl10GS) and RNA decaneucleotide UGU8
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sex determination, primary response to X:A ratio / germarium-derived cystoblast division / epithelium regeneration / somatic sex determination / female germ-line sex determination / oocyte differentiation / female sex determination / imaginal disc growth / sex determination / regulation of stem cell division ...sex determination, primary response to X:A ratio / germarium-derived cystoblast division / epithelium regeneration / somatic sex determination / female germ-line sex determination / oocyte differentiation / female sex determination / imaginal disc growth / sex determination / regulation of stem cell division / poly-pyrimidine tract binding / negative regulation of RNA export from nucleus / sex-chromosome dosage compensation / sex differentiation / alternative mRNA splicing, via spliceosome / poly(A) binding / pre-mRNA binding / regulation of mRNA splicing, via spliceosome / positive regulation of smoothened signaling pathway / reciprocal meiotic recombination / poly(U) RNA binding / oogenesis / regulation of alternative mRNA splicing, via spliceosome / negative regulation of mRNA splicing, via spliceosome / negative regulation of translational initiation / mRNA regulatory element binding translation repressor activity / positive regulation of RNA splicing / mRNA 3'-UTR binding / mRNA 5'-UTR binding / negative regulation of translation / protein stabilization / ribonucleoprotein complex / mRNA binding / protein-containing complex / RNA binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 |
| 生物種 |  synthetic construct (人工物) |
 引用 引用 |  ジャーナル: ACS Comb Sci / 年: 2018 ジャーナル: ACS Comb Sci / 年: 2018タイトル: A General Small-Angle X-ray Scattering-Based Screening Protocol Validated for Protein-RNA Interactions. 著者: Po-Chia Chen / Pawel Masiewicz / Vladimir Rybin / Dmitri Svergun / Janosch Hennig /  要旨: We present a screening protocol utilizing small-angle X-ray scattering (SAXS) to obtain structural information on biomolecular interactions independent of prior knowledge, so as to complement ...We present a screening protocol utilizing small-angle X-ray scattering (SAXS) to obtain structural information on biomolecular interactions independent of prior knowledge, so as to complement affinity-based screening and provide leads for further exploration. This protocol categorizes ligand titrations by computing pairwise agreement between curves, and separately estimates affinities by quantifying complex formation as a departure from the linear sum properties of solution SAXS. The protocol is validated by sparse sequence search around the native poly uridine RNA motifs of the two-RRM domain Sex-lethal protein (Sxl). The screening of 35 RNA motifs between 4 to 10 nucleotides reveals a strong variation of resulting complexes, revealed to be preference-switching between 1:1 and 2:2 binding stoichiometries upon addition of structural modeling. Validation of select sequences in isothermal calorimetry and NMR titration retrieves domain-specific roles and function of a guanine anchor. These findings reinforce the suitability of SAXS as a complement in lead identification. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-モデル
| モデル #1877 |  タイプ: mix / ソフトウェア: (2.1) / ダミー原子の半径: 1.90 A / カイ2乗値: 0.777 / P-value: 0.240677  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
|---|---|
| モデル #1878 |  タイプ: mix / ソフトウェア: (2.1) / ダミー原子の半径: 1.90 A / カイ2乗値: 0.777 / P-value: 0.240677  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #1879 |  タイプ: mix / ソフトウェア: (2.1) / ダミー原子の半径: 1.90 A / カイ2乗値: 0.777 / P-value: 0.240677  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
- 試料
試料
 試料 試料 | 名称: 1:1 Mixture between Protein sex-lethal mutant (Sxl10GS) and RNA decaneucleotide UGU8 試料濃度: 1 mg/ml / Entity id: 1008 / 1009 |
|---|---|
| バッファ | 名称: 50% dilution of protein buffer {10 mM KP, 50 mM NaCl, 10 mM DTT pH 6} with {milliQ-water pH 7} suspended RNA pH: 6 |
| 要素 #1008 | 名称: Sxl10GS / タイプ: protein / 記述: Protein sex-lethal mutant / 分子量: 20.432 / 分子数: 2 / 由来: Drosophila melanogaster / 参照: UniProt: P19339 配列: GAMASNTNLI VNYLPQDMTD RELYALFRAI GPINTCRIMR DYKTGYSFGY AFVDFTSEMD SQRAIKVLNG ITVRNKRLKV SYARPGGGSG SGGGGSGESI KDTNLYVTNL PRTITDDQLD TIFGKYGSIV QKNILRDKLT GRPRGVAFVR YNKREEAQEA ISALNNVIPE GGSQPLSVRL AEEHGK |
| 要素 #1009 | 名称: UGU8 / タイプ: RNA / 記述: RNA decaneucleotide UGU8 / 分子量: 3.119 / 分子数: 2 / 由来: synthetic construct 配列: UGUUUUUUUU |
-実験情報
| ビーム | 設備名称: ESRF BM29 / 地域: Grenoble / 国: France  / 線源: X-ray synchrotron / 波長: 0.09919 Å / スペクトロメータ・検出器間距離: 2.867 mm / 線源: X-ray synchrotron / 波長: 0.09919 Å / スペクトロメータ・検出器間距離: 2.867 mm | |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 1M | |||||||||||||||||||||||||||||||||
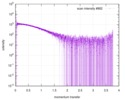
| スキャン | 測定日: 2017年4月12日 / 保管温度: 20 °C / セル温度: 20 °C / 照射時間: 0.5 sec. / フレーム数: 10 / 単位: 1/nm /
| |||||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| |||||||||||||||||||||||||||||||||
| 結果 | コメント: This synchrotron session suffered from misalignment of the beamstop resulting in parasitic scattering at low angles. Note that the uploaded EOM ensemble is chosen as the 2:2-complex ...コメント: This synchrotron session suffered from misalignment of the beamstop resulting in parasitic scattering at low angles. Note that the uploaded EOM ensemble is chosen as the 2:2-complex with one RNA monomer as fully bound by two Sxl10GS molecules in the canonical position from Protein data bankk (PDB) entry 1B7F.pdb. The second RNA monomer is placed in the canonical position bound to the free RRM2, resulting in three rigid bodies: RRM2+RNA, RRM1+RRM2+RNA, and RRM1.
|
 ムービー
ムービー コントローラー
コントローラー


 SASDD25
SASDD25