+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  |
|---|---|
 試料 試料 | Nucleolysin TIA-1 isoform p40 in complex with U15 RNA
|
| 機能・相同性 | Isoform Short of Cytotoxic granule associated RNA binding protein TIA1 機能・相同性情報 機能・相同性情報 |
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
 引用 引用 |  ジャーナル: Angew Chem Int Ed Engl / 年: 2017 ジャーナル: Angew Chem Int Ed Engl / 年: 2017タイトル: Segmental, Domain-Selective Perdeuteration and Small-Angle Neutron Scattering for Structural Analysis of Multi-Domain Proteins. 著者: Miriam Sonntag / Pravin Kumar Ankush Jagtap / Bernd Simon / Marie-Sousai Appavou / Arie Geerlof / Ralf Stehle / Frank Gabel / Janosch Hennig / Michael Sattler /   要旨: Multi-domain proteins play critical roles in fine-tuning essential processes in cellular signaling and gene regulation. Typically, multiple globular domains that are connected by flexible linkers ...Multi-domain proteins play critical roles in fine-tuning essential processes in cellular signaling and gene regulation. Typically, multiple globular domains that are connected by flexible linkers undergo dynamic rearrangements upon binding to protein, DNA or RNA ligands. RNA binding proteins (RBPs) represent an important class of multi-domain proteins, which regulate gene expression by recognizing linear or structured RNA sequence motifs. Here, we employ segmental perdeuteration of the three RNA recognition motif (RRM) domains in the RBP TIA-1 using Sortase A mediated protein ligation. We show that domain-selective perdeuteration combined with contrast-matched small-angle neutron scattering (SANS), SAXS and computational modeling provides valuable information to precisely define relative domain arrangements. The approach is generally applicable to study conformational arrangements of individual domains in multi-domain proteins and changes induced by ligand binding. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: Nucleolysin TIA-1 isoform p40 in complex with U15 RNA Entity id: 659 / 660 |
|---|---|
| バッファ | 名称: 10 mM Potassium Phosphate 50 mM NaCl 10 mM DTT / pH: 6 |
| 要素 #659 | 名称: TIA-1 wild type / タイプ: protein / 記述: Nucleolysin TIA-1 isoform p40 / 分子量: 30.457 / 分子数: 1 / 由来: Homo sapiens / 参照: UniProt: P31483-2 配列: MEDEMPKTLY VGNLSRDVTE ALILQLFSQI GPCKNCKMIM DTAGNDPYCF VEFHEHRHAA AALAAMNGRK IMGKEVKVNW ATTPSSQKKD TSNHFHVFVG DLSPEITTED IKAAFAPFGR ISDARVVKDM ATGKSKGYGF VSFFNKWDAE NAIQQMGGQW LGGRQIRTNW ...配列: MEDEMPKTLY VGNLSRDVTE ALILQLFSQI GPCKNCKMIM DTAGNDPYCF VEFHEHRHAA AALAAMNGRK IMGKEVKVNW ATTPSSQKKD TSNHFHVFVG DLSPEITTED IKAAFAPFGR ISDARVVKDM ATGKSKGYGF VSFFNKWDAE NAIQQMGGQW LGGRQIRTNW ATRKPPAPKS TYESNTKQLS YDEVVNQSSP SNCTVYCGGV TSGLTEQLMR QTFSPFGQIM EIRVFPDKGY SFVRFNSHES AAHAIVSVNG TTIEGHVVKC YWGK |
| 要素 #660 | 名称: U15 / タイプ: RNA / 記述: poly U 15mer / 分子量: 4.611 / 分子数: 1 配列: UUUUUUUUUU UUUUU |
-実験情報
| ビーム | 設備名称: Rigaku BioSAXS-1000 / 地域: Munich / 国: Germany  / 線源: X-ray in house / 線源: X-ray in house | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 100K / Pixsize x: 172 mm | ||||||||||||||||||||||||||||||||||||
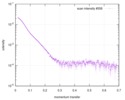
| スキャン | 測定日: 2016年4月28日 / 単位: 1/A /
| ||||||||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||||||||||||||||||||
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー



 SASDCC3
SASDCC3