+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  |
|---|---|
 試料 試料 | Cyclopentadecanone monooxygenase, NADP+, wild-type
|
| 機能・相同性 | Pyridine nucleotide-disulphide oxidoreductase / : / monooxygenase activity / FAD/NAD(P)-binding domain superfamily / Cyclopentadecanone 1,2-monooxygenase 機能・相同性情報 機能・相同性情報 |
| 生物種 |  Pseudomonas sp. HI-70 (バクテリア) Pseudomonas sp. HI-70 (バクテリア) |
 引用 引用 |  ジャーナル: Biochim Biophys Acta / 年: 2016 ジャーナル: Biochim Biophys Acta / 年: 2016タイトル: The role of conformational flexibility in Baeyer-Villiger monooxygenase catalysis and structure. 著者: Brahm J Yachnin / Peter C K Lau / Albert M Berghuis /  要旨: BACKGROUND: The Baeyer-Villiger monooxygenases (BMVOs) are a group of microbial enzymes that have garnered interest as industrial biocatalysts. While great strides have been made in recent years to ...BACKGROUND: The Baeyer-Villiger monooxygenases (BMVOs) are a group of microbial enzymes that have garnered interest as industrial biocatalysts. While great strides have been made in recent years to understand the mechanism of these enzymes from a structural perspective, our understanding remains incomplete. In particular, the role of a twenty residue loop (residues 487-504), which we refer to as the "Control Loop," that is observed in either an ordered or disordered state in various crystal structures remains unclear. 手法: Using SAXS, we have made the first observations of the Loop in solution with two BVMOs, cyclohexanone monooxygenase (CHMO) and cyclopentadecanone monooxygenase. We also made a series of ...手法: Using SAXS, we have made the first observations of the Loop in solution with two BVMOs, cyclohexanone monooxygenase (CHMO) and cyclopentadecanone monooxygenase. We also made a series of mutants of CHMO and analyzed them using SAXS, ITC, and an uncoupling assay. RESULTS: These experiments show that Control Loop ordering results in an overall more compact enzyme without altering global protein foldedness. We have also demonstrated that the Loop plays a ...RESULTS: These experiments show that Control Loop ordering results in an overall more compact enzyme without altering global protein foldedness. We have also demonstrated that the Loop plays a critical and complex role on enzyme structure and catalysis. The Control Loop appears to have a direct impact on the organization of the overall structure of the protein, as well as in influencing the active site environment. CONCLUSIONS: The data imply that the Loop can be divided into two regions, referred to as "sub-loops," that coordinate overall domain movements to changes in the active site. GENERAL SIGNIFICANCE: A better understanding of the mechanistic role of the Control Loop may ultimately be helpful in designing mutants with altered specificity and improved catalytic efficiency. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: Cyclopentadecanone monooxygenase, NADP+, wild-type / 試料濃度: 2.25-9.00 |
|---|---|
| バッファ | 名称: 50 mM Tris 2 mM TCEP 5 mM NADP+ / pH: 8 |
| 要素 #362 | タイプ: protein / 記述: Cyclopentadecanone 1,2-monooxygenase / 分子量: 69.13 / 分子数: 1 / 由来: Pseudomonas sp. HI-70 / 参照: UniProt: T2HVF7 配列: GSLEASMHMS QLIQEPAEAG VTSQKVSFDH VALREKYRQE RDKRLRQDGQ EQYLEVAVTC DEYLKDPYAD PIVRDPVVRE TDVFIIGGGF GGLLAAVRLQ QAGVSDYVMV ERAGDYGGTW YWNRYPGAQC DIESYVYMPL LEEMGYIPTE KYAFGTEILE YSRSIGRKFG ...配列: GSLEASMHMS QLIQEPAEAG VTSQKVSFDH VALREKYRQE RDKRLRQDGQ EQYLEVAVTC DEYLKDPYAD PIVRDPVVRE TDVFIIGGGF GGLLAAVRLQ QAGVSDYVMV ERAGDYGGTW YWNRYPGAQC DIESYVYMPL LEEMGYIPTE KYAFGTEILE YSRSIGRKFG LYERTYFQTE VKDLSWDDEA ARWRITTDRG DKFSARFVCM STGPLQRPKL PGIPGITSFK GHSFHTSRWD YSYTGGDQTG NLEGLKDKRV AIIGTGATSI QAVPHLAAYA QELYVIQRTP ISVGFRGNKP TDPEWAKSLQ PGWQQARMDN FNAITHGMPV DVDLVQDSWT KIFGEIGVFL GSDGSRAQMV DFQLMEQIRA RVDQEVKDPA TAESLKPYYN IMCKRPGFHD SYLPSFNKPN VTLVDTQGAG VERITEKGLV VNGREYEVDC LIYATGFEYQ TKLSRRNGYE IHGRNGQPLS DKWKDGLSTL WGYHIRDFPN CFILGNGQSA VTPNFTHMLN EAGKHVAYVV KHCLDERVDV FEPTAEAEQA WVDHVMSFAG IKQQYDRECT PSYYNNEGQV NDVALTRNNF YPGGAVAFIN ILREWREKGD FAQFQQRKR |
-実験情報
| ビーム | 設備名称: Advanced Light Source (ALS) 12.3.1 (SIBYLS) / 地域: Berkeley, CA / 国: USA  / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 1.5 mm / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 1.5 mm | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: MAR 165 CCD | |||||||||||||||||||||
| スキャン | 測定日: 2012年10月2日 / セル温度: 20 °C / フレーム数: 4 / 単位: 1/A /
| |||||||||||||||||||||
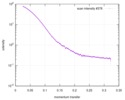
| 距離分布関数 P(R) |
| |||||||||||||||||||||
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー



 SASDBV5
SASDBV5