+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8a8n | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of self-assembling engineered protein nanocage (EPN) fused with hepatitis A pX protein | |||||||||||||||||||||
 要素 要素 | EPN-pX | |||||||||||||||||||||
 キーワード キーワード | STRUCTURAL PROTEIN / hepatitis A / pX / VP1-pX / nanocage / icosahedral / protein nanoparticle / virus assembly / enveloped viruses | |||||||||||||||||||||
| 機能・相同性 | KDPG/KHG aldolase / KDPG and KHG aldolase / Viral coat protein subunit / Aldolase-type TIM barrel / lyase activity / symbiont entry into host cell / virion attachment to host cell / 2-dehydro-3-deoxyphosphogluconate aldolase/4-hydroxy-2-oxoglutarate aldolase / Genome polyprotein 機能・相同性情報 機能・相同性情報 | |||||||||||||||||||||
| 生物種 |  Human immunodeficiency virus type 1 BH10 (ヒト免疫不全ウイルス) Human immunodeficiency virus type 1 BH10 (ヒト免疫不全ウイルス) | |||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.7 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Duyvesteyn, H.M.E. / Stuart, D.I. | |||||||||||||||||||||
| 資金援助 |  米国, 米国,  英国, 6件 英国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2022 ジャーナル: PLoS Pathog / 年: 2022タイトル: Nonlytic cellular release of hepatitis A virus requires dual capsid recruitment of the ESCRT-associated Bro1 domain proteins HD-PTP and ALIX. 著者: Takayoshi Shirasaki / Hui Feng / Helen M E Duyvesteyn / William G Fusco / Kevin L McKnight / Ling Xie / Mark Boyce / Sathish Kumar / Rina Barouch-Bentov / Olga González-López / Ryan ...著者: Takayoshi Shirasaki / Hui Feng / Helen M E Duyvesteyn / William G Fusco / Kevin L McKnight / Ling Xie / Mark Boyce / Sathish Kumar / Rina Barouch-Bentov / Olga González-López / Ryan McNamara / Li Wang / Adriana Hertel-Wulff / Xian Chen / Shirit Einav / Joseph A Duncan / Maryna Kapustina / Elizabeth E Fry / David I Stuart / Stanley M Lemon /   要旨: Although picornaviruses are conventionally considered 'nonenveloped', members of multiple picornaviral genera are released nonlytically from infected cells in extracellular vesicles. The mechanisms ...Although picornaviruses are conventionally considered 'nonenveloped', members of multiple picornaviral genera are released nonlytically from infected cells in extracellular vesicles. The mechanisms underlying this process are poorly understood. Here, we describe interactions of the hepatitis A virus (HAV) capsid with components of host endosomal sorting complexes required for transport (ESCRT) that play an essential role in release. We show release of quasi-enveloped virus (eHAV) in exosome-like vesicles requires a conserved export signal located within the 8 kDa C-terminal VP1 pX extension that functions in a manner analogous to late domains of canonical enveloped viruses. Fusing pX to a self-assembling engineered protein nanocage (EPN-pX) resulted in its ESCRT-dependent release in extracellular vesicles. Mutational analysis identified a 24 amino acid peptide sequence located within the center of pX that was both necessary and sufficient for nanocage release. Deleting a YxxL motif within this sequence ablated eHAV release, resulting in virus accumulating intracellularly. The pX export signal is conserved in non-human hepatoviruses from a wide range of mammalian species, and functional in pX sequences from bat hepatoviruses when fused to the nanocage protein, suggesting these viruses are released as quasi-enveloped virions. Quantitative proteomics identified multiple ESCRT-related proteins associating with EPN-pX, including ALG2-interacting protein X (ALIX), and its paralog, tyrosine-protein phosphatase non-receptor type 23 (HD-PTP), a second Bro1 domain protein linked to sorting of ubiquitylated cargo into multivesicular endosomes. RNAi-mediated depletion of either Bro1 domain protein impeded eHAV release. Super-resolution fluorescence microscopy demonstrated colocalization of viral capsids with endogenous ALIX and HD-PTP. Co-immunoprecipitation assays using biotin-tagged peptides and recombinant proteins revealed pX interacts directly through the export signal with N-terminal Bro1 domains of both HD-PTP and ALIX. Our study identifies an exceptionally potent viral export signal mediating extracellular release of virus-sized protein assemblies and shows release requires non-redundant activities of both HD-PTP and ALIX. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8a8n.cif.gz 8a8n.cif.gz | 48.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8a8n.ent.gz pdb8a8n.ent.gz | 34.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8a8n.json.gz 8a8n.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8a8n_validation.pdf.gz 8a8n_validation.pdf.gz | 729.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8a8n_full_validation.pdf.gz 8a8n_full_validation.pdf.gz | 731.6 KB | 表示 | |
| XML形式データ |  8a8n_validation.xml.gz 8a8n_validation.xml.gz | 22.6 KB | 表示 | |
| CIF形式データ |  8a8n_validation.cif.gz 8a8n_validation.cif.gz | 28.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/a8/8a8n https://data.pdbj.org/pub/pdb/validation_reports/a8/8a8n ftp://data.pdbj.org/pub/pdb/validation_reports/a8/8a8n ftp://data.pdbj.org/pub/pdb/validation_reports/a8/8a8n | HTTPS FTP |
-関連構造データ
| 関連構造データ | 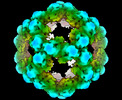 15234MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 実験データセット #1 | データ参照:  10.1101/2022.04.26.489493 / データの種類: other data 10.1101/2022.04.26.489493 / データの種類: other data |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 32934.234 Da / 分子数: 1 / 由来タイプ: 組換発現 詳細: Model just contains EPN-01 component since associated tag and pX could not be confidentally resolved.,Model just contains EPN-01 component since associated tag and pX could not be confidentally resolved. 由来: (組換発現)  Human immunodeficiency virus type 1 BH10 (ヒト免疫不全ウイルス) Human immunodeficiency virus type 1 BH10 (ヒト免疫不全ウイルス)細胞株 (発現宿主): HEK293T / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: Q9WXS1, UniProt: V9Z3B5 Homo sapiens (ヒト) / 参照: UniProt: Q9WXS1, UniProt: V9Z3B5 |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: EPN-pX / タイプ: COMPLEX 詳細: Associated model just contains EPN-01 component since associated tag and pX could not be confidentally resolved. Entity ID: all / 由来: RECOMBINANT |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:  Homo sapiens (ヒト) / 細胞: HEK293T Homo sapiens (ヒト) / 細胞: HEK293T |
| 緩衝液 | pH: 7 / 詳細: PBS |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 200 divisions/in. / グリッドのタイプ: Quantifoil R2/2 |
| 急速凍結 | 装置: GATAN CRYOPLUNGE 3 / 凍結剤: ETHANE / 湿度: 90 % / 凍結前の試料温度: 293 K / 詳細: Blot time 4-5 s. |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: TFS GLACIOS |
|---|---|
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 92000 X / 最大 デフォーカス(公称値): 3000 nm / 最小 デフォーカス(公称値): 700 nm / C2レンズ絞り径: 50 µm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 46 e/Å2 / 検出モード: INTEGRATING フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 撮影したグリッド数: 1 / 実像数: 1803 |
- 解析
解析
| EMソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | |||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1830 / 詳細: Manual selection. | |||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: I (正20面体型対称) | |||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 6.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 1830 / 対称性のタイプ: POINT | |||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL / Target criteria: Correlation coefficient 詳細: Rigid body fit monomer in chimera, generated biological unit then checked in coot and residues to be altered done so in coot followed by one cycle of rigid body refinement of entire ...詳細: Rigid body fit monomer in chimera, generated biological unit then checked in coot and residues to be altered done so in coot followed by one cycle of rigid body refinement of entire icosahedral assembly in phenix, defining each monomeric unit as a separate body and considering ncs constraints to check for clashes. | |||||||||||||||||||||||||||||||||
| 原子モデル構築 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj

