+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7z14 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Torpedo nicotinic acetylcholine receptor in complex with a short-chain neurotoxin. | |||||||||||||||||||||
 要素 要素 |
| |||||||||||||||||||||
 キーワード キーワード | MEMBRANE PROTEIN / Ion channel / toxin | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報acetylcholine-gated monoatomic cation-selective channel activity / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / transmembrane signaling receptor activity / postsynaptic membrane / neuron projection 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 | synthetic construct (人工物) | |||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.15 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Nys, M.A.E.M. / Zarkadas, E. / Ulens, C. / Nury, H. | |||||||||||||||||||||
| 資金援助 |  ベルギー, ベルギー,  英国, 英国,  フランス, European Union, 6件 フランス, European Union, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: The molecular mechanism of snake short-chain α-neurotoxin binding to muscle-type nicotinic acetylcholine receptors. 著者: Mieke Nys / Eleftherios Zarkadas / Marijke Brams / Aujan Mehregan / Kumiko Kambara / Jeroen Kool / Nicholas R Casewell / Daniel Bertrand / John E Baenziger / Hugues Nury / Chris Ulens /       要旨: Bites by elapid snakes (e.g. cobras) can result in life-threatening paralysis caused by venom neurotoxins blocking neuromuscular nicotinic acetylcholine receptors. Here, we determine the cryo-EM ...Bites by elapid snakes (e.g. cobras) can result in life-threatening paralysis caused by venom neurotoxins blocking neuromuscular nicotinic acetylcholine receptors. Here, we determine the cryo-EM structure of the muscle-type Torpedo receptor in complex with ScNtx, a recombinant short-chain α-neurotoxin. ScNtx is pinched between loop C on the principal subunit and a unique hairpin in loop F on the complementary subunit, thereby blocking access to the neurotransmitter binding site. ScNtx adopts a binding mode that is tilted toward the complementary subunit, forming a wider network of interactions than those seen in the long-chain α-Bungarotoxin complex. Certain mutations in ScNtx at the toxin-receptor interface eliminate inhibition of neuronal α7 nAChRs, but not of human muscle-type receptors. These observations explain why ScNtx binds more tightly to muscle-type receptors than neuronal receptors. Together, these data offer a framework for understanding subtype-specific actions of short-chain α-neurotoxins and inspire strategies for design of new snake antivenoms. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7z14.cif.gz 7z14.cif.gz | 360.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7z14.ent.gz pdb7z14.ent.gz | 291.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7z14.json.gz 7z14.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7z14_validation.pdf.gz 7z14_validation.pdf.gz | 1.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7z14_full_validation.pdf.gz 7z14_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  7z14_validation.xml.gz 7z14_validation.xml.gz | 50.2 KB | 表示 | |
| CIF形式データ |  7z14_validation.cif.gz 7z14_validation.cif.gz | 72.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/z1/7z14 https://data.pdbj.org/pub/pdb/validation_reports/z1/7z14 ftp://data.pdbj.org/pub/pdb/validation_reports/z1/7z14 ftp://data.pdbj.org/pub/pdb/validation_reports/z1/7z14 | HTTPS FTP |
-関連構造データ
| 関連構造データ | 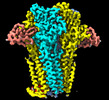 14440MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Acetylcholine receptor subunit ... , 4種, 5分子 ADBCE
| #1: タンパク質 | 分子量: 50168.164 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  #2: タンパク質 | | 分子量: 53731.773 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #3: タンパク質 | | 分子量: 57625.711 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #4: タンパク質 | | 分子量: 56335.684 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|
-タンパク質 , 1種, 2分子 FG
| #5: タンパク質 | 分子量: 6848.960 Da / 分子数: 2 / 由来タイプ: 組換発現 / 詳細: Consensus sequence from Elapidae family (8602) / 由来: (組換発現) synthetic construct (人工物) / 発現宿主:  |
|---|
-糖 , 4種, 6分子 
| #6: 多糖 | | #7: 多糖 | #8: 多糖 | alpha-D-mannopyranose-(1-6)-alpha-D-mannopyranose-(1-6)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1- ...alpha-D-mannopyranose-(1-6)-alpha-D-mannopyranose-(1-6)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose | タイプ: oligosaccharide / 分子量: 951.875 Da / 分子数: 1 / 由来タイプ: 組換発現 #9: 糖 | ChemComp-NAG / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Cryo-EM structure of Torpedo nicotinic acetylcholine receptor in complex with a short-chain neurotoxin. タイプ: COMPLEX / Entity ID: #1, #3, #2, #4-#5 / 由来: MULTIPLE SOURCES |
|---|---|
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: TFS GLACIOS |
|---|---|
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2200 nm / 最小 デフォーカス(公称値): 700 nm |
| 撮影 | 電子線照射量: 40 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.15 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 26581 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 精密化 | 交差検証法: NONE 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 89.67 Å2 | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj


